1.进化树1.0版本
上一期已经给大家介绍根据基因或者蛋白ID在NCBI上进行BLAST并下载FASTA序列,然后将序列导入MEGA软件中进行比对,最后生成.meg的序列比对文件。
那么用这个文件是不是就能构建进化树呢,当然可以。方法也很简单,单击菜单【File】→【Open A File/Session】,打开上述.meg文件,点击工具栏中的【PHYLOGENY】→【Construct/Test Neighbor-Joining Tree】,表示用邻接法构建进化树。在弹出的对话框中选择参数,一般默认即可。这样就生成了一个最简单的进化树。
现在问题来了,进化树是有了,但这棵树长得着实有点丑,最大的问题在于后面一长条英文看得人脑壳疼。那么怎么解决呢?
2.进化树2.0版本
2.1序列预处理
我们知道进化树的基本作用是反映物种的进化关系,而在分子生物学的研究中,进化树也能反映某单个或几个基因的进化情况。上一期的推送里我们的目的基因是水稻绿色革命基因SD1,这是一个赤霉素20氧化酶基因。我们在NCBI下载其同源序列后得到的FASTA文件是这样的:

现在发现问题了吧,从NCBI下载的序列中,每条序列的名称非常繁琐,这也直接导致了我们后面的进化树出现了一条条很长的英文。解决办法也很容易,直接将“>”后面序列名称改成简称就行了。比如第一条序列是我们的目的基因SD1,第二条序列可以按照简写直接命名成AtGA20ox1,其中的At是拟南芥的简写,GA20ox1表示赤霉素20氧化酶1。这也是UniProt网站上蛋白序列的命名规则。

另外一个问题是假阳性的问题,既然我们的目的基因是一个赤霉素20氧化酶,那么我们只需要关注这个家族的蛋白,因此像下面这些蛋白序列则需要手动删除。

2.2 重新构建进化树
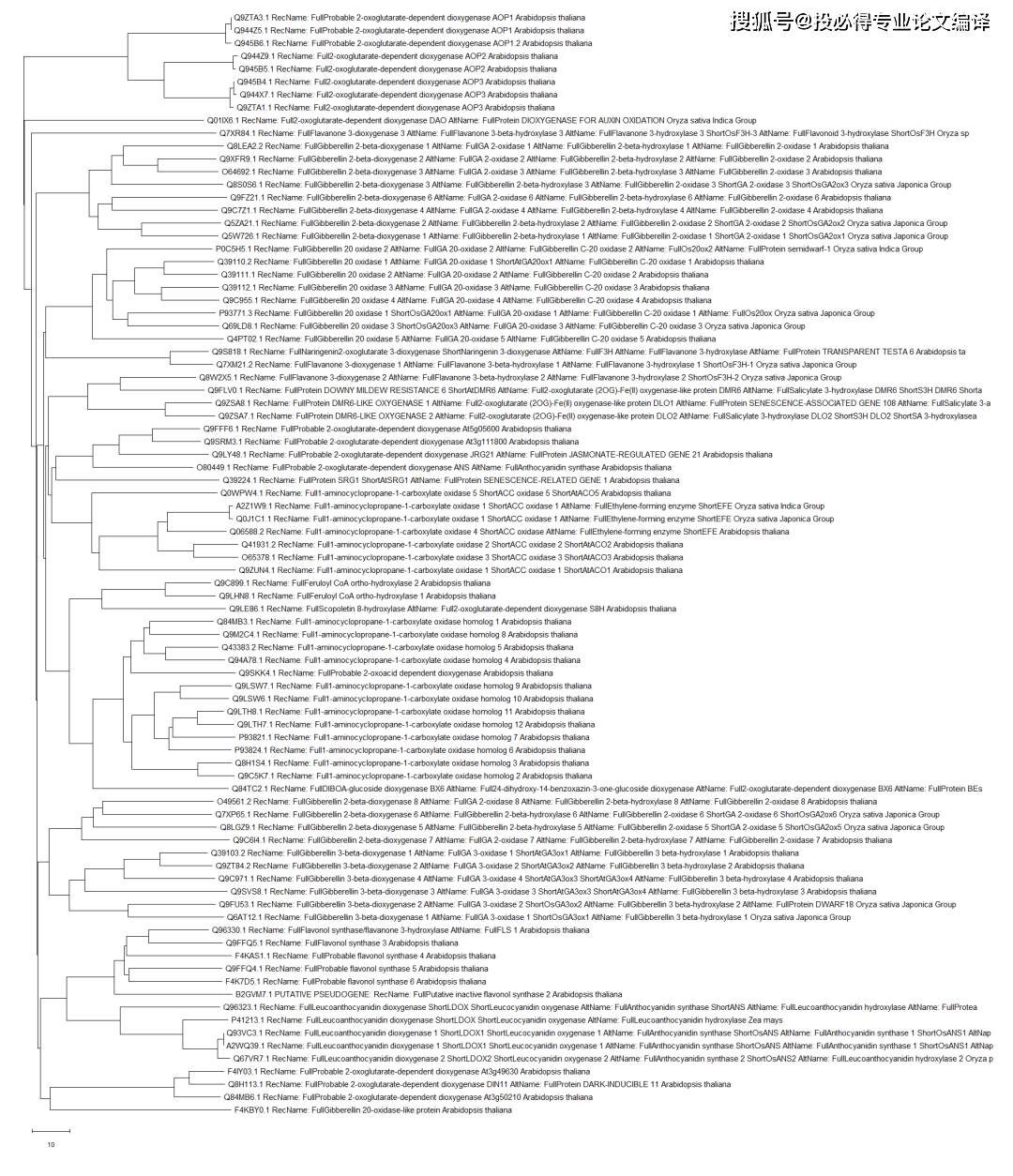
序列处理完成后,按照上面的方法重新构建进化树,这样就得到了我们的进化树2.0版本。看起来好看多了!

3.进化树3.0版本
MEGA软件本身提供了很多进化树编辑的工具,在生成进化树的Tree Explorer窗口提供了多种小工具,这里仅列举几种。
3.1 改变进化树形状
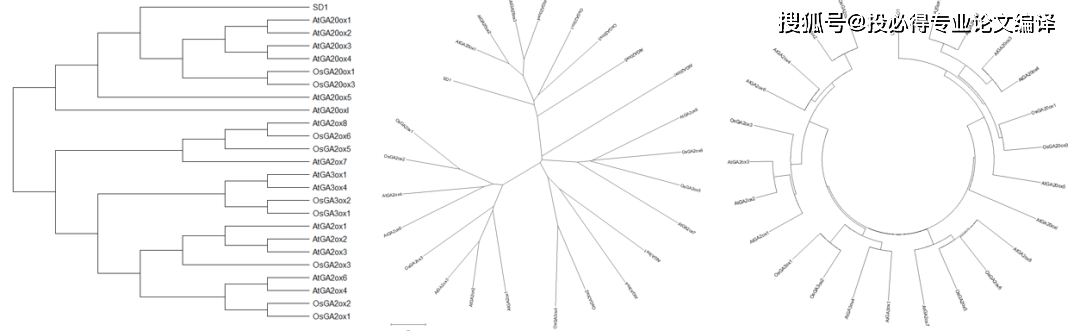
上面一排有两个树形的按钮,可用于改变进化树的形状,包括矩形、辐射形和圆圈形(下图从左到右),也可以调整序列名称是否对齐。
3.2 显示分支节点属性
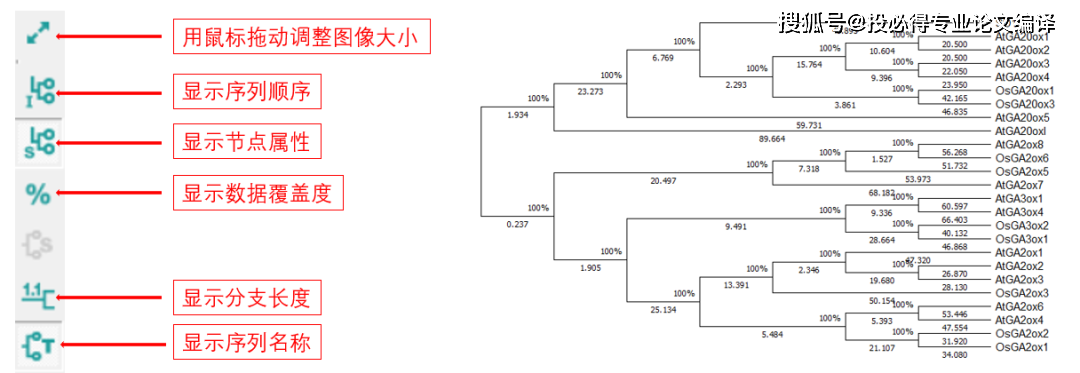
Tree Explorer窗口左侧一栏也有大量的选项,下图列举了部分功能。这些按钮能在进化树上显示出相应的信息,常用的有节点属性、分支长度和序列名称等信息。
3.3 调整序列名称字体和颜色
单击上方工具栏中的工具按钮,选择【Lables】,在弹出的窗口中可修改字体和颜色,并且能给相应的序列添加不同的符号。
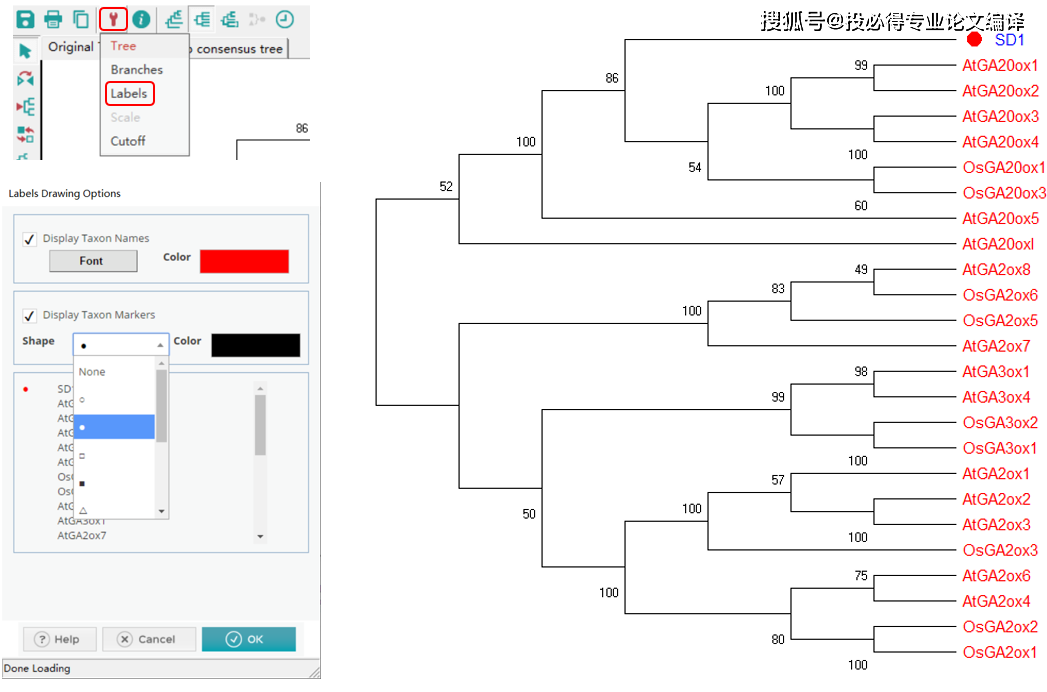
4.进化树终极版本
调整好这些参数后我们的进化树已经十分美观了,但这就够了吗?并不是!这里给大家安利一个专门管理和编辑进化树的网站ITOL(http://itol.embl.de/)。请收藏这个神奇的网站,它能够按照预设好的注释文件编辑进化树,并且能够导出可编辑的矢量图文件。下面简单介绍其用法。
4.1 上传文件
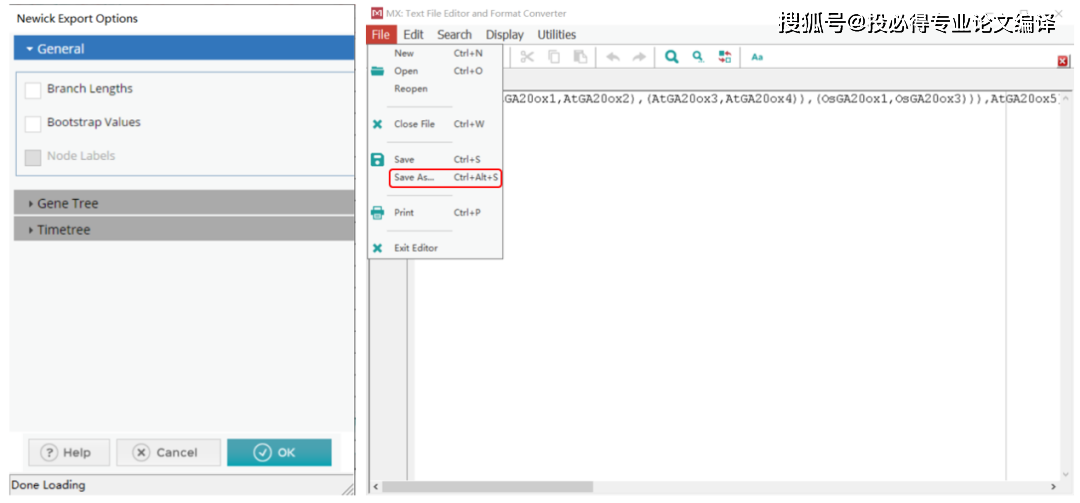
ITOL只能识别纯文本文件,包括Newick、Nexus、PhyloXML、Text和Jplace等,其中最常见的是Newick文件。MEGA就能导出Newick文件,方法是在Tree Explorer窗口中单击【File】→【Export Curren Tree (Newick)】,在新弹出的窗口中选择General,然后在新的窗口中另存为文件即可。
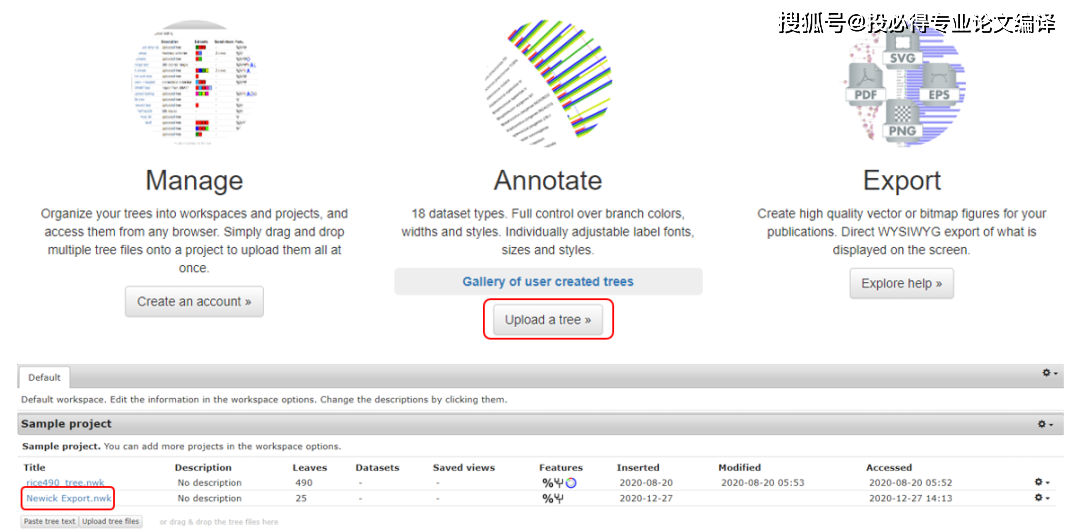
打开ITOL网站,第一次使用需要先注册。注册完成后选择【Upload a tree】,将上面生成的Newick文件上传。
4.2 编辑进化树
点击相应的进化树文件即可进入编辑窗口,如下图所示,左侧是进化树文件,右侧是工具栏。ITOL中提供了各种美化进化树的方法,工具栏中可以调整字体、颜色、分支长度等参数,用鼠标右键点击序列名称也可以调整字体。读者可自行尝试。

4.3 导出文件
ITOL支持包括PNG、PDF、SVG等多种格式文件导出。导出按钮在右侧工具栏中的Export选项。导出后的PDF文件能在其他文件如AI(参看科研论文作图系列-从PPT到AI (一))中进一步编辑和美化。
结语
关于MEGA和ITOL构建进化树就先说到这里。事实上ITOL是一个非常强大的进化树编辑工具,还能够识别进化树注释文件。读者可在ITOL网站上的help中查看注释文件的说明和实例。如果想要进一步了解ITOL的读者,也欢迎留言告诉小编。
- 本文固定链接: https://maimengkong.com/image/830.html
- 转载请注明: : 萌小白 2022年1月22日 于 卖萌控的博客 发表
- 百度已收录
