火山图(Volcano plot)是一种比较“远古”的一种散点图,广泛应用于转录组、蛋白组等组间差异分析结果的展示。图表的横轴一般展示差异倍数的变化,而纵轴表示差异分析结果的可靠性。
我这里对传统的火山图做了进一步的优化,比如通过调整点的大小突出展示感兴趣基因对应的点,并将感兴趣基因的名称以标签的方式展示出来,如下。
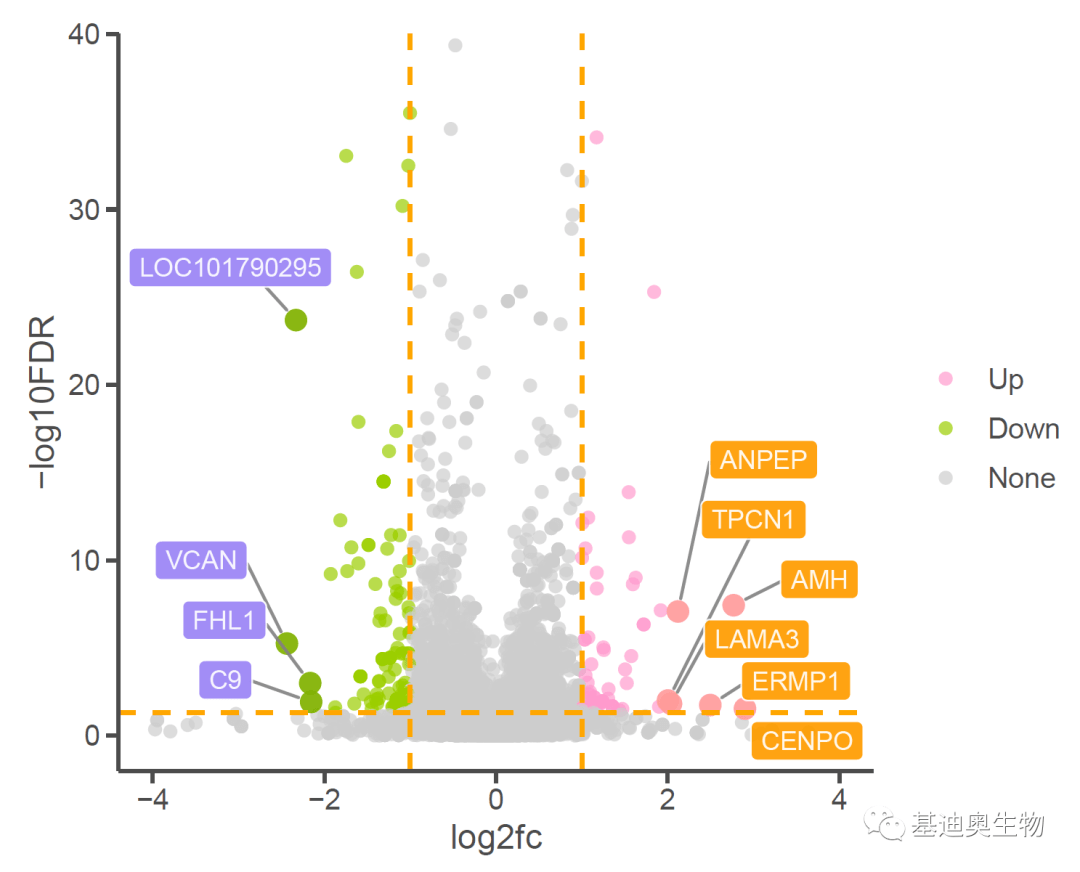
那么,如何绘制这般个性又好看的火山图呢?我这里主要用到的还是最常见的ggplot2包。
#设置工作目录;
setwd( "C:/Users/MHY/Desktop/火山图的绘制")
dir
#读入数据;
data<- read.csv( "protein_dia_simple.csv",header = T)
head(data)
#载入相关的R包;
library(dplyr)
library(ggplot2)
library(ggrepel)
#转成tibble便于后续使用,去掉不需要的列;
dt<- as_tibble(data[c(-2,-3,-5)])
head(dt)
#对Q值取对数;
dt$log10FDR <- -log10(dt$qvalue)
#生成显著上下调数据的分组标签;
dt$group <- case_when(dt$log2fc > 1 & dt$qvalue < 0.05 ~ "Up",
dt$log2fc < -1 & dt$qvalue < 0.05 ~ "Down",
abs(dt$log2fc) <= 1 ~ "None",
dt$qvalue >= 0.05 ~ "None")
head(dt)
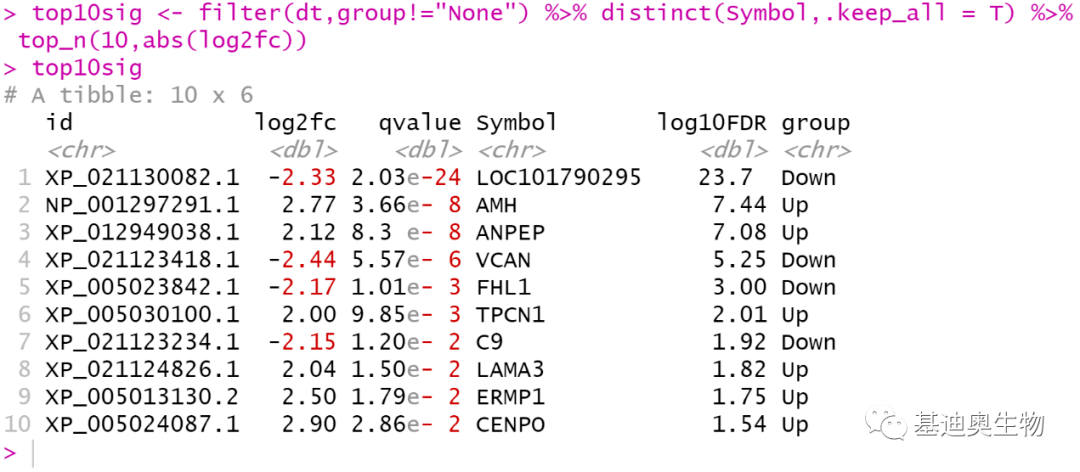
#获取表达差异最显著的10个基因;
top10sig<- filter(dt,group!= "None") %>% distinct(Symbol,.keep_all = T) %>% top_n(10,abs(log2fc))
top10sig
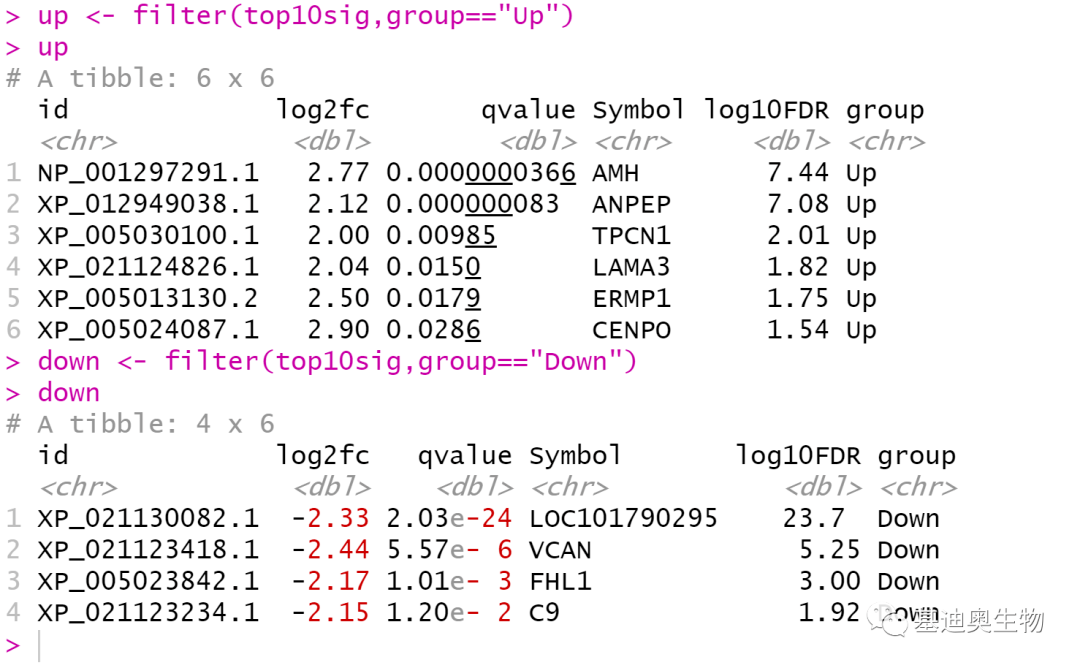
#将差异表达Top10的基因表格拆分成up和down两部分;
up<- filter(top10sig,group== "Up")
up
down<- filter(top10sig,group== "Down")
down
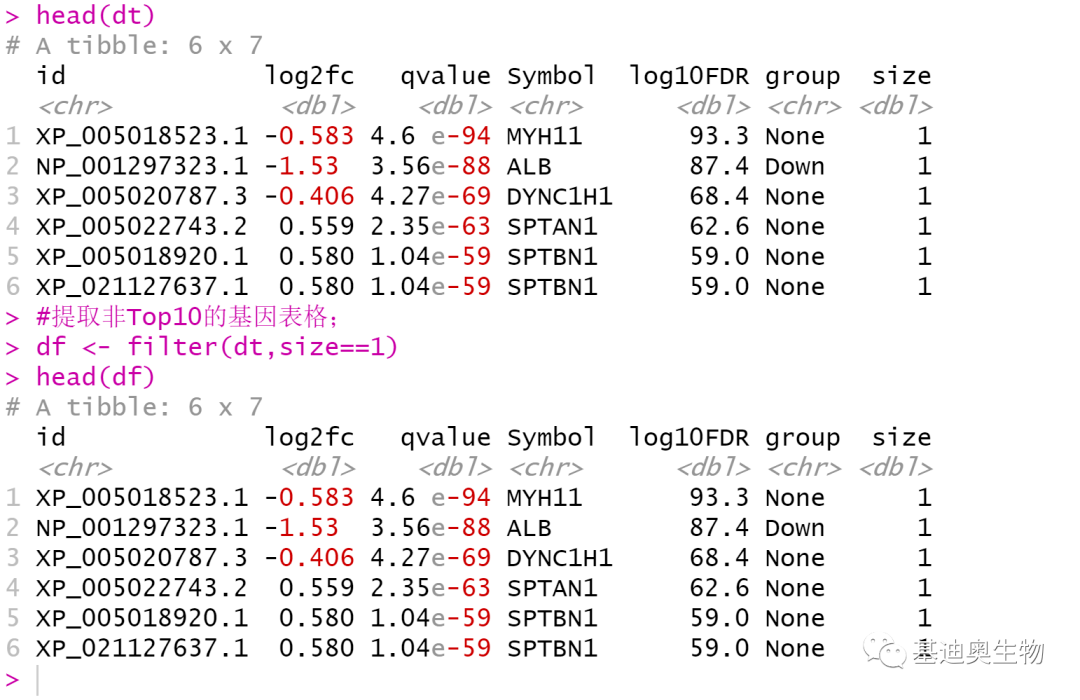
#新增一列,将Top10的差异基因标记为2,其他的标记为1;
dt$size <- case_when(!(dt$id %in% top10sig$id)~ 1,
dt$id %in% top10sig$id ~ 2)
head(dt)
#提取非Top10的基因表格;
df<- filter(dt,size==1)
head(df)
#指定绘图顺序,将group列转成因子型;
df$group <- factor(df$group,
levels= c( "Up", "Down", "None"),
ordered= T)
#开始绘图,建立映射;
p0<-ggplot(data=df,aes(log2fc,log10FDR,color=group))
#添加散点;
p1<- p0+geom_point(size=1.6)
p1
#自定义半透明颜色(红绿);
mycolor<- c( "#FF9999", "#99CC00", "gray80")
p21<- p1 + scale_colour_manual(name= "",values=alpha(mycolor,0.9))
p21
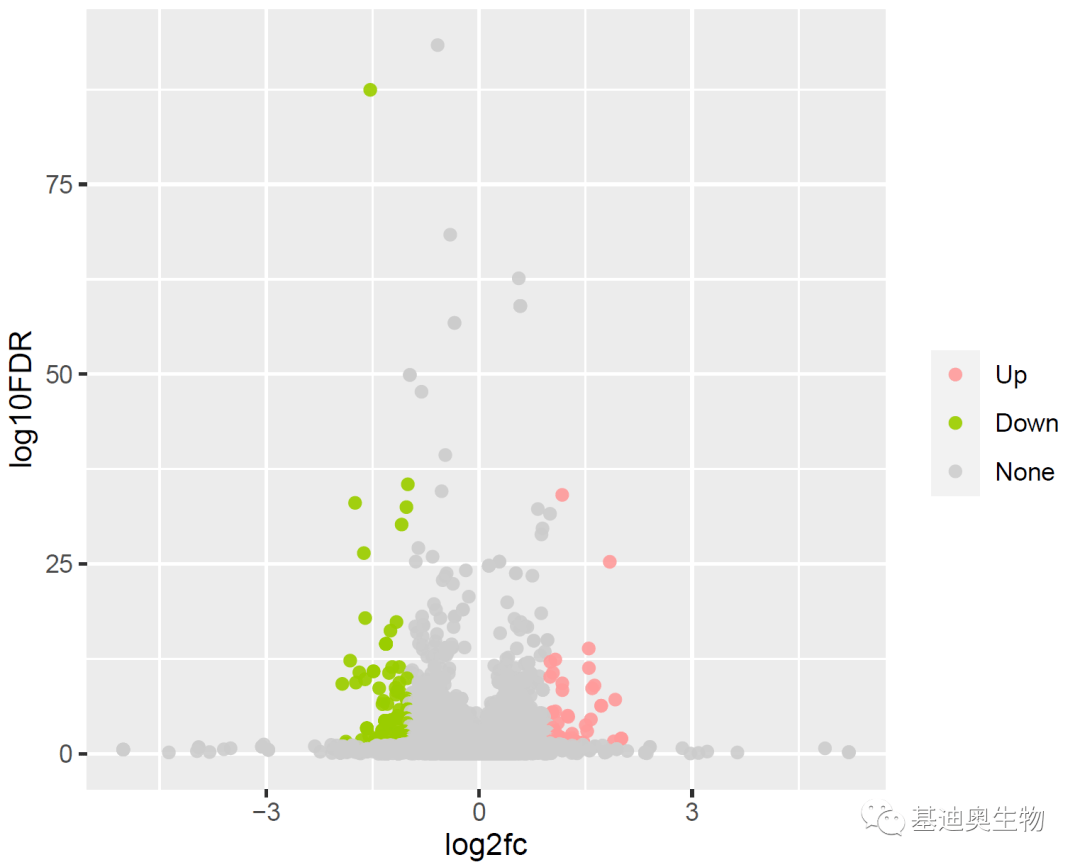
#其他配色方案:
mycolor<- c( "#FF99CC", "#99CC00", "gray80")
p22<- p1 + scale_colour_manual(name= "",values=alpha(mycolor,0.7))
p22
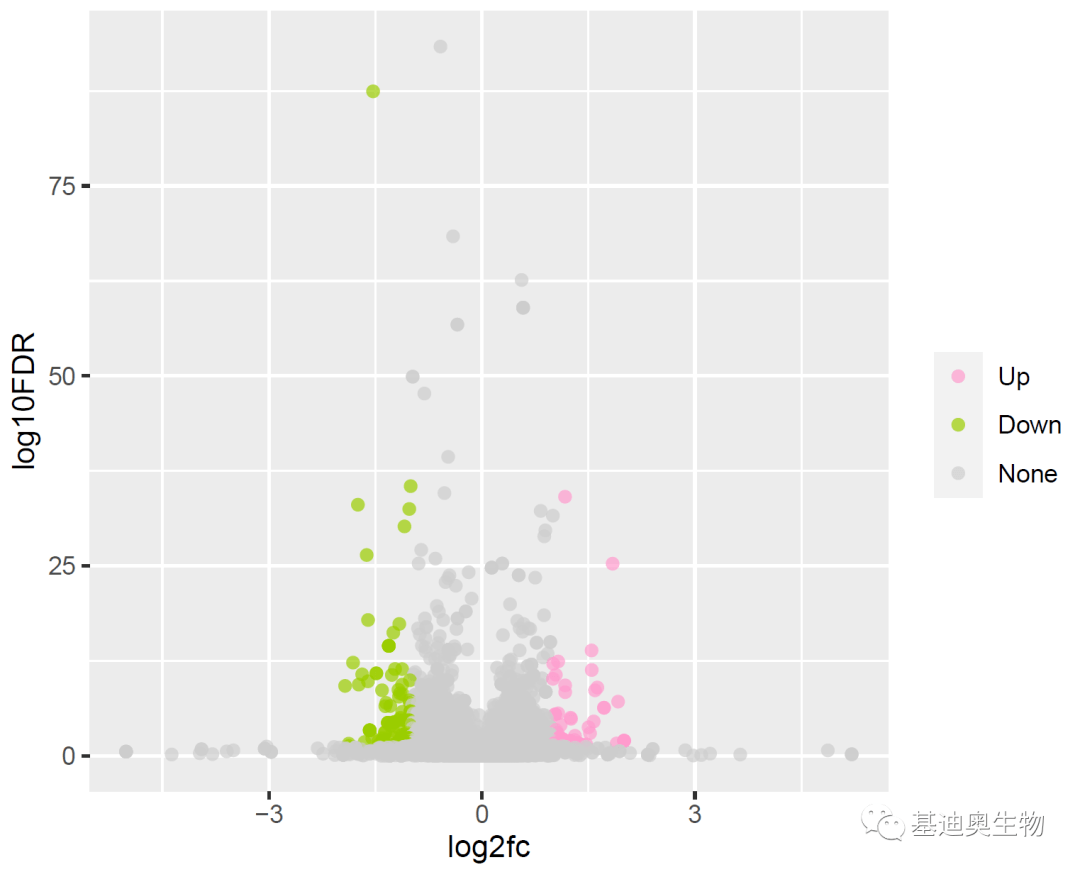
#继续添加Top10基因对应的点;
p2<- p22+geom_point(data=up,aes(log2fc,log10FDR),
color= "#FF9999",size=3,alpha=0.9)+
geom_point(data=down,aes(log2fc,log10FDR),
color= "#7cae00",size=3,alpha=0.9)
p2
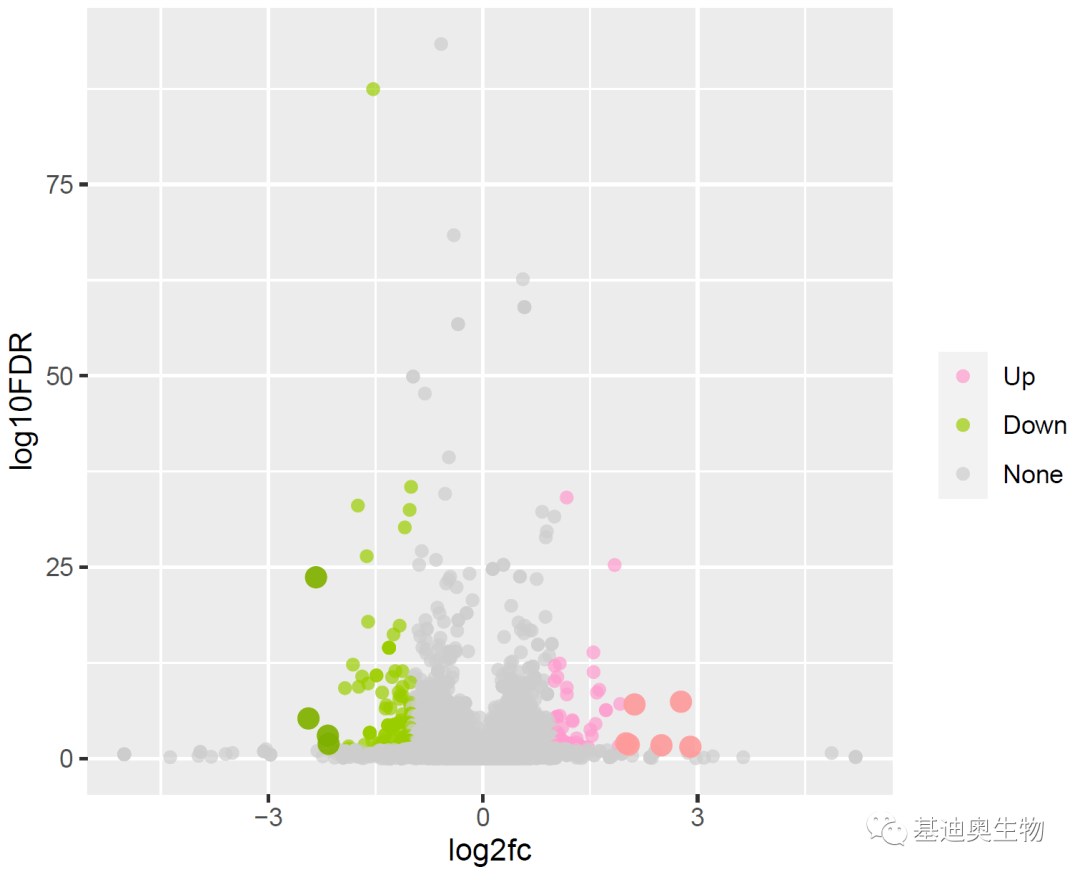
调整横轴和纵轴绘图区域的范围,重点突出Top10基因;设置y轴范围(上下两端的空白区域设为1),修改刻度标签。
#expansion函数设置坐标轴范围两端空白区域的大小;mult为“倍数”模式,add为“加性”模式;
p3<-p2+labs(y= "-log10FDR")+
scale_y_continuous(expand=expansion(add = c(2, 0)),
limits= c(0, 40),
breaks= c(0,10,20,30,40),
label= c( "0", "10", "20", "30", "40"))+
scale_x_continuous(limits = c(-4, 4),
breaks= c(-4,-2,0,2,4),
label= c( "-4", "-2", "0", "2", "4"))
p3
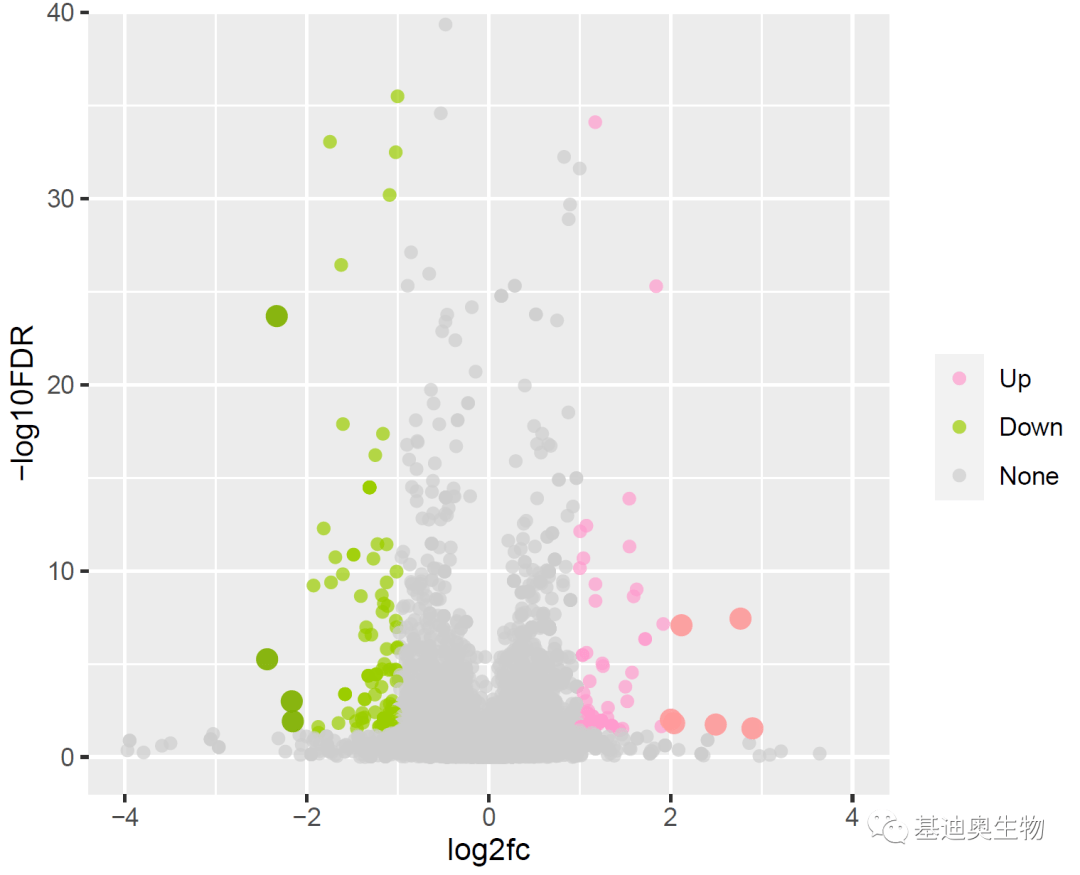
接下来为top10基因对应的散点添加指引线,我们主要用到geom_text_repel和geom_label_repel这两个函数,下面是比较重要的一些参数:
nudge_x/y:数据点与相应数据标签的距离,例如1表示标签在点右/上的1个单位处,而-2.2表示标签在点左/下2.2个单位处;
direction:标签分布方向,x表水平分布,y 表示垂直分布,both 表示随机分布;
segment.size:指定线段的粗细;
point.padding:表示点周围的空余区域,决定连接线端点到到数据点中心的距离,单位为line。
#添加箭头;
set.seed(007)
p4<- p3+geom_text_repel(data=top10sig,aes(log2fc,log10FDR,label=Symbol),
force=80,color= "grey20",size=3,
point.padding = 0.5,hjust = 0.5,
arrow= arrow(length = unit(0.01, "npc"),
type= "open", ends = "last"),
segment.color= "grey20",
segment.size=0.2,
segment.alpha=0.8,
nudge_x=0,
nudge_y=1)
p4
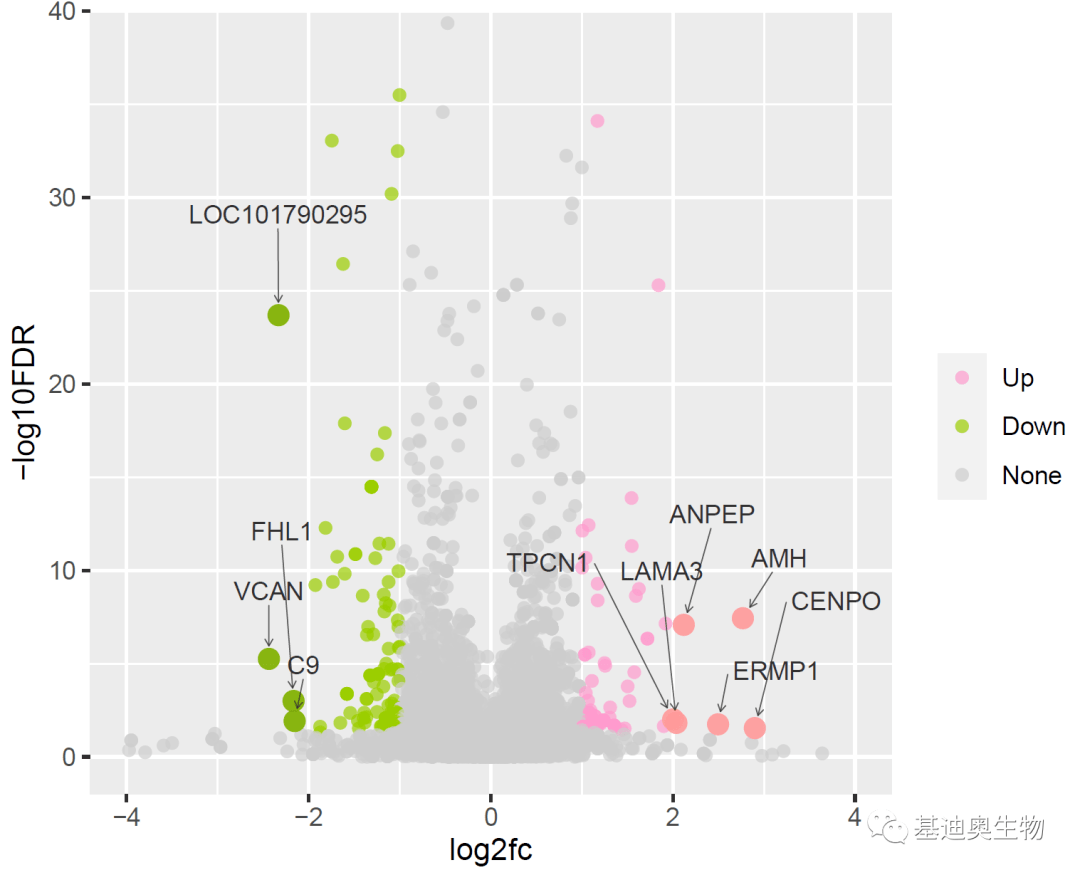
#自定义图表主题,对图表做精细调整;
top.mar=0.2
right.mar=0.2
bottom.mar=0.2
left.mar=0.2
#隐藏纵轴,并对字体样式、坐标轴的粗细、颜色、刻度长度进行限定;
mytheme<-theme_classic+
theme(text=element_text(family = "sans",colour = "gray30",size = 12),
axis.line = element_line(size = 0.6,colour = "gray30"),
axis.ticks = element_line(size = 0.6,colour = "gray30"),
axis.ticks.length = unit(1.5,units = "mm"),
plot.margin=unit(x=c(top.mar,right.mar,bottom.mar,left.mar),
units= "inches"))
#应用自定义主题;
p4+mytheme
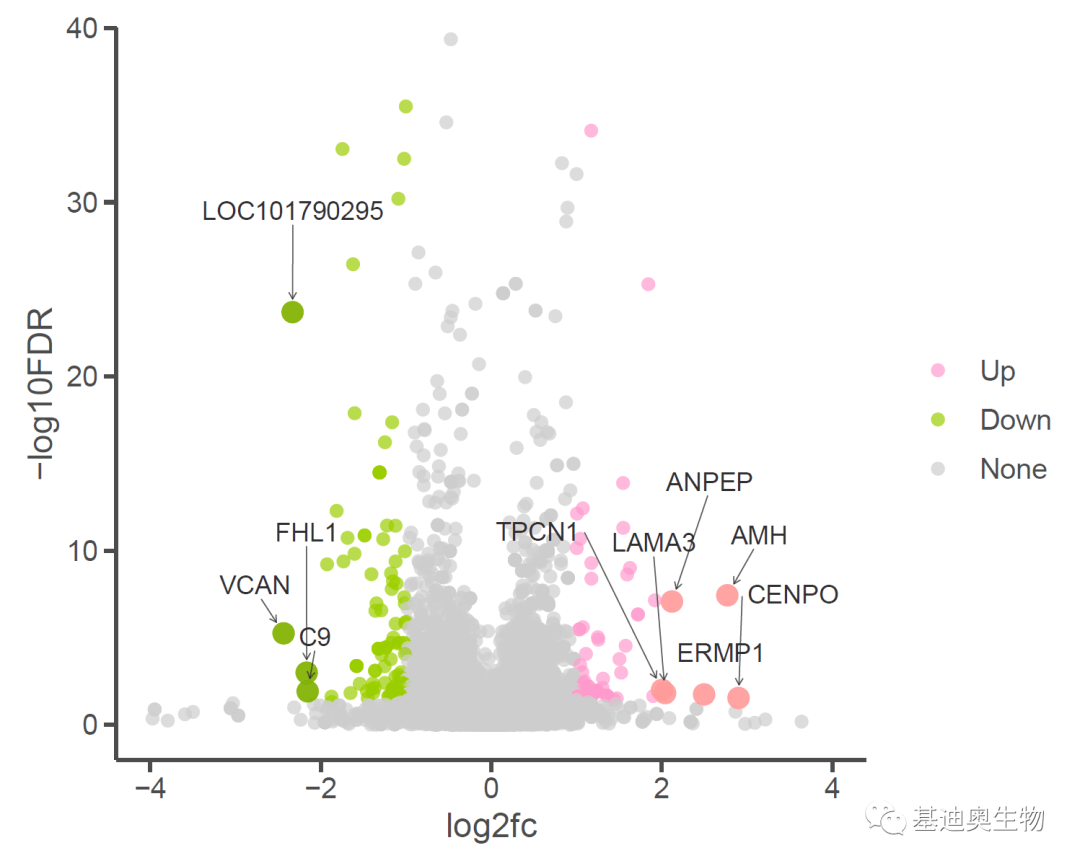
#添加辅助线;
p5<- p3+geom_hline(yintercept = c(-log10(0.05)),
size= 0.7,
color= "orange",
lty= "dashed")+
geom_vline(xintercept = c(-1,1),
size= 0.7,
color= "orange",
lty= "dashed")
p5
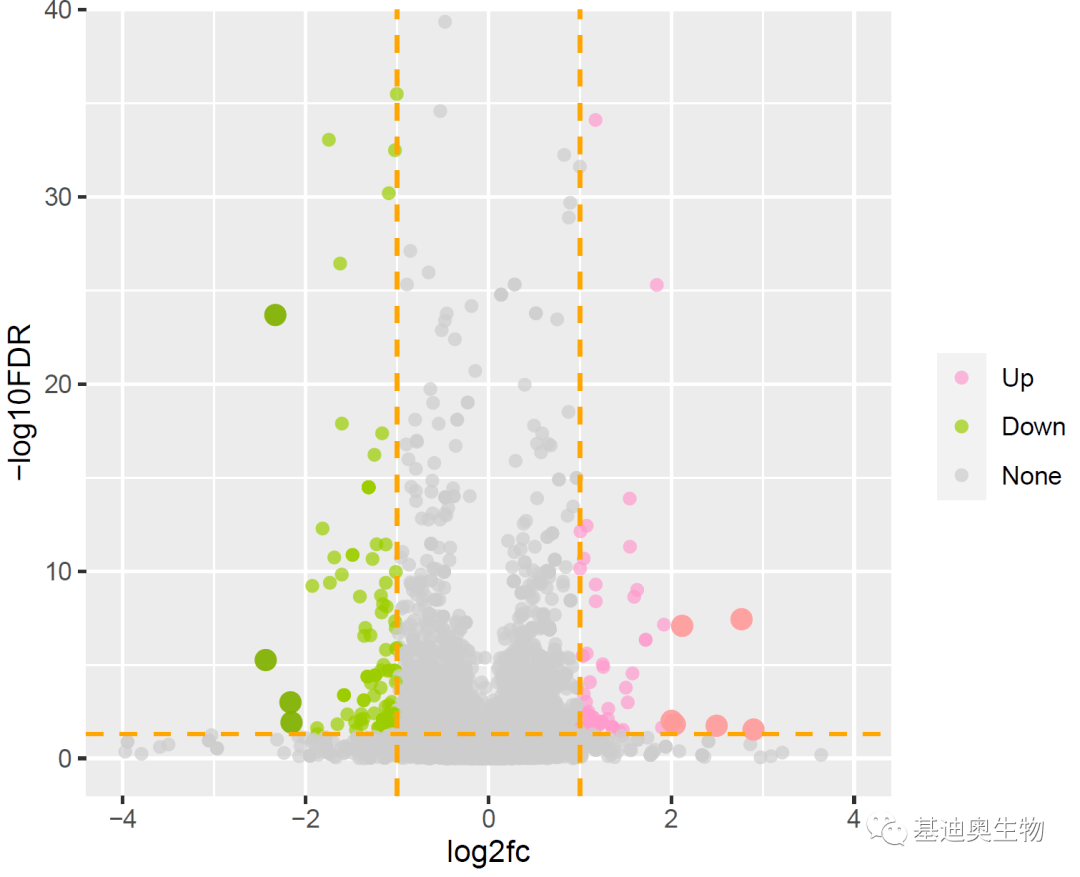
#添加其他样式的标签;
#为了方便自定义左右区域的标签,这里使用up、down两个独立的子表格;
p6<- p5+geom_label_repel(
data= up,aes(log2fc,log10FDR,label=Symbol),
nudge_x= 1,
nudge_y= 5,
color= "white",
alpha= 0.9,
point.padding = 0.5,
size= 3,
fill= "#96C93D",
segment.size = 0.5,
segment.color = "grey50",
direction= "y",
hjust= 0.5) +
geom_label_repel(
data= down,aes(log2fc,log10FDR,label=Symbol),
nudge_x= -1,
nudge_y= 3,
color= "white",
alpha= 0.9,
point.padding = 0.5,
size= 3,
fill= "#9881F5",
segment.size = 0.5,
segment.color = "grey50",
direction= "y",
hjust= 0.5)
#应用自定义主题;
p7<- p6+mytheme
p7
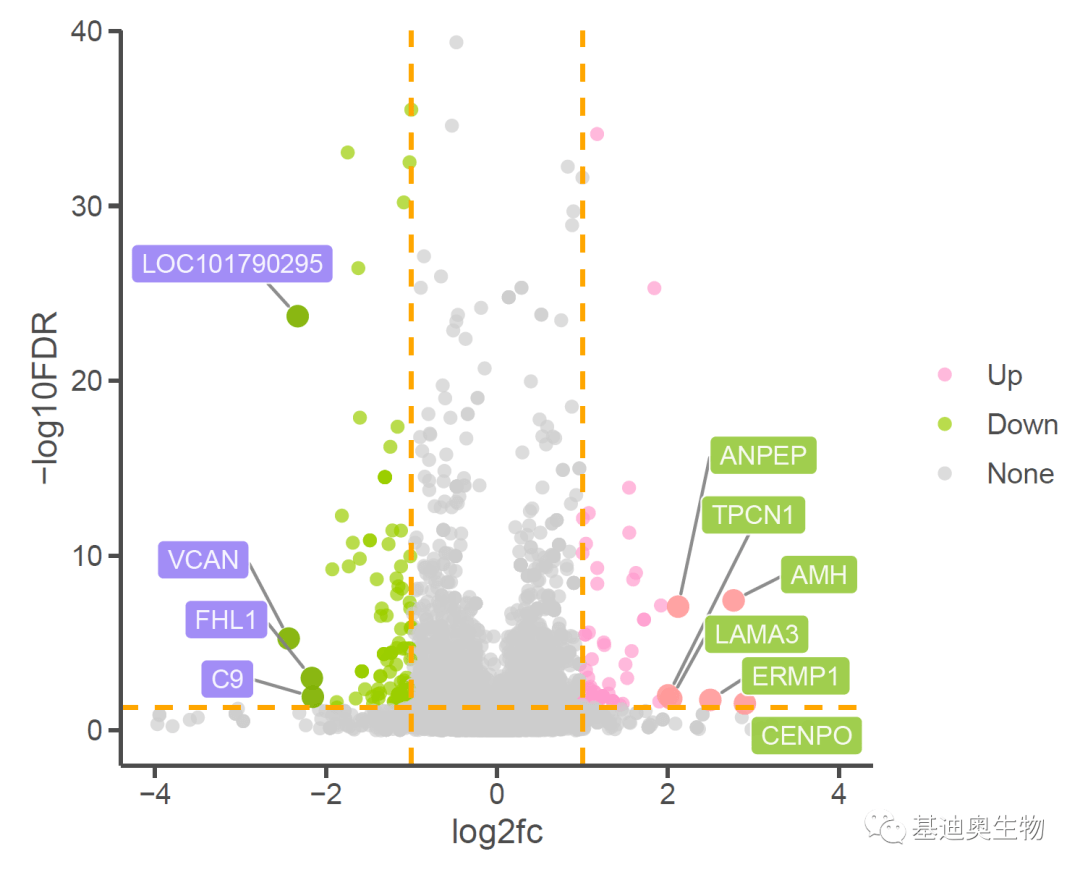
好啦,以上就是今天分享的全部内容,你学会了吗?
转自:基迪奥生物
- 本文固定链接: https://maimengkong.com/image/1055.html
- 转载请注明: : 萌小白 2022年6月28日 于 卖萌控的博客 发表
- 百度已收录
