本文根据 2016 年 8 月复旦大学倪挺教授在「表观基因组学暑期国际讲习班」中的报告整理而成,本文采用第一人称叙述,文中的“我”皆指倪挺教授。报告原视频详见: 表观遗传系列视频13 | 复旦倪挺:表观遗传调控与基因剪接(附PPT) ,视频全长约 2h34min,文字约 1.6 万字。虽然是四年前的视频,但内容依然不过时,可帮助我们快速建立对转录水平表观调控的认识。
倪挺博士,复旦大学博士生导师。2000 年获北京大学生命科学学院生物化学及分子生物学系学士学位,2000-2006 年硕博连读于北京大学生命科学学院,并于 2006 年获植物学博士学位。2007-2010 年在美国杜克大学基因组科学与政策研究所从事博士后研究。2010-2012 年在美国国立卫生研究院 (NIH) 担任助理研究员 (Research Fellow)。2012 年受聘为复旦大学生命科学学院研究员。
以下为正文:
大家好,今天跟大家分享「表观遗传与基因剪接」。
我此前的研究主要针对转录过程开发一些方法,探究转录如何起始、中间调控机制以及 3’-端的加尾,此外还包括 3’-UTR和反义 RNA 的调控等。2012 年来到复旦大学后,我主要选择了两个体系:细胞衰老和细胞活化,聚焦 RNA 水平的调控,特别关注内含子保留这种可变剪接类型,及其在 T 细胞活化过程中的功能;此外,还研究选择性多聚腺苷酸化(alternative polyadenylation),即决定 RNA 在哪个位置加上 Poly(A) 尾巴的机制,从而对 mRNA 的命运产生影响。研究手段主要采用干湿结合,试图观察新现象并探讨其生物学机制。
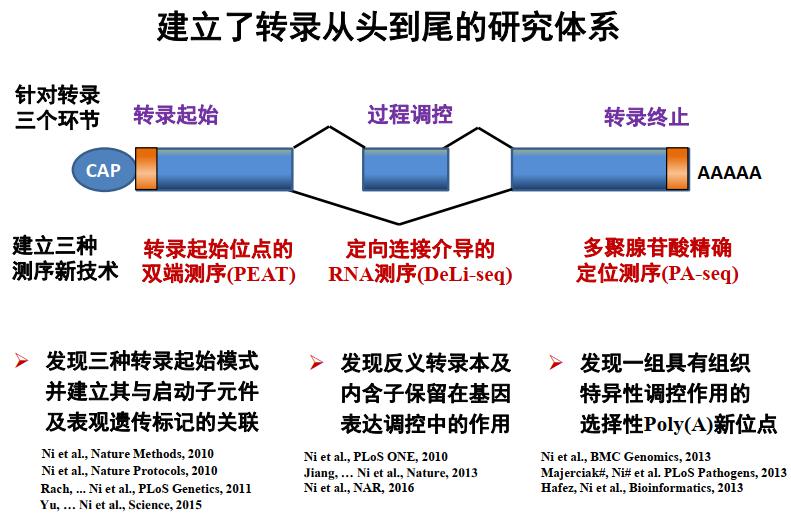
图1. 倪挺研究员的主要研究内容
此前 颉伟老师其实已经把 DNA 甲基化和组蛋白修饰之间的关系 讲得比较清楚了,DNA 甲基化可以影响组蛋白修饰,反过来,组蛋白修饰也可以影响 DNA 甲基化。我们今天讲的选择性剪接,能否与 DNA 甲基化和组蛋白修饰联系在一起呢?
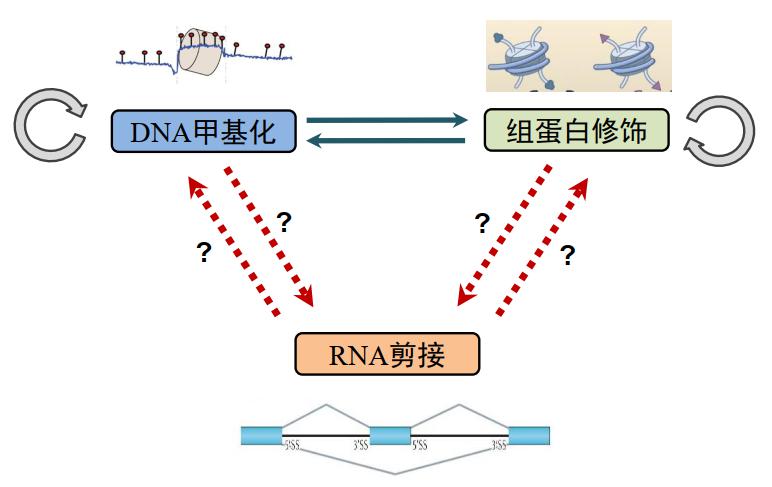
图2.可变剪接与表观遗传修饰之间的联系
通过这个课程,希望大家思考图 2 中的这四个箭头是否成立?如果成立,证据是什么?可能的机制是什么?这四个问题的内容是: (1) 组蛋白修饰是否可以影响 RNA 剪接?(2) RNA 剪接是否可以影响组蛋白修饰?(3) DNA 甲基化是否会影响 RNA 剪接?(4) RNA 剪接本身对 DNA 甲基化有没有影响?
针对这几个主要问题,我的课分为以下六个部分: (1) 首先回顾可变剪接的生物学意义;(2) 有了意义之后,我们想要更好地去检测它,从单基因的层面如何检测和验证,以及如何检测整个基因组的变化;(3) 如果存在这样的变化,上游的调控机制是什么?是什么东西导致了它的改变?(4) 组蛋白修饰和 (5) DNA 甲基化跟它之间的关系是怎样的?(6) 染色质修饰与基因转录终止的关系。选择性剪接是中间的环节,mRNA 的加工最后一个环节是加上 Poly(A) 尾巴,必须完成这个过程,mRNA 才能发挥它的作用。
可变剪接的生物学意义
现代的遗传学主要探究基因型和环境如何相互作用,从而决定生物的表型。在这个框架下,中心法则至关重要,也就是说,基因型是如何通过表达产生蛋白质,从而对表型产生影响。在这个过程中,环境可以通过多种方式发挥作用,比如可能引起 DNA 的改变;除了引起 DNA 的改变,环境更多地只影响基因的表达,那么通过什么方式呢?我们认为,DNA 甲基化和组蛋白修饰以及其他的因素在里面起到非常重要的作用,即可以通过影响中心法则的中间环节影响表型。
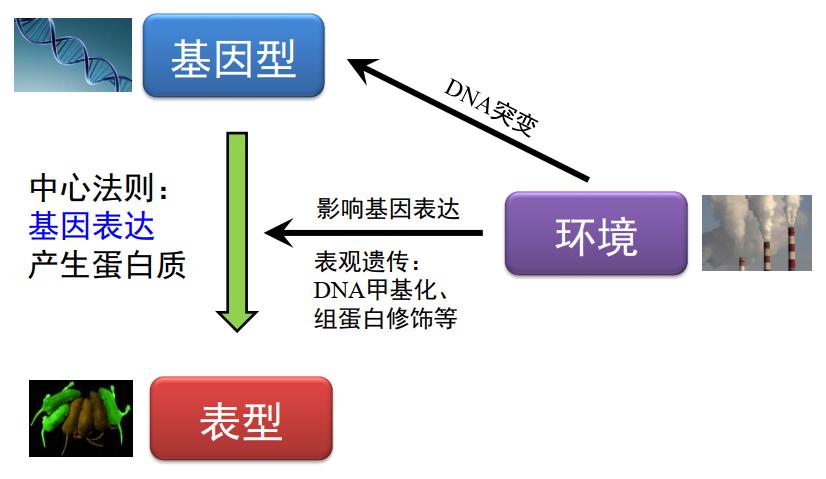
图3. 基因和环境共同作用决定表型
中心法则看起来非常简单,DNA 通过转录产生 RNA,RNA 通过翻译产生蛋白质。但在这个简单的过程中,存在非常复杂的调控。在原核和真核生物中,虽然执行的都是中心法则,但调控不一样。在原核生物中,转录和翻译几乎是同时进行的,转录出来的 RNA 马上有核糖体结合上去翻译出蛋白质。但真核生物有细胞核,转录出来的 mRNA 需要加工为成熟的 mRNA,被转移到细胞质中,才能发挥它的作用;在这个过程中,存在位置的调控;另外,mRNA 还面临着非常复杂的加工。因此,原核和真核生物在遗传信息的流动和调控上是非常不一样的。
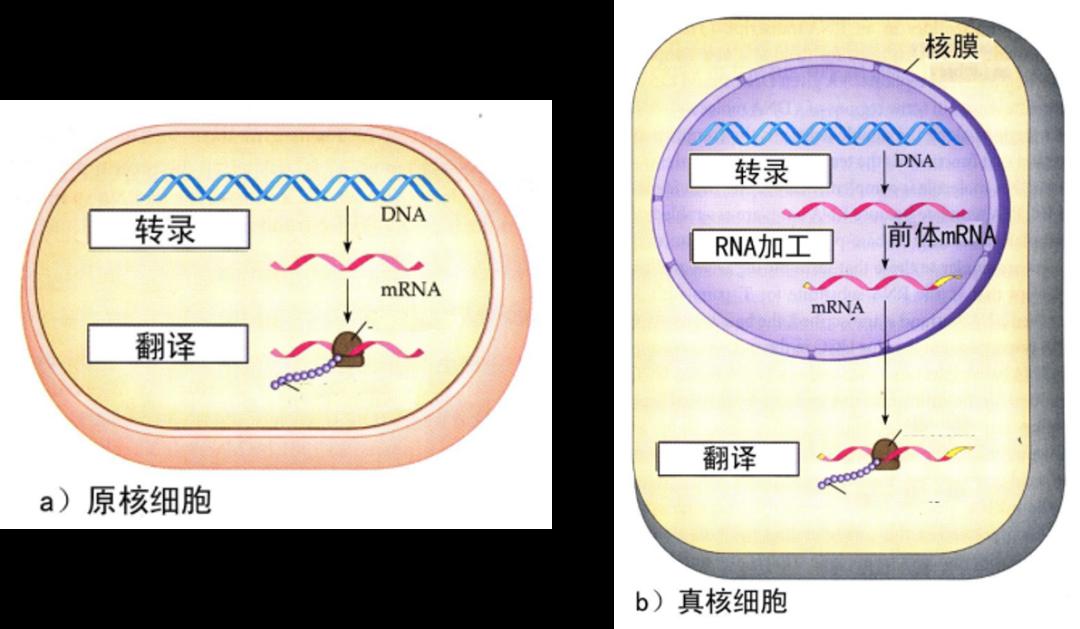
图4. 遗传信息流在原核与真核细胞间的差别
我们仔细看一下真核生物的基因表达调控 (图5),这是一个多步骤的调控,分为细胞核和细胞质中的调控。在细胞核里面,DNA 甲基化和染色质结构的改变可调控基因转录,还存在转录的起始和转录过程的调控。最初转录出来的是初始转录本,里面包含内含子,在此后的加工过程中,5’-端需要加上帽子,内含子需要切掉,还需要加上 Poly(A) 尾巴,才能变成成熟的 mRNA。在这个过程中,还有一系列的蛋白结合上去,最后输出到细胞质中。在细胞质中,会面临多重命运,一开始会翻译,翻译到一定丰度后,还会面临降解的命运,不同 mRNA 降解的速率也不一样,这里面也存在一些调控。在蛋白质水平,翻译出来的多肽也面临着蛋白质的加工和折叠,以及蛋白质激活(比如磷酸化或去磷酸化)和蛋白质降解。真核生物的基因表达调控是一个多步骤的非常复杂的过程,任何一个环节的失调都可能导致生理的失衡,最终导致疾病。
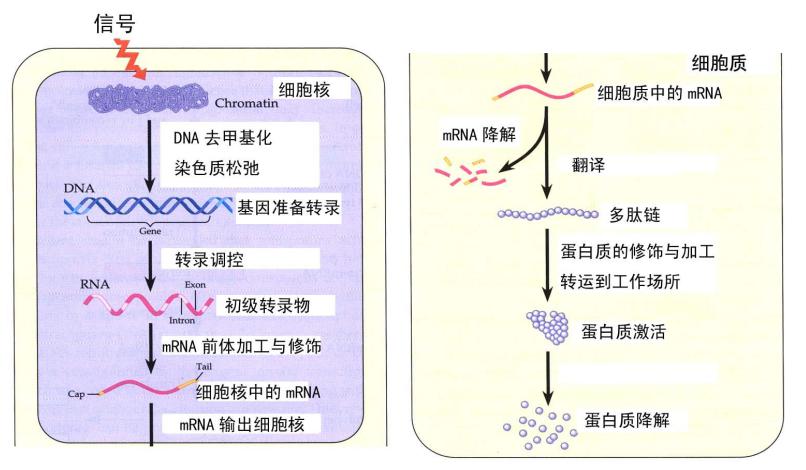
图5. 真核生物基因表达是个多步调控过程
举几个例子,第一个例子是早衰 (premature),核纤层蛋白基因 LMNA 的可变剪接的失衡是导致早衰最重要的原因。在早期对病理进行检查时发现,在父母中都没有出现突变,但是在小孩中出现了突变,即这是一种新发突变 ( de novo mutation),这种突变并不是由父代传给子代的,但这种突变会导致很严重的问题,从基因的角度来看,这种突变对转录产物造成很严重的影响。在正常情况下, LMNA 有三个外显子形成正常的剪接,翻译出的蛋白含有 C-端结构域;但是由于突变的存在,导致了剪接方式的改变,这种突变使它发生了 5’-可变剪接位点 (alternative site) 的选择,使得可翻译的区域变短了,最后使得蛋白质的 C-端完全不一样,失去定位的信号,蛋白在细胞核的分布变得紊乱。
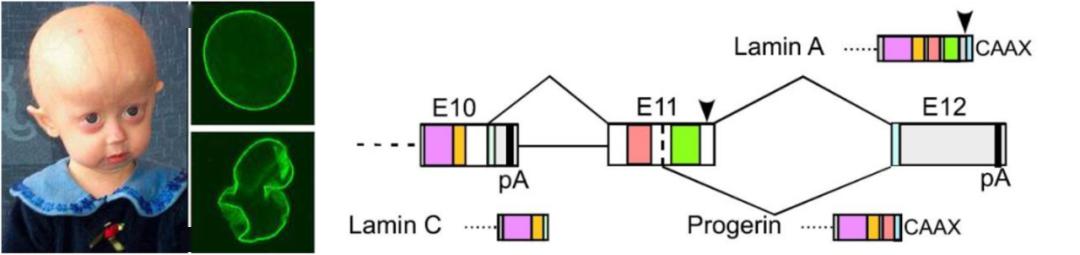
图6. 核纤层结构蛋白基因 LMNA 突变影响可变剪接,最终导致早衰
核纤层蛋白支撑着细胞的整个架构,如果发生紊乱的话,原来的细胞核构架坍塌。这种变化跟细胞的整体表达有关,核纤层的架构支撑了很多基因的表达,这样一些关键结构蛋白的失衡,导致了核纤层的坍塌,进而影响很多下游基因表达的改变,最终导致了早衰的表型。早衰儿在很早的时候就去世了。在这个例子中,我们可以看到,一个非常关键基因的可变剪接的变化就导致了早衰的产生,这提示可变剪接在生物体中十分重要。
另一个例子,不是 DNA 层面的突变,而是表达层面的。RBM4 是一个 RNA 结合蛋白,同时参与剪接过程,这个蛋白在很多癌症病人中是下降的,导致了很多基因剪接方式的改变。如果 RBM4 表达较高,它会让基因选择一种比较短的形式进行剪接;如果它的表达量低的话,会让基因选择一种比较长的形式,而这种长的形式会抑制细胞的死亡,即让细胞无限增殖,导致了癌症的发生和发展。在这个过程中,只有 RBM4 保持合适的数量,基因的剪接才会正常,细胞呈现正常的状态;基因的表达如果改变的话,会引起下游一系列的变化。
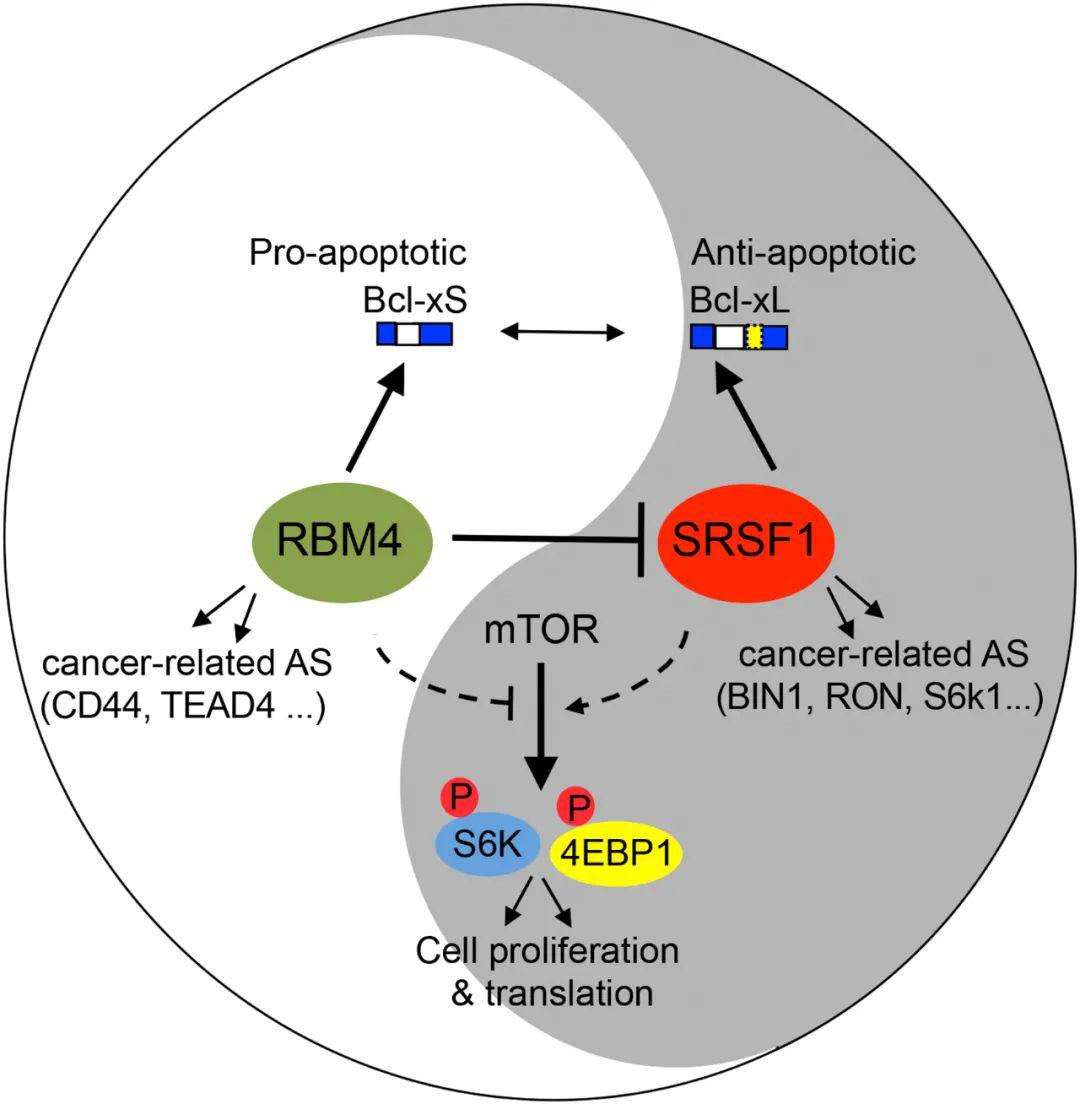
图7. RBM4 基因表达量下降促进肿瘤发展和转移[1]
特别要强调的是,剪接依赖于顺式作用元件(DNA 上的序列)和反式作用因子(与这些序列结合的蛋白)。而这两个例子,一个是可变剪接的顺式作用元件的突变,另一个是反式作用因子剪接因子的改变,这两者都可能导致剪接方式的改变,最终引起疾病。
可变剪接是蛋白质组丰富度的重要贡献者,但近来的研究发现,很多基因也存在着很多 Poly(A) 位点的选择 (alternative cleavage and polyadenylation, APA),这种选择也很大程度上贡献了蛋白质组的选择性。
在这个例子中,并没有改变编码区的序列,翻译出来的蛋白质是一样的,但是由于选择了近端的 Poly(A)位点,使得多出来的 3’-UTR 不仅被 miRNA 调控,同时也会被特殊的 RNA 结合蛋白调控。miRNA 在 3’-UTR 的结合会切割 mRNA,或者抑制 mRNA 的翻译,最终使得蛋白水平下降,有些 RNA 结合蛋白也会起到类似的效果。有的是抑制,有的是促进,效果迥异,取决于序列和蛋白本身。通过这种方式,逃脱 miRNA 的调控。
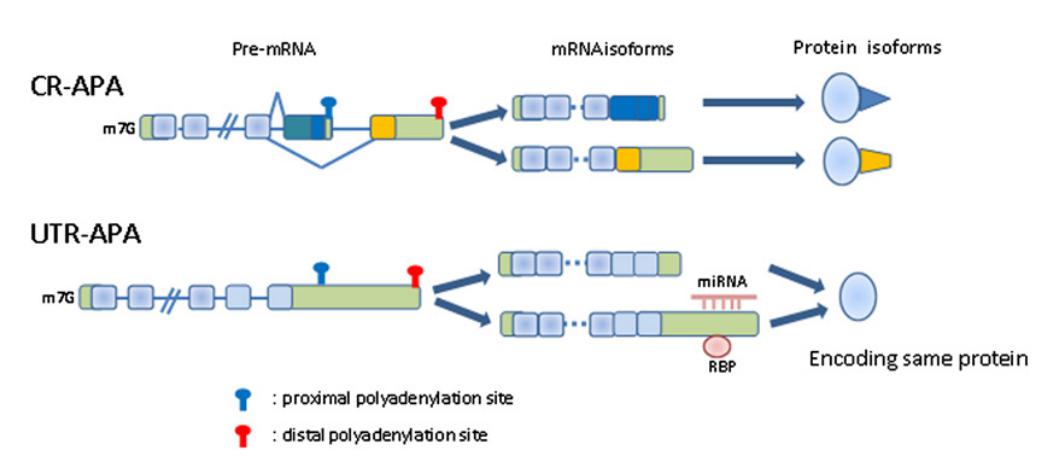
图8. mRNA 3'-端 Poly(A) 加尾异常可导致癌变 [2]
2009 年的一项研究发现,相对于癌旁细胞,癌细胞中有很多倾向于使用短的 3’-UTR 的转录本,即转录本是以短的形式存在的,比如 IGF2BP1/IMP-1 因为使用短的 Poly(A) 而逃脱了 miRNA 的调控[3],使得蛋白质水平急剧上升,导致细胞的无限增殖,是引起细胞癌变的重要因素。这个例子说明,RNA 加工过程(特别是可变剪接和末端的加工)的失衡也是引起疾病的重要因素。
可变剪接的检测及生信分析策略
研究可变剪接和 3’-端的加工具有重要的意义,可以帮我们理解很多重要的生物学事件。既然它们如此重要,那么如何更好地发现和分析呢?这就涉及到可变剪接的检测及生信分析策略。
可变剪接的类型
大部分基因含有内含子,在编码的过程中,内含子的信息不需要传递给蛋白质。在转录完成后形成的初始 mRNA 后,所有的内含子都切掉,这是一种正常的剪接方式,即组成型剪接 (constitutive splicing)。可变剪接 (alternative splicing) 跟它有一定区别,例如在正常情况下,三个外显子在一起;如果发生了可变剪接,由于某些原因中间的外显子被跳过,形成的 mRNA 不一样,这种序列的不一样可能使蛋白质中间少了一段,或者中间的外显子不是 3 的倍数的话,那么它会使得后面的蛋白质完全不一样了,改变了蛋白质的组成。
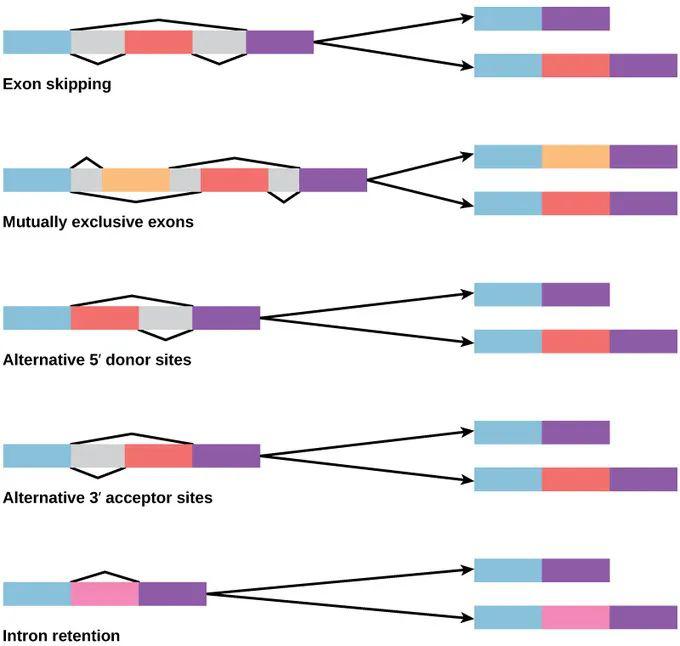
图9. 可变剪接的 5 种形式
(图片来自:https://bio.libretexts.org)
可变剪接分为哪几种类型呢?复杂的话可分为 8 种,简单的看至少有 5 种比较重要。第一种叫外显子跳跃 (exon skipping),中间的外显子可以被跳掉,取决于中间外显子两边的顺式作用元件是什么以及有哪些反式作用因子跟它结合。另一种类型,5’-端的选择,比如我们刚才举的例子,RBM4 去调控基因剪接,实际上就是通过这种方式。5’-端是看具体选择哪个位置,取决于位点的强弱。3’-端面临同样的情况,到底选择哪个3’-端,这也就是 alternative 3’ splice site;另一种比较有意思的是互斥外显子 (mutually exclusive exons),中间有两个外显子,但是细胞只会选择中间的一个来发挥作用,在蛋白质上的体现是,蛋白质的 domain 是换成这个还是那个,从而决定了蛋白质的功能。另一个是内含子保留,在很多时候,那段序列被认为是内含子,被剪切掉了,但是在特殊的情况下,细胞认为这可能是外显子的信号,在这个过程中会被保留下来,即内含子保留。
这几种方式,从造成的效应上来看,它们改变蛋白的“质”和“量”。“质”的话,在早衰症的例子中,蛋白质的 C-端完全不一样,更多的是改变了蛋白质的“质”;如何理解改变蛋白质的“量”呢,在这几个例子中,好像都没有体现。实际上,一个基因可变剪接,并不是切掉或者完全保留,而是有一部分是剪切掉,有一部分是留着的,如果说被剪切掉的 isoform(异构体)没有功能的话,与最后翻译出来的另一种剪接形式的异构体形成了竞争关系,原来 100% 都剪接掉了,现在分流给了另一个不翻译的转录本,原来那个翻译的蛋白的量就相对减少,这就是改变了特定蛋白的量。所以,可变剪接至少可以通过改变蛋白的“质”和“量”对下游产生影响。
可变剪接的鉴定及生信分析策略
如何发现可变剪接?可通过 RNA-seq 和 RT-PCR。最早的时候,大家实际上是在单基因上进行验证,可通过 RT-PCR 或 Northern Blot 来看;如果要从整个基因组水平来看的话,早期可通过 microarray,设计剪切特异性的探针就能够达到这个目的。但近年来,通过 RNA-seq 结合一些生信的办法就可以实现。
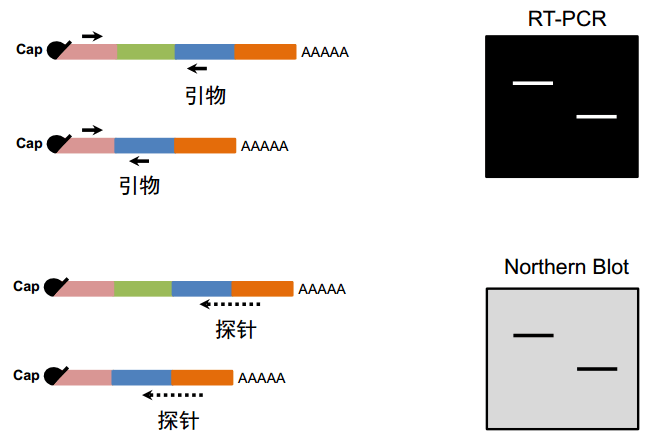
图10. 单基因水平检测可变剪接
从原理性上讲,如果有几个不同的外显子,可在不同的外显子上设计引物,或者说跨外显子的引物。如果发生了外显子跳跃,设计类似图 10 中的引物,两个不同的 isoform 扩增出来的产物是不一样的,直接跑胶就可以看到差别。实际上并不是一个有或无,可能是强和弱的关系,这个只是个示意图。对于 Northern Blot,同样也是设计 jumping 特异性的探针,跳过外显子区来设计,这是比较常规的思路。
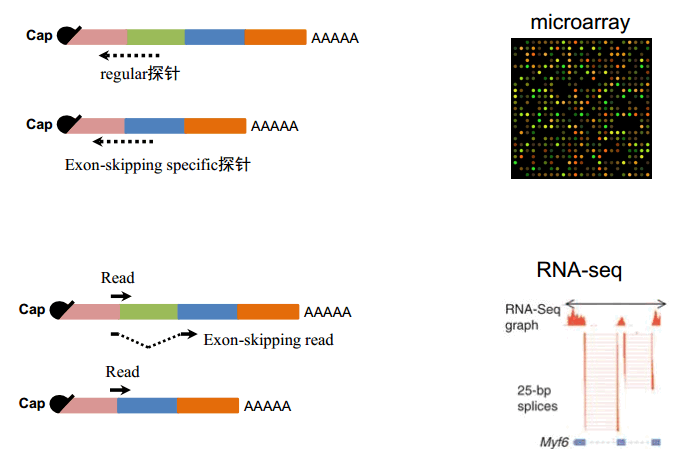
图11. 转录组水平检测可变剪接
对于整个基因组,也可以设计一些 jumping 特异的探针,如果你看到有信号的话,说明这个 isoform 存在,信号强弱可以初步反映这个异构体丰度的高低。对于 RNA-seq,它实际上测到的是一个短序列,通过这个短序列去比对到基因组和转录组上,其实可以区分。通过这样一种方式,就可以知道几个不同的 isoform 之间的比例变化。
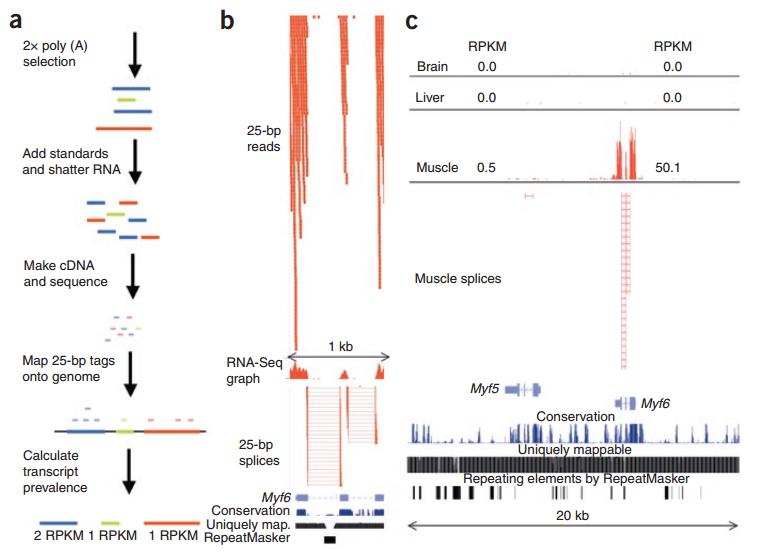
图11. RNA-seq的实验及生物信息学分析全过程 [4]
目前 RNA-seq 是发现全基因组剪接变化最有效的方法,当然这里面还存在着诸多问题和挑战。RNA-seq 通常要做两轮 Poly(A) 选择,把这些短序列去 mapping,通过分析计算有没有出现外显子的跳跃或者其他形式,还可以比较两个不同组织或者处理前后的改变,帮助你推断它可能参与的生物学过程。我想强调的一点是,数据产生的好坏对你后面的分析是非常重要的。如果实验有问题,后面的分析 pipeline 再靠谱也无济于事。比如说处理正常和疾病的两组样本,今天做正常的,明天做疾病的,这个时候再进行比较就可能出问题,因为存在批次效应。
可变剪接分析涉及的过程很多,包括怎样把这些位置 mapping 回去,怎样组装转录本以及推测其表达量,这个表达量当然是整体的表达量,还要根据 junction 的位置去预测不同 isoform 的表达量,虽然不一定那么准,但可在基因表达比较高时给你一个还不错的参考,然后还可以进行一些可视化。接下来还有一些对于剪接特异性的分析,有各种不同的软件,都有它自己的优缺点,一定要非常仔细的去看它的 manual 以及相关文章,还要和作者进行密切的交流。利用 RNA-seq 进行初步的基因表达差异和转录本分析,TopHat 目前用的比较多。
原理上是怎么区分不同的转录本的呢?举个例子,一个基因有三个转录本,A、B 两个转录本的转录起始位点是一样的,但另一个转录本 C,用了不同的转录起始位点(在内含子区域),对于使用相同的两个转录起始位点的话,它唯一的差别实际上是内含子跳跃与否。对于这样的三个基因,如果基因表达比较高,测的短序列 reads 比较高,你可以初步地对每个占多少比例进行分析,用的策略是看它特有的 reads。
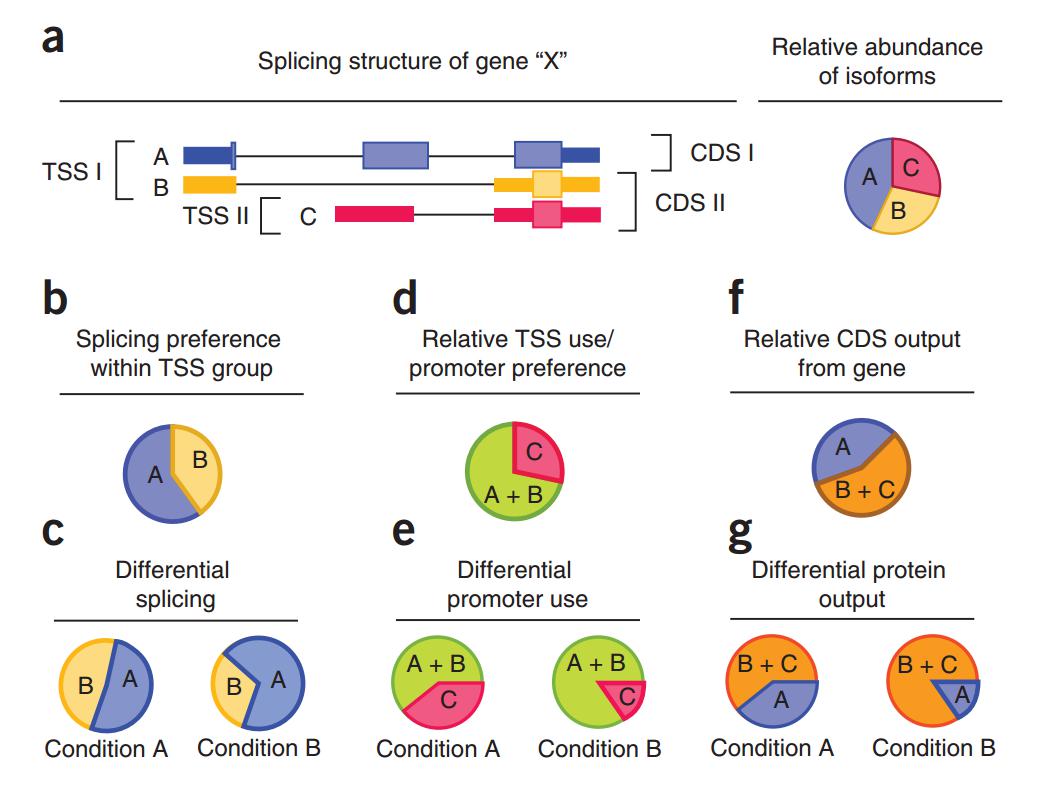
图12. 可区分高表达基因的具有明显特征的不同转录本 [5]
比如对于 A、B,如果看到第一个外显子,你至少可以知道 A、B 两个转录本的总量,那么在这两个转录本中怎样区分比例多少,就看中间外显子信号的多少,实际上就是用 isoform 特有的区域来区分;对于转录本 C,你可以看到 C 中左边的信号。通过这样的比较,你就可以区分不同的特征的比例多少,前提是基因的表达量比较高,基因测序的 reads 分布比较均匀,可以给你大致的线索,然后你再设计一些 isoform 特异性的引物来验证。
有了这些之后,你可以进行初步的推断。你可以运用一些工具画出三个或者四个不同转录本在不同细胞或者不同时期的变化。如果看到了差别,仅说明转录本存在与否,但对于生物学过程来说,这个存在与否的调控看不出来,它没有动态的改变,而如果特定的转录本存在动态改变的话,实际上是对它功能的暗示,在这个过程中,它有可能是参与了这个过程,当然也有可能是一个伴随的现象,但至少可以给你一些线索。
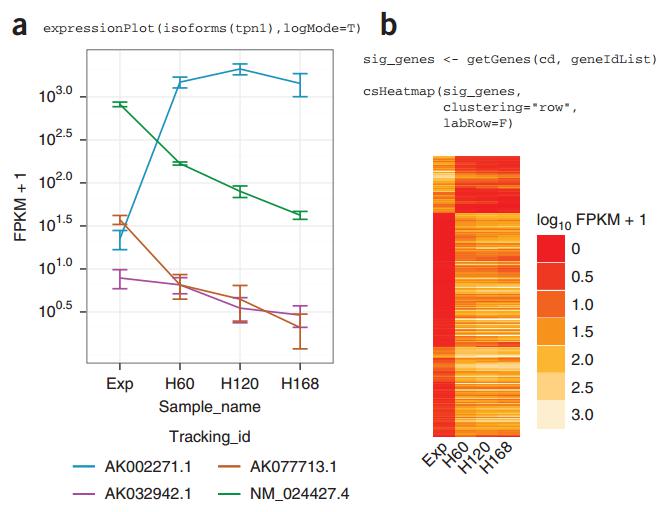
图 13. CummeRbund 工具可画出不同转录本的表达动态图 [5]
在可视化的展示(图14)中,对 regucalcin 基因来讲,两个不同状态之间基因总体的表达量差了约两倍,但如果这个基因有四种不同的 isoform,那么是每个 isoform 都增加了两倍,还是说其中只有几个 isoform 发生了改变?因为研究功能时要聚焦于不同的 isoform,不同 isoform 翻译的蛋白可能不一样,这个时候能够把不同 isoform 区分开的话,你可能就会发现,实际上其中一个 isoform 占的比例比较大,可能是它增加了两倍,所以后面的功能验证或分析就可以集中在这个 isoform 上。通过这样的可视化,你也知道原来这个 isoform 是真的存在的,通过这样一个侧面的证据可以帮助你更好地去锁定研究对象。
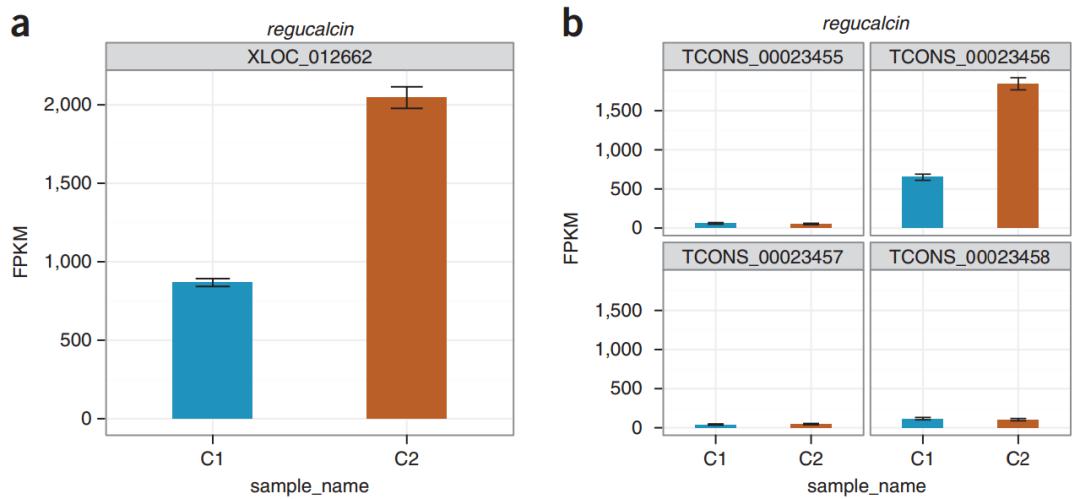
图14. 特定基因 regucalcin 的可视化展示
这个实际上是一个很整体很笼统的分析,具体做的时候实际上存在很多的挑战。相对于基因总体的表达量来说,分析它的可变剪接的难度是相对较高的。以前测的序列比较短,现在虽然测的比较长,但总体上来讲仍然不是那么长,比如说能够覆盖两个 exon,但覆盖不了整个转录本,你可能只能发现这个内含子和这个外显子包括与否,但如果说这边有一个外显子跳跃,后面还有一个外显子跳跃,你无法判断这两个外显子的跳跃是不是在一个转录本上,短的序列仍然不能回答。所以以后在读长上要有突破,包括加强测序质量,测序的时候末端序列不是那么准的,但我们去看 junction reads,通常把它一劈为二,这边多一点,这边少一点,mapping 回去;有的地方测序质量比较差的话,mapping 回去也会差,mapping 出来的基因可能也是错的,这是我们需要注意的。
另一个我们需要注意的问题是读长分布的不均匀。测到的 reads 分布在转录本的不同区域,但你去看各种不同的 track 图时,有的地方特别高,有的地方特别低,原因多种多样。比如有的地方 GC 含量特别高,就不容易测出来,特别的低也不容易测出来,或者有重复序列。这样的 reads mapping 回去的时候,它不知道是这个基因的,所有我们就把它过滤掉了,因为没办法告诉你是哪个地方来源的。这个时候 junction reads 比较高的地方,你就能够判断得比较准,但有的 junction 根本没有 reads 在这个地方 cover,这时候也会对可变剪切的分析造成影响。这种基因内部分布的不均匀可能是导致 RNA-seq 分析逐渐下降的一个很重要的原因。另外,有些剪切本本身表达量就比较低,意味着 reads 中能够贡献到这个地方的量就特别的小。对可变剪切分析而言,你可能只能分析表达量相对比较高的地方,至少可以给你得出一些线索。另外,不同的分析软件得到的结果是不一样的,原因多种多样,可能只能根据技术不停的发展和认识的深入去克服这个问题。
但我认为很重要的一个标准是怎样更好地和实验结合起来,如果分析的好的话,可能 60-70% 可以验证;分析不好的话,假阳性特别高,只有 20-30% 能够验证。在开始做的时候,我的建议是你宁愿让标准设的严一点,发现的少一点,但让假阳性低一点,可验证程度高一点,可能是一个更好的策略。
刚才讲了不均一性,简单来讲可减少 PCR 的循环数,因为 PCR 循环数减少的话,这种情况稍微得到改善,但最好的是你不做任何的扩增,但除非起始的 RNA 的量特别多,一般 RNA 比较少,尤其像做少量细胞甚至单细胞 RNA-seq 的时候,你必须进行一些扩增,扩增带来的问题就是不均一性会更强,但实际上这是对你要研究这个问题的妥协,你为了研究它,你只能在目前技术不允许的前提下,你只能用一个相对将就的办法,但至少可以给你拿到一些新的发现。
另外,去开发更高效可靠的分析软件。当然说起来容易,做起来不一定。另外增加读长,如果说我们能够做到一个全转录本测序的话,实际上是一个比较好的策略。因为刚开始讲的,如果不同的剪接事件在同一个 isoform 上发生的话,存在一些短序列,实际上是没有办法解决的,这个可以用三代测序,但这里面也存在着一些问题,因为它虽然能够测全长,但是错误率比较高,价格也非常贵,而且同样的价格覆盖的转录本是比较少的,所以这个也有赖于技术的进步。
基因可变剪接的调控——
顺式作用元件和反式作用因子
接下来我们讲一下可变剪接的调控,可变剪接的调控是非常复杂的,概括一下我个人认为可以分为三点,一个是顺式作用元件,就是 DNA 的序列是什么;第二个是反式作用因子,哪些 RNA 结合蛋白在这个区域与它结合;这两者之间的组合很大程度上决定了可变剪接是如何发生的;第三是一些表观遗传因子可以通过影响顺式作用元件和反式作用因子对它整个进行调控。
我们对整个调控层面进行理解也可分为三个层面,第一个是 RNA 层面,反式作用因子和顺式作用元件怎样互作,互作的网络可能是比较复杂的。第二个是转录机器层面的调控,就是 RNA 聚合酶的快慢或者所反映出来的延伸速度实际上在很多时候决定了剪接位点。第三个是表观遗传层面,不管是染色质结构、DNA 甲基化、组蛋白修饰,甚至 RNA 本身的修饰都会发挥作用。
先来讲顺式作用元件和反式作用因子。顺式作用元件里面很重要的一个因素就是剪接位点本身的强弱决定了在这个位置是否会跳掉,如果说它更愿意与后面外显子结合的话,中间的外显子就容易被跳掉,所以涉及到位置的竞争。另外,除了序列本身,我们知道不同的 U1 和 U2 以及其他的一些 snRNP 实际上跟特定的序列结合,而这种结合的 pairing 非常重要。

图15. 顺式作用元件:剪接位点本身的强弱 [6]
这里有个概念叫 exon definition,它是怎样定义呢?如图 15 所示,如果 5’SS 结合了 U1,3’SS 结合了 U2,这个时候就告诉剪接机器这个位置是外显子,如果有这样的信息外显子是被保留的。但如果是另外一种情况,前一个外显子的 5’SS 与中间外显子的 3’SS 形成一个 cap,中间外显子的 5’SS 与下一个外显子的 3’SS 形成一个 cap,这个剪切机器会更多地识别这两个 cap,造成外显子的跳跃,所以在这个时候,顺式作用元件和反式作用因子是一个相互的作用。
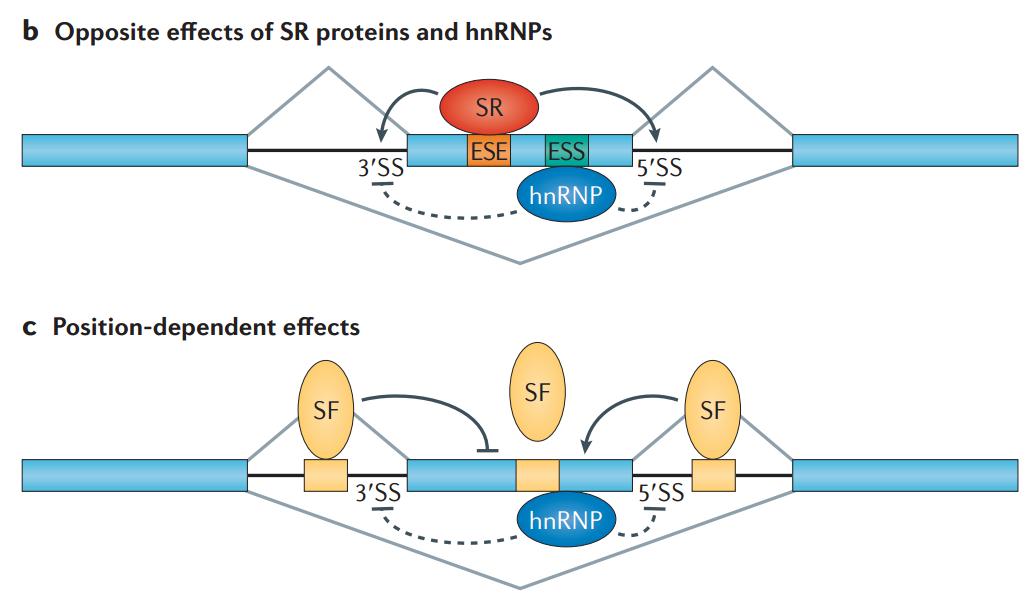
图16. 反式作用因子如何与顺式作用元件互作 [6]
总的来看,可变剪接的调控有两个特征:1.序列依赖性,2.位置依赖性。我们该如何理解呢?很多调控序列在外显子内或内含子里面,先看一下在外显子内部的这些序列,包括 ESE 和 ESS,第一个 E 代表 exon,第二个代表 splicing,第三个是 enhancer 或者 silencer,即促进或抑制剪接。对不同的序列本身,有不同的 RNA 结合蛋白结合。通常来讲,两类 RNA 结合蛋白或剪接因子是比较重要的,一个是 SR 蛋白,即丝氨酸和精氨酸富集的蛋白;另外一种是 hnRNP,它们识别的位点不一样,如果 SR 结合在剪接的 enhancer 上,那么告诉这个位点是需要选择的,最终会造成图 16 中上图的跳跃;如果 hnRNP 结合在 ESS 上,那么告知这个位点不要剪接,这样造成了外显子跳跃。所以在 exon 上的序列,其实跟反式作用因子相互作用就能够决定外显子的保留与否;当然,问题没有这么那么简单,即便对同一个转录因子的话,这个时候涉及到位置效应,它结合到不同地方,也能够调控剪接方向,比如说它结合在上游的 intron 或 exon 里面的话,起到抑制作用;如果结合在下游的 intron 上面,对它产生促进作用,所以这样导致了即便有序列和剪接因子的话,不同位置的结合,效应是不一样的。我们可以简单理解,第一种情况是序列依赖性,第二种情况是位置依赖性,让调控变得异常复杂。
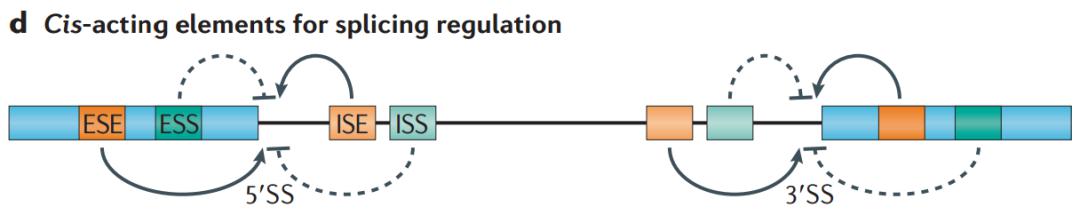
图17. 顺式作用元件对剪接位点的选择既可以正向也可以反向 [6]
刚才我们讲到了 exon 里面的结合的效应,intron 里面的结合也会有同样的效应,有的是促进,有的是抑制,即在内含子和外显子里面都有正向和负向的元件。既然存在这些元件,怎样才能更好地发现和鉴定这些 DNA 序列到底起到什么作用,这个时候涉及到用 mini 基因进行筛选。
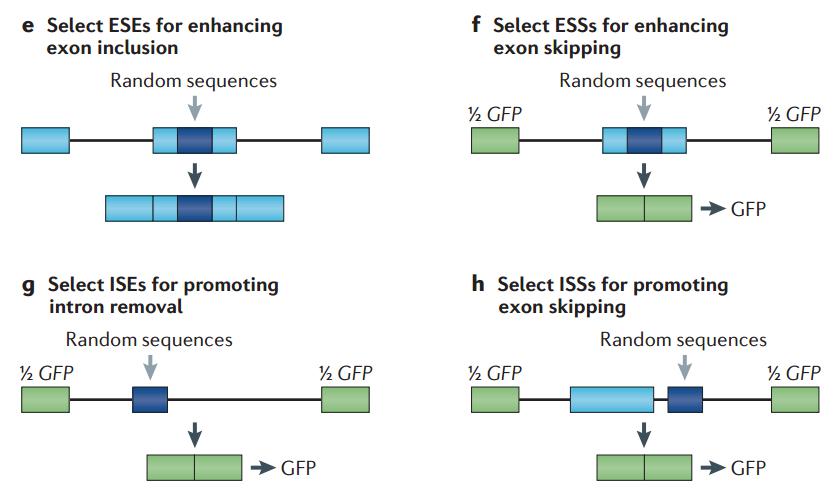
图18. 如何设计实验筛选对剪接有促进或抑制作用的顺式作用元件 [6]
比如我们想要筛选 ESE,一个简单的办法是,把这个地方做成简单的随机序列库,如果说这个序列对于外显子保留是有作用的话,就能够看到相应的 isoform,通过随机序列不停的选择,最后细胞里面出现更多的保留形式 isoform 的话,那么中间的序列更多的是 ESE。对于外显子里面的 silencer,用图 18 右上图的那个质粒来鉴定,如果外显子跳跃,两端正好拼成一个 GFP 序列,找到含有荧光的细胞,然后在细胞里面去看序列是什么,就能够找到让外显子跳跃的序列。如果要找到 intron 去除的序列,可以在图 18 左下图中蓝色区域设计一个随机的序列,如果出现 GFP,说明 intron 被很好地切割掉了,那么这个序列就是促进内含子切割的序列。另一个是 exon skipping,怎样找到下游 intron 上的一些序列,能够促进 exon 的 skipping,可以在图 18 右下图中蓝色区域设计,如果出现 GFP,证明确实出现了 exon skip。
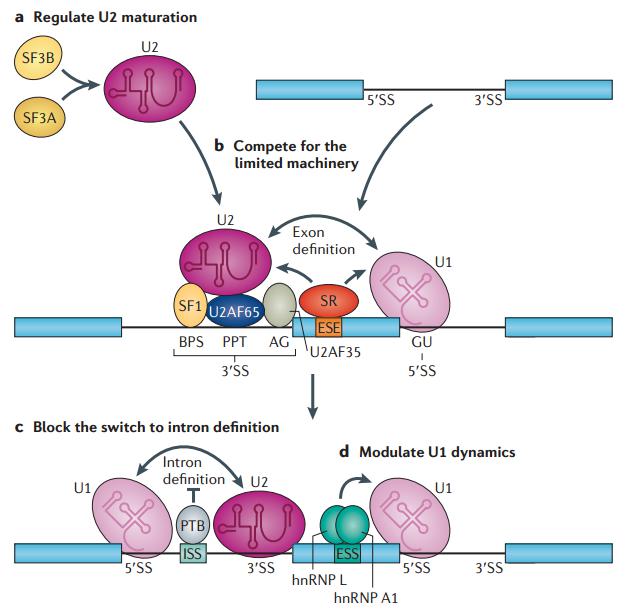
图19. 通过控制核心剪接机器的组装来调控可变剪接 [6]
除了顺式作用元件和反式作用因子,其实剪接调控和剪接体本身也有密切联系。如果 U1 和 U2 作为 tag 的话,那么中间的序列就定义为 exon。如果说有一些因子能够在那里进行竞争,那么 U1 不跟它直接结合,这个时候 exon definition 其实就发生了一些改变,中间的外显子可能被跳跃掉,这种竞争关系也是可以存在的。实际上,不光是外显子的序列,exon enhancer 和蛋白本身就能够对剪接进行促进,几个因素结合在一起发挥作用。图 19 中的 c 图,U1 和 U2 形成 pair 以后,如果有一个 ISS,实际上会造成 intron retention,这样的顺式作用元件或招募反式作用因子 PTB,就能够影响 intron definition,让它成为一个 exon,剪接机器就不再把这个 intron 给切割掉了,这是 intron retention 的一种机制。还可以对 U1 本身的占位进行调控,如果没有 U1 的话,intron definition 也就没有了。通过这样一种比较复杂的方式,决定了 intron 被切割与否,或外显子被跳跃与否。
这些所有讲的都是在 RNA 层面进行的调控,第二个层面是发生在转录机器上,具体来讲,是 RNA 聚合酶 II 在这个地方跑的快还是慢,研究者在果蝇中发现了一个 RNA Pol II 变慢的突变体,发现在 RNA Pol II 快慢两种情况下,一些基因的可变剪接是不一样的。
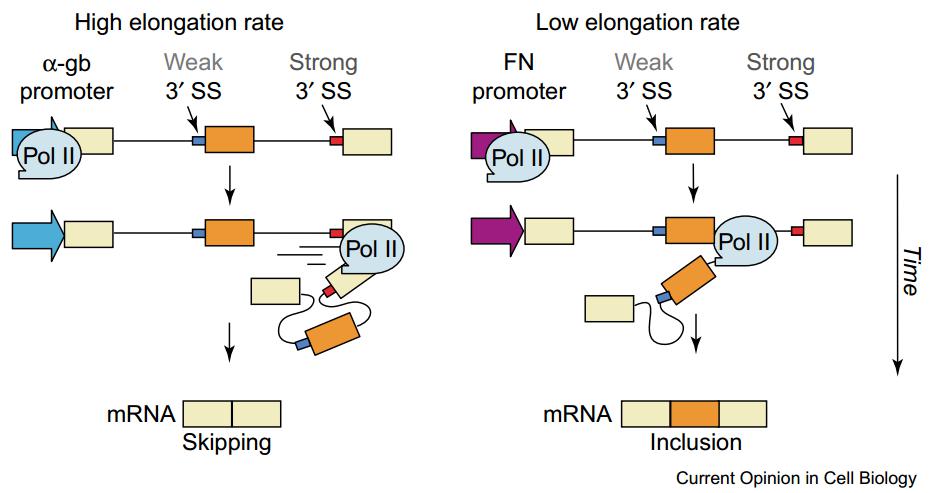
图20. RNA 聚合酶 II (Pol II) 的延伸速度可决定特定外显子的跳跃与否[7]
当 RNA 聚合酶 II 跑的很快时,中间是一个很弱的 3’SS,根本没有来得及去剪切,这个时候强的 3’SS 占了主导,原来比较弱的位点处发生了 exon skipping,这个过程中并没有其他反式作用因子来参与。如果转录的时候相对比较慢,这个时候剪切机器有足够的时间来识别比较弱的剪切位点,这样就可以发生正常的剪切,此时 exon 包含。在这个例子中,弱的剪切信号可以通过 RNA 聚合酶 II 跑的快慢决定包含与否,这是一个最简单直观的例子。
这个方式听起来很简单,也很合理,较慢的 Pol II 延伸速度一定导致外显子的包含吗?生物学里面总能找到例外。这个时候,可以把反式作用因子也引进来。Pol II 跑的比较慢的时候,虽然可能发生了正常的剪接,但是 Pol II 上同时还结合了抑制性的反式作用因子的话,可以发生外显子跳跃。你会发现,后面的很多染色质跟它的调控,无非都是通过这种套路来解释,有了方向以后,再去找,到底是哪个反式作用因子,是不是真的能解释这个现象?从原理上,不难提出假说。
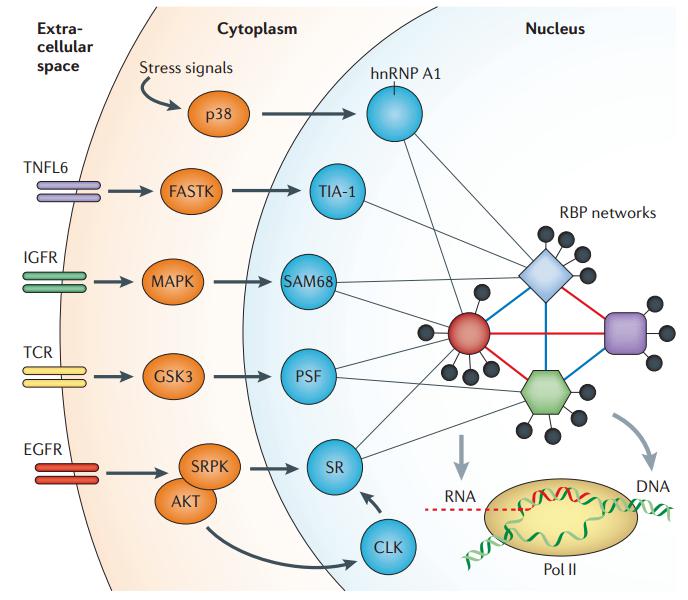
图21. 通过特定信号通路来调控细胞核内的可变剪接 [6]
我们整个组织细胞是一个动态的过程,剪接如何发生以及被调控,可以通过细胞外的信号传递进来,这些通路发生改变后,通过级联反应,可改变特定蛋白的磷酸化和其他修饰,而剪接因子也可以发生很多修饰,这些修饰发生改变,也可以调控细胞核中的剪接事件发生的多少,通过这种方式,就可以将刚才讲的剪接方式与外界的因素联系在一起,在生理或病理情况下,可能就是通过这种形式来体现的。
组蛋白修饰与基因可变剪接
既然我们已经知道了比较基础的调控方式,怎么样解释组蛋白和基因可变剪接的关系?在转录过程中,之前我们的模式图显示,RNA 离开染色体后单独加工;如果我们去看实际情况,转录出来的 RNA 和各种组蛋白修饰、RNA 聚合酶 II 还是连成了一个整体,在它还没有从 DNA/染色质中下来的时候,物理空间是相邻的,而这样一种相邻为两者之间的相互关系提供了一种可能性——RNA 上可以结合一些组蛋白修饰的酶,组蛋白修饰的酶可以对特定的位点加上修饰;反过来,这些修饰可以通过其他方式结合一些剪接因子,有可能作用在 RNA 上,这样就存在剪接调控。当然,具体是哪些分子参与,哪些组蛋白修饰发挥作用,还需要具体鉴定。
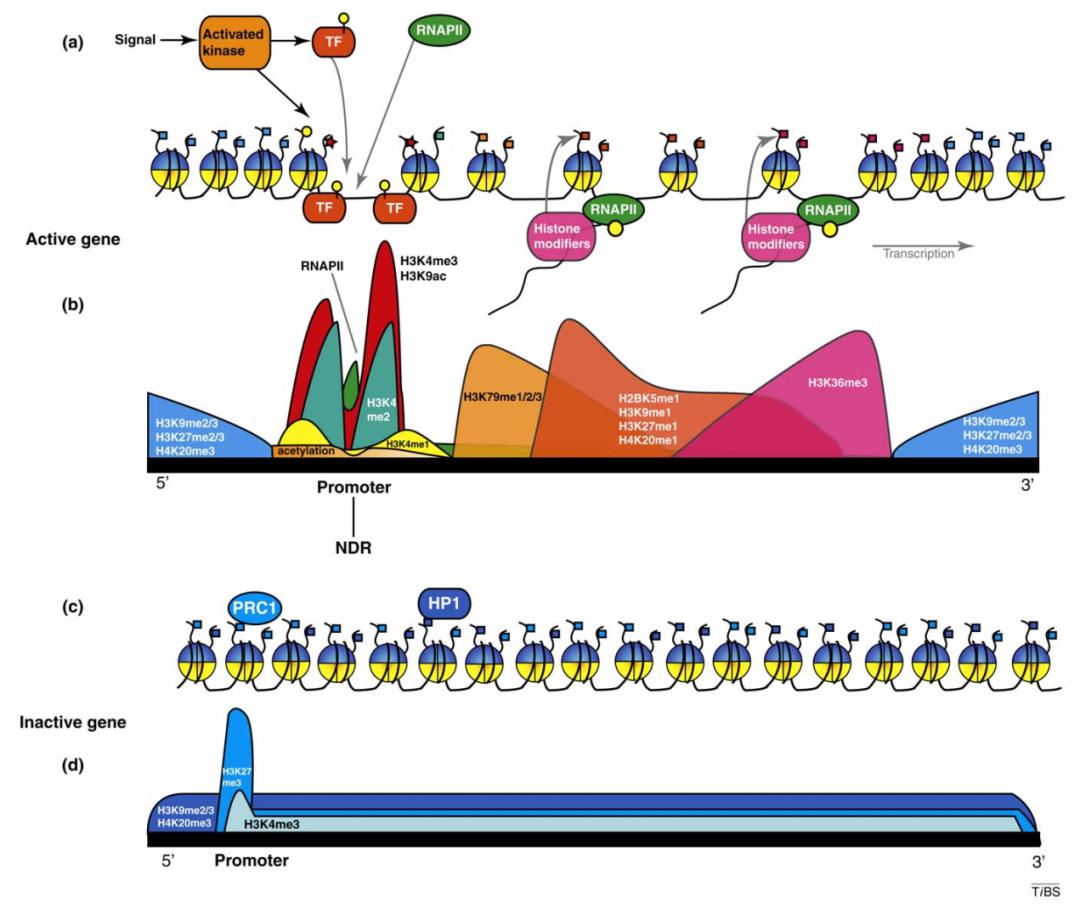
图22. 转录过程中 pre-mRNA 与组蛋白修饰间有物理上的相邻 [8]
我们从不同组蛋白修饰在基因区域上的分布来看,很多 marker 在启动子区域,标记基因是否活跃;在活跃基因的内部,还有各种不同的修饰,中间的一些组蛋白修饰与可变剪接关系可能比较紧密。从位置上,我们可以猜测,如果发生修饰的话,中间的修饰对剪接的影响可能更大。
举个例子,染色体中含有一个比较弱的剪接位点,中间有不同的核小体分布,以及组蛋白修饰,这些可以影响 RNA 聚合酶 II 的延伸速度。在比较弱的剪接位点,延伸速度比较快的话,就发生了跳跃;延伸速度比较慢的话,外显子就能保留。所以,下半部分的机制,完全是借用了 RNA 聚合酶 II 的延伸速度的快慢,唯一需要证明的是,特定组蛋白修饰和核小体分布是否会影响 RNA 聚合酶的快慢。很多机制上的研究,只要把重要的一环证明,这样一种机制就可能成立。
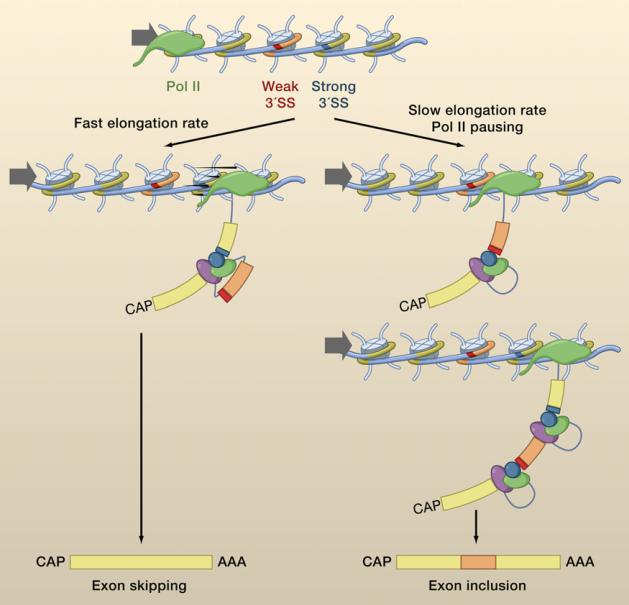
图23. 核小体密度决定 RNA Pol II 延伸速度,Pol II 速度决定外显子跳跃 [9]
另外一种调控方式,特定组蛋白修饰(比如 H3K36me3)的 reader,进一步可以结合剪接因子,可以促进也可以是抑制。如果修饰不存在,或者换成其他修饰了,即便你有splicing factor,这种组蛋白修饰仍然不能对可变剪接进行调控,这样就造成了外显子保留。要么在特定位点招募了一些特定蛋白,这个蛋白只要跟剪接调控因子结合在一起,就能够有效地调控剪接。当然,有个前提是,里面有个位点是比较弱的,因为如果比较强的话,可能根本就不需要调控,或者也没办法调控。
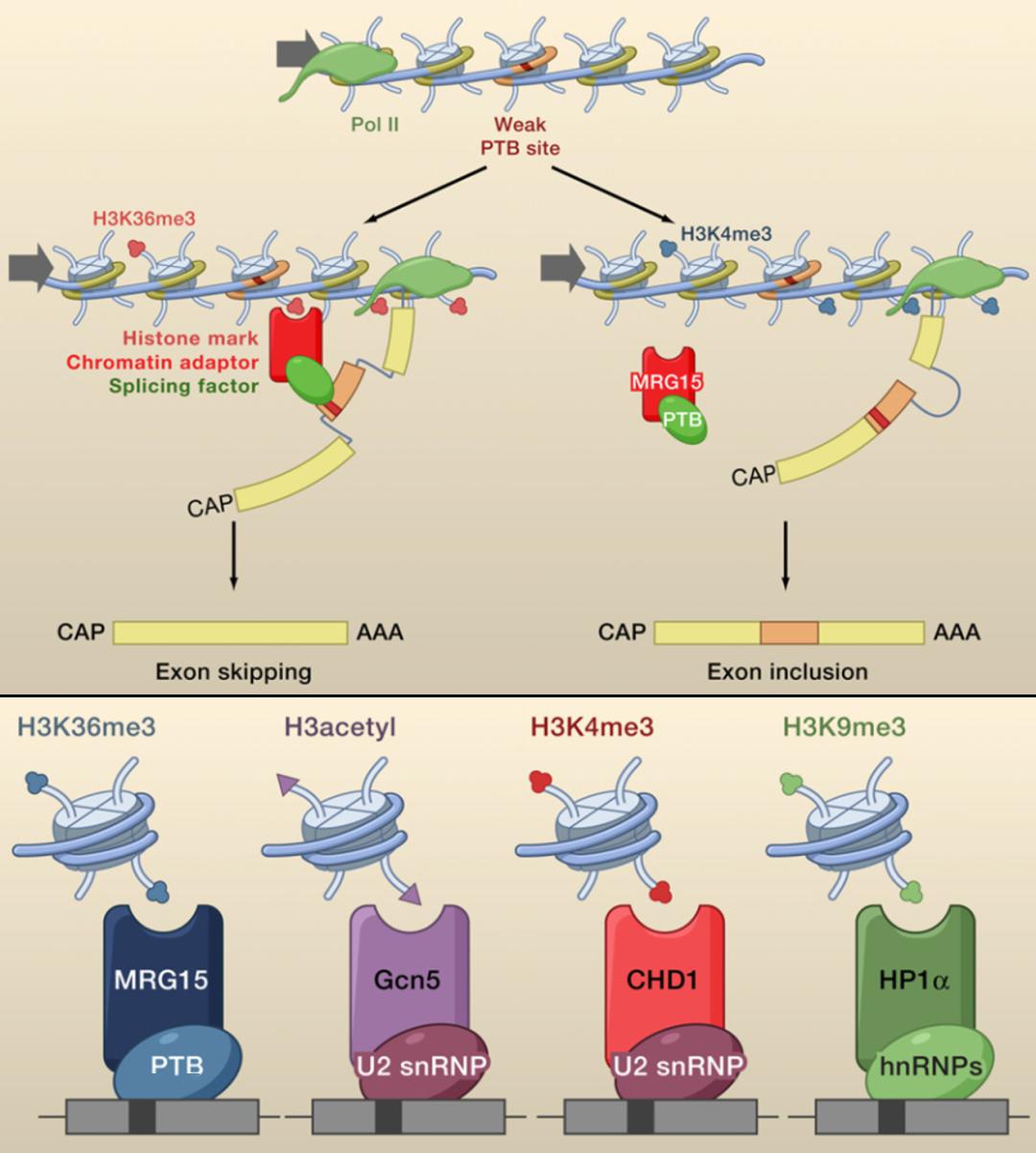
图24. 组蛋白修饰通过招募特定 reader 进而结合剪接因子从而影响可变剪接 [9]
具体来讲,文献中还报道了其他组蛋白修饰的 reader,比如 H3ac、H3K4me3、H3K9me3,也会招募其他剪接因子,在特定的位点调控可变剪接。如何发现组蛋白修饰对可变剪接的影响?从转录组层面,如果你关心特定的组蛋白修饰,你可以看它改变前后一些组蛋白修饰酶的改变,可能会影响组蛋白修饰的改变,然后进行 ChIP-seq,就知道组蛋白修饰分布的情况;再做 RNA-seq,看一下 splicing 的改变。看两者之间的关联。进行一些分析和验证。在单基因水平上验证,采用 RT-PCR、Northern Blot、ChIP-PCR 等等。
这里我们举一个例子,是蓝斐老师他们发现的 H3.3K36me3,通过 BS69 并招募剪接因子复合物,调控特定基因的 intron retention。在正常细胞和 BS69 敲低细胞中做 RNA-seq,进行生信分析,发现 BS69 影响内含子保留。
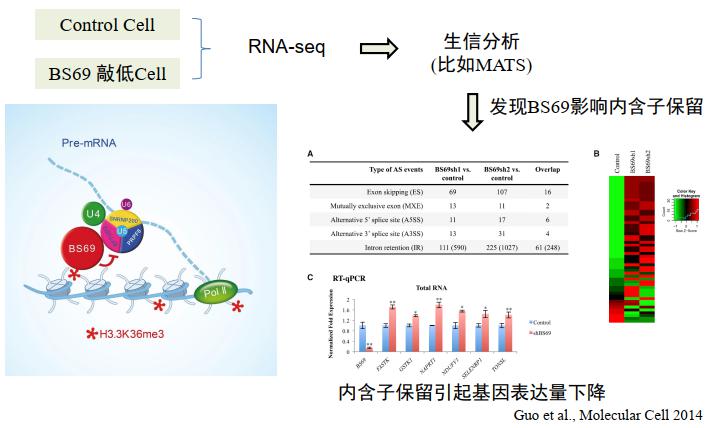
图25. 转录组水平系统发现 [10]
为什么呢?植物中的 intron retention 是比较多的,而动物中一个因素改变后,更多的是改变其他的可变剪接类型,比如 exon skipping,但他们发现的 intron retention 是改变最显著的,这样就可以建立 BS69 与 intron retention 的联系。而且发现基因表达也出现改变,跟我们的结果也是比较吻合的,而且他们也在单基因水平上进行了验证。
通过这个例子,回答了中间一个环节,组蛋白修饰至少可以通过两种方式影响 RNA 剪接,那么反过来是不是成立的?RNA 剪接会不会影响组蛋白修饰呢?
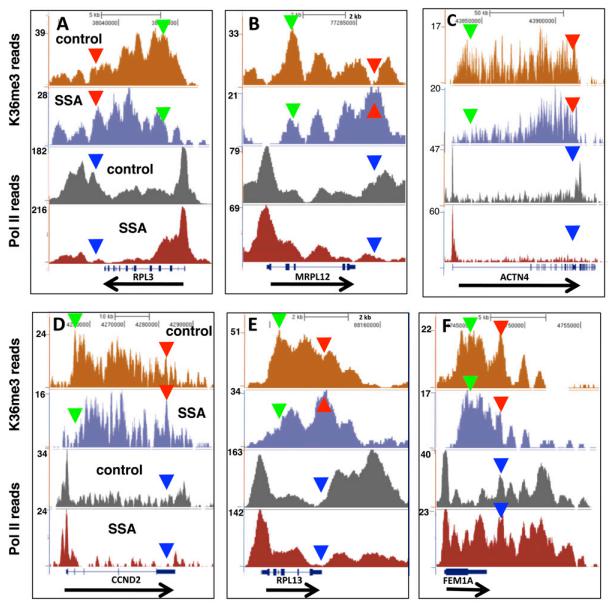
图 26. 剪接抑制剂可显著改变 H3K36me3 和 Pol II 分布 [11]
如果要验证可变剪接改变对组蛋白修饰的影响,首先要改变可变剪接,研究者用了化学药物,抑制可变剪接的效率,后来进一步证明,这样一种药物模拟了剪接位点的突变,这样就改变了可变剪接,然后做 ChIP-seq,发现 H3K36me3 分布显著改变,而且也发现 RNA 聚合酶 II 的分布也发生了与 H3K36me3 类似的改变,这暗示剪接的改变很可能影响组蛋白修饰,而且很可能与 RNA 聚合酶 II 位置上的变动有关。进一步的机制还不是很清楚,只是提供了中间的线索。但至少证明了一点,可变剪接可能影响组蛋白修饰。
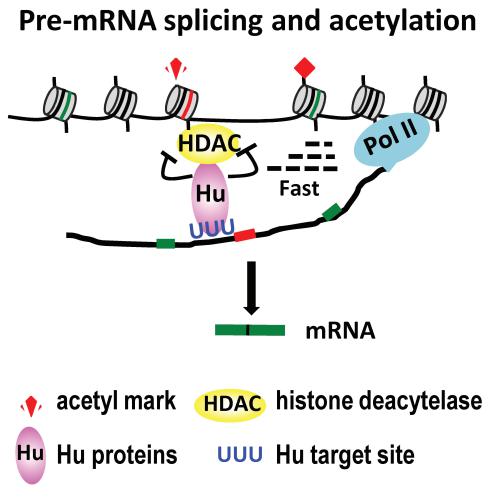
图27. RNA 结合蛋白可招募组蛋白去乙酰化影响组蛋白修饰 [12]
在植物中也进一步证明了这一点,RNA 结合蛋白会结合到不同的序列上,Hu 蛋白会招募 HDAC,由于这两者物理位置上的接近,会对邻近的组蛋白的乙酰基去掉,通过这种方式对组蛋白修饰产生影响。
现在的研究还不是很多,目前的例子也就这些。但已经说明了一点,组蛋白修饰可能影响剪接,剪接也会影响组蛋白修饰。在有机体内,这两者可能会进一步的相互作用,甚至反馈调控,可能是正反馈也可能是负反馈,从而使机体达到一种稳态。生物体之间存在各种各样的联系,关键是找到证明其存在的证据以及其背后的逻辑。
DNA 甲基化与基因可变剪接
既然 DNA 甲基化修饰与可变剪接相关,一个简单的办法就是对全基因组 DNA 甲基化进行分析。怎么分析呢?分组,分为外显子和内含子,区别外显子和内含子数据哪些是一样的,哪些是不一样的。有的 GC 含量一样,有的不一样。为了避免 GC 含量因素对结果的影响,他们做了一个设定,只分析外显子和内含子内 GC 含量一样的组别,这样得出来的结论消除 GC 含量的影响,结果发现外显子上有更高的 DNA 甲基化。
这个比较有意思,要看两者之间是否有相互作用,先看有无相关性,有相关性并不代表有因果关系。但通常如果有因果关系,相关性方面应当有些线索;如果什么都没有,这个事情就变得更困难。进一步分析发现,剪接位点附近的甲基化程度相对于内含子里边其他的更远的地方,水平明显不一样。这为我们提供了一个很好的线索,如果甲基化会影响 splicing 的话,它在这两个不同位置的甲基化程度给它的作用提供了一个基础。进一步再想,如果 DNA 甲基化可以影响选择性剪接,那么通过什么样的方式?
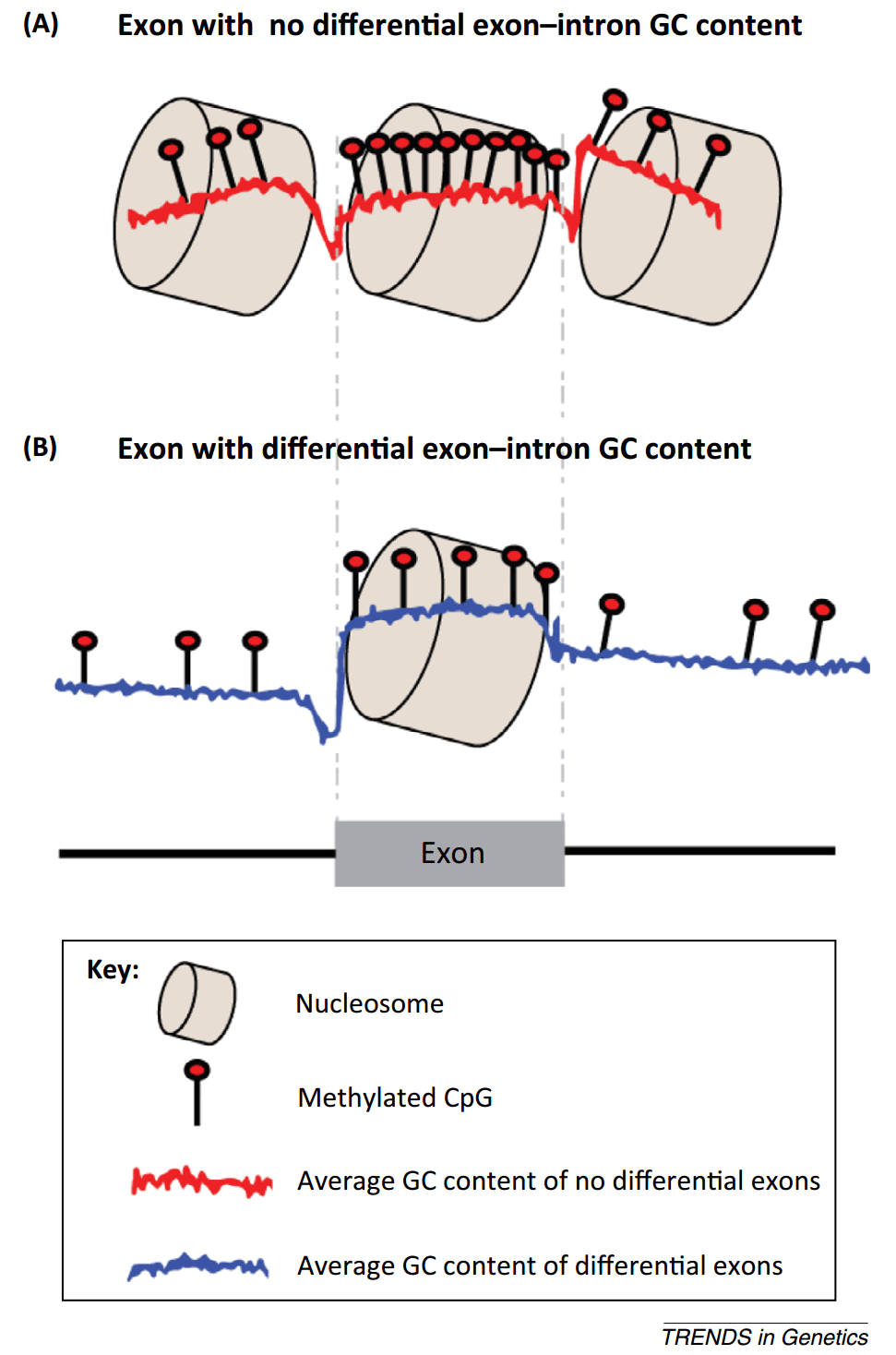
图28. 两种不同的内含子-外显子结构 [13]
第一种方式,DNA 甲基化可以间接地通过影响组蛋白的修饰从而影响剪接。这在理论上是成立的,但实际能不能找到这样的例子还未知。另外一个办法,我们可以去关注 DNA 甲基化的结合蛋白。大家知道,甲基化有专门的结合蛋白,类似于组蛋白修饰中的 reader,你也可以认为这个结合蛋白是 DNA 甲基化的 reader。但与组蛋白修饰有点不一样,DNA 甲基化之后,某些蛋白对 DNA 的结合能力不同。有些蛋白质不喜欢甲基化,因此 DNA 甲基化之后蛋白质不能结合,有的必须要 DNA 甲基化之后蛋白才结合。那么一旦蛋白结合在这个地方,这些蛋白有没有可能通过下游的因素影响剪接呢?这也许是个可能。
很多研究发现了介导这一过程的因素,是一些大家耳熟能详的因子,比如 CTCF,它与 DNA 甲基化的结合与它的一个 domain 有关系。但是大家发现,这个因子居然同样可以影响它的剪接;另一个是 MeCP2,它是一个甲基化结合蛋白。这两个因子就是刚才讲的,一个通过结合在非甲基化序列上影响剪接,一个通过结合在甲基化序列上影响剪接。还有一个是 HP1,它是组蛋白 H3K9 甲基化 reader。DNA 甲基化修饰影响剪接主要通过三种机制来实现的:一是改变速率,一旦改变速率,后面的机制就容易解释;二是招募剪接因子;三是通过 Pol II 的快慢来实现,基本是由这三种机制来实现。
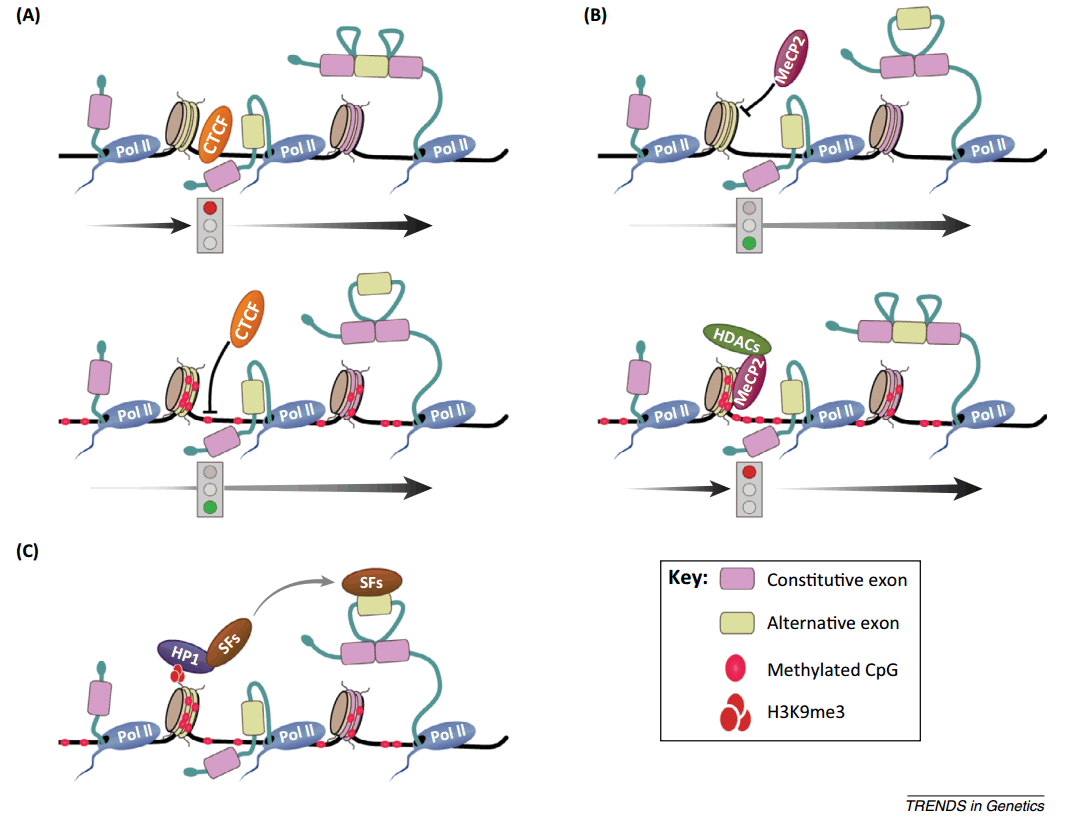
图29. DNA 甲基化通过三种机制调控可变剪接 [13]
第一个例子中的 CTCF,它的结合具有序列特异性,CTCF 与特定 DNA 序列结合后,会让 RNA 聚合酶 II 变慢。RNA 聚合酶 II 变慢之后,弱的剪接性号也能作为外显子,因此外显子包含,这个时候形成一个三个外显子转录本。如果这个地方发生甲基化,那么 CTCF就没有办法结合在这个 DNA 序列上。如果 RNA 聚合酶 II 跑的快,由于这个位点比较弱被剪接掉,实际上会造成外显子跳跃。从原理上来讲,这种作用方式解释的相对比较清楚,但却没能解释在什么情况下这个位置的 RNA 聚合酶跑的快或慢。
那么作为一个 DNA 甲基化结合蛋白,MeCP2 如何影响 splicing?正好相反。DNA 发生甲基化,会促进 MeCP2 结合上去,而且 MeCP2 还会招募一些特定的组蛋白修饰酶,如 HDAC,在下游的因子的作用下,它会让 RNA 聚合酶 II 变慢。如何变慢,没有解释清楚,至少给出了一个线索,就是 MeCP2 结合上去会让 RNA 聚合酶 II 会变得慢,变慢之后会造成外显子正常的切割包含,如果 DNA 没有甲基化,MeCP2 不会与之结合,RNA 聚合酶 II 快速移动,造成外显子跳跃。这两个例子都是蛋白结合在这里造成 RNA 聚合酶转录或延伸速率,决定外显子跳跃与否。
第三个例子是通过组蛋白修饰影响剪接。可以看到,如果 DNA 发生甲基化,会进一步使组蛋白上发生 H3K9me3,这种修饰会招募 HP1。HP1 是一个识别 H3K9me3 的蛋白,有了这个蛋白之后,它会再去招募 splicing factor。如果这个 splicing factor 是促进 exon skipping 的,在这个过程中,两个 splicing factor 的位置比较近,促使外显子发生跳跃。这个假设走了一条很长的路,很奇怪,从 DNA 修饰到组蛋白修饰,再到 reader,然后再 splicing factor,解释起来比较麻烦。这个机制给大家的研究造成很大的困难,如果你要证明这个方式确实存在,首先要证明是 H3K9 甲基化而不是其他的,接下来解释转到 splicing factor,到底结合哪个 splicing factor,这个还得去找,这个给研究带来了更大的难度。
刚刚讲了这三种机制,大家觉得从原理上讲哪种更普遍,哪种更特异性?从原理上来讲,CTCF 识别特定序列,只有某些特定基因才有这样的序列,这样它可能特异性更强;对于 MeCP2,只要是基因发生甲基化,它就能与之结合,对基因的序列要求没有那么高,从而调控更多基因的剪接;HP1 与它的作用方式类似,因为它可以识别 H3K9me3,H3K9me3 在很多核小体上都能存在,因此它对基因选择性没有那么强。初步的猜想就是 CTCF 调控的基因更少,后两者更多一点。
好像一切看上去都很完美,很好地解释了 DNA 甲基化确实会影响 splicing,但你仔细看一下我们之前说的关联研究和后面的机制研究,存在一个让人想不通的事情。你看一下被跳掉的 alternative exons,它含有的 DNA 甲基化水平实际上比其他外显子的甲基化水平低。但之前我们说外显子上的 DNA 甲基化比较高,我们之前认为外显子高的甲基化水平跟它的剪接是相关的,DNA 甲基化是 promote 外显子的定义。但实际上我们去看里面的数据,去推广这个假说,其实是不成立的。因为在特定的胚胎干细胞中看到的现象是 DNA 甲基化可以促进或抑制特定 alternative exons inclusion [14]。原来认为简单的 DNA 甲基化决定外显子,实际上是有的时候甲基化高的区域作为外显子保留,有的时候又作为内含子被切掉,这与假说中的情况不完全一样。
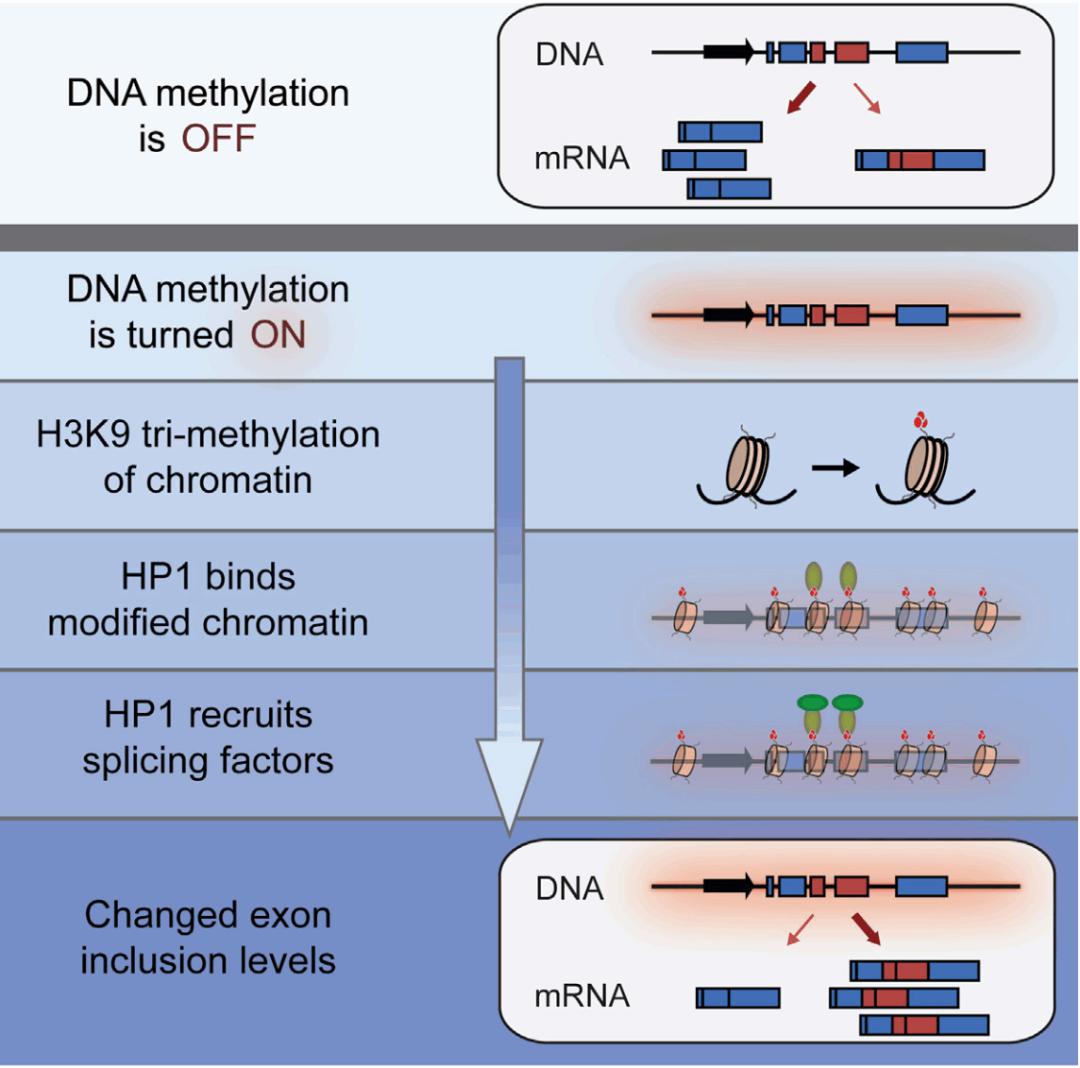
图30. DNA 甲基化可促进或抑制特定 alternative exons inclusion [14]
除了我们讲的 alternative exon,其实还存在一种组成型外显子 constitute exon。我们看到的甲基化水平高可能只是在组成型外显子显示的甲基化水平高,但是如果将组成型和可变的外显子放在一起比较,与原来的假设相矛盾了。实际上对于 constitute exon,它更多的是由这个强的剪接信号来决定,这时甲基化水平高低只是一种伴随现象,不管甲基化水平高或低,旁边的 intron 都会被剪掉。alternative exon 一直都被认为是一个外显子,甲基化水平可能对这个可变外显子的影响更大,这样可以更好解释为什么全基因组水平和单基因水平机制验证有可能存在矛盾。
以上这些分析可以得出一个初步的结论,DNA 甲基化通过多种方式影响 RNA 剪接,那么反过来会不会成立,根据前面一个猜想肯定也是成立,遗憾的是并没有这方面证据。
染色质修饰与基因转录终止
最后稍微再讲一点,RNA 加工不仅是一个可变剪接的过程,还有加 Poly(A) 尾。Poly(A) 尾在哪个地方加也是非常重要的,很多剪接因子影响加 Poly(A) 尾,两者关系密切。表观遗传修饰与转录终止之间有什么关系?转录终止提示在哪个位置加 Poly(A) 尾巴。如图 31 所示,gene body 有不同修饰,尤其是在 3’-端存在很多组蛋白修饰。那这种特异修饰有没有可能与 3’-端的 Poly(A) 尾存在联系,答案是肯定的。
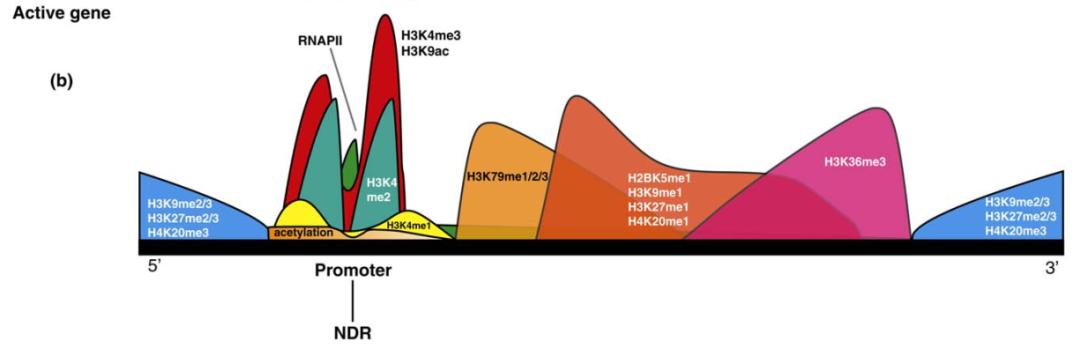
图31. 基因组上不同区域的组蛋白修饰分布
我们先简要了解一下 alternative polyadenylation,之前讲过如果在 3’-UTR 区选择不一样 3’-UTR 长度,对蛋白质的量会造成影响,但是 alternative polyadenylation (APA) 也可以影响蛋白质的质。因为如果 APA 在 intron 区域,会使开发阅读框后面发生改变,造成蛋白质 C-端改变,功能可能不一样。这两种方式在细胞内广泛存在并发挥作用,具体作用非常多。70% 以上基因都存在 APA 现象,它参与很多过程,像肿瘤、细胞分化、发育、组织特异性,而且更有意思的是在细胞增殖过程中,增殖比较快的细胞,癌细胞或癌旁,更多基因会选择近端 APA 位点,让基因表达量产生明显的改变,提示这种现象有非常重要的生物学意义,在这个过程中研究如何被表观遗传调控很有意义。
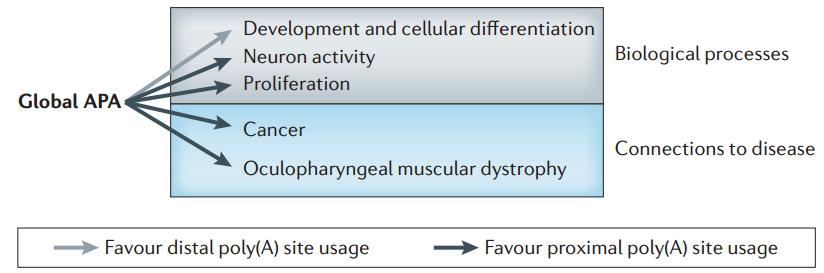
图32. APA 相关的生物学过程 [15]
通过组学手段发现很多细胞里面,如果说一个基因有两个 Poly(A) 位点,如果去看这两个位点附近各种组蛋白修饰,明显表现出有很多不同特征,如下图 33,组蛋白修饰在上游 APA 位点和下游 APA 位点分布很不一样,H3K36me3 在近端和远端 Poly(A) 位点也不一样,那么这样一种相关性其实提示组蛋白修饰有可能调控 Poly(A) 位点的选择。
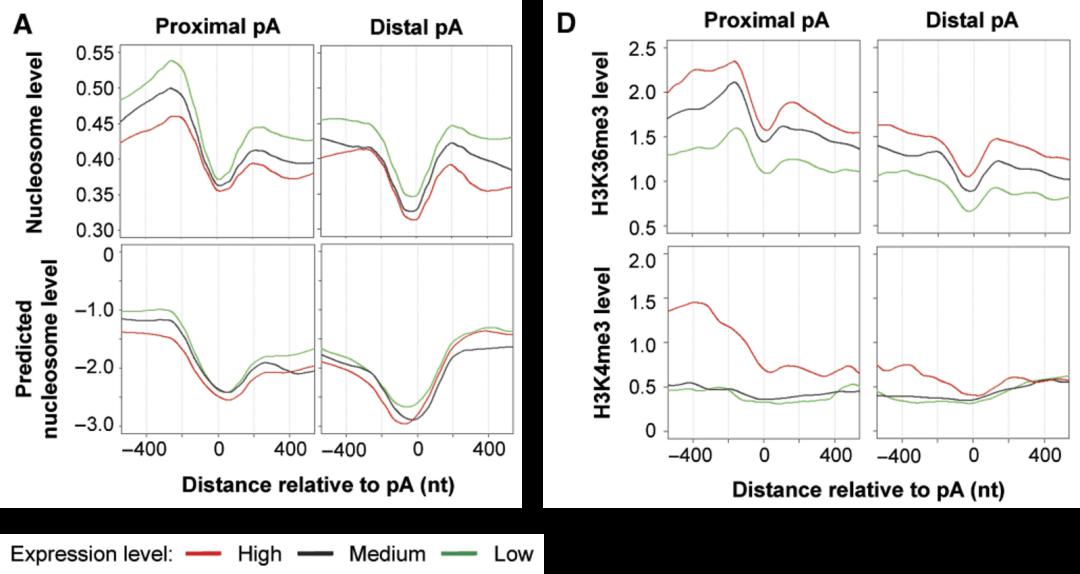
图33. 不同 APA 位点的组蛋白修饰 [16]
在植物里面也发现了类似的现象,组蛋白修饰确实影响 Poly(A) 位点选择。比如有两个转录本,选择近端或者说上游端 Poly(A) 位点,这个是常规的,在这个附近有很多修饰,既有 DNA 甲基化,又有组蛋白修饰,组蛋白修饰有很多不同的 reader,这些 reader 最后调控加尾因素的选择,最后抑制或促进 Poly(A) 位点选择,这样组蛋白修饰或表观遗传标记对选择哪个地方结尾有了因果关系。
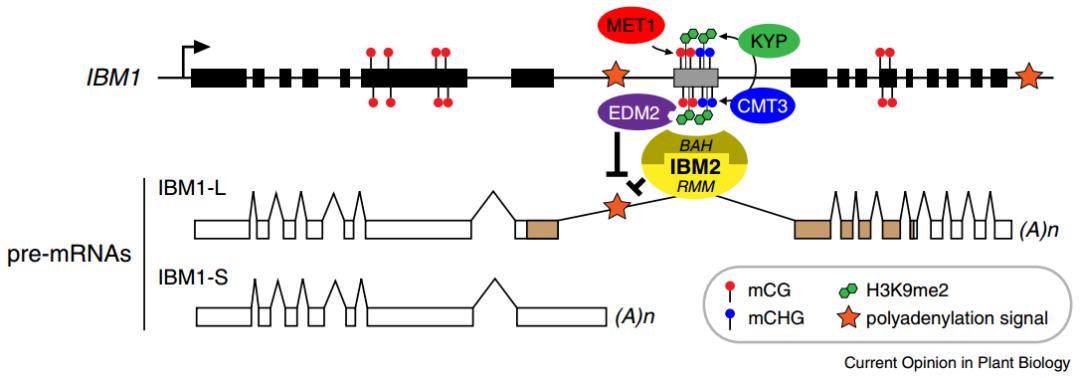
图34. 组蛋白修饰影响 Poly(A) 尾的选择 [17]
总结一下,DNA 甲基化、组蛋白修饰、可变剪接之间可以相互影响。影响可变剪接的因素包括顺式作用元件本身强弱,和它的反式因子结合或剪接因子浓度、以及是不是可以影响 RNA 聚合酶 II 的延伸快慢。同时组蛋白修饰能够影响 RNA 末端形成,剪接因子不仅影响 RNA 剪接,还影响 RNA 3’-端的形成。但有一点仍不清楚,RNA 剪接发生之后会不会对 DNA 甲基化造成影响。除此之外,RNA 修饰会不会对 RNA 剪接造成影响,杨运桂老师也讲了,特定 RNA 修饰可以通过改变特定蛋白,最后对 RNA 剪接造成一些影响,而这样一些影响可以进一步来推,比如说 RNA 修饰影响 splicing 之后,会不会再影响组蛋白修饰,再进一步影响甲基化,我觉得这也是完全有可能的。
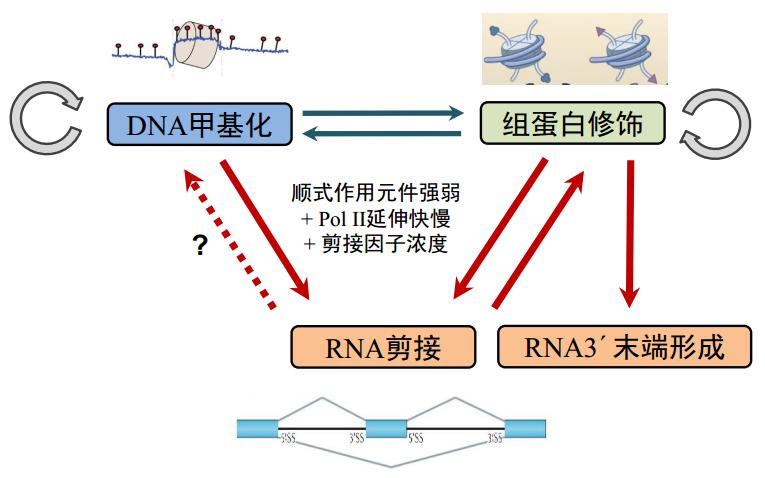
图35. DNA 甲基化、组蛋白修饰、RNA 剪接和 Poly(A) 尾形成的关系
这样在中心法则下面 RNA 作为一个中间传递者,在它的水平上发生一个很多的调控,这些调控不仅对下面的蛋白质造成影响,可能反过来影响上游的遗传物质,起到一个很重要的承上启下的作用,甚至是一个很中心的位置。RNA 加工过程中存在很多未知因素,从不同角度去看,会有很多新的发现,如果能够把这些新的发现的一个上游因素阐述的相对比较清楚,对过程理解会更深刻。
整理:复旦大学生物医学研究院徐鹏、重庆医科大学蔡莹、中科院上海药物所徐晓伟
责编:复旦大学生物医学研究院徐鹏
参考文献:
1. Wang, Y., et al., The splicing factor RBM4 controls apoptosis, proliferation, and migration to suppress tumor progression. Cancer Cell,2014. 26(3): p. 374-389.
2. Di Giammartino,D.C., K. Nishida, and J.L. Manley, Mechanisms and consequences of alternative polyadenylation. Mol Cell, 2011. 43(6): p. 853-66.
3. Mayr, C. andD.P. Bartel, Widespread shortening of 3'UTRs by alternative cleavage and polyadenylation activates oncogenes in cancer cells. Cell, 2009. 138(4):p. 673-84.
4. Mortazavi, A., et al., Mapping and quantifying mammalian tranomes by RNA-Seq. Nat Methods, 2008. 5(7): p. 621-8.
5. Trapnell, C., et al., Differential gene and tran expression analysis of RNA-seq experiments with TopHat and Cufflinks. NatProtoc, 2012. 7(3): p. 562-78.
6. Fu, X.-D. and M.Ares Jr, Context-dependent control of alternative splicing by RNA-binding proteins. Nature Reviews Genetics,2014. 15: p. 689.
7. Kornblihtt, A.R., Promoter usage and alternative splicing. Current opinion in cell biology, 2005. 17(3): p. 262-268.
8. Barth, T.K. and A. Imhof, Fast signals and slow marks: the dynamics of histone modifications. Trends Biochem Sci, 2010. 35(11): p. 618-26.
9. Luco, R.F., et al., Epigenetics in alternative pre-mRNA splicing. Cell, 2011. 144(1): p.16-26.
10. Guo, R., et al., BS69/ZMYND11 reads and connects histone H3.3lysine 36 trimethylation-decorated chromatin to regulated pre-mRNA processing. Mol Cell, 2014. 56(2): p. 298-310.
11. Kim, S., et al., Pre-mRNA splicing is a determinant of histone H3K36 methylation. Proc Natl Acad Sci U S A, 2011. 108(33): p. 13564-9.
12. Zhou, H.L., et al., Regulation of alternative splicing by local histone modifications: potential roles for RNA-guided mechanisms. Nucleic Acids Res, 2014. 42(2): p.701-13.
13. Lev Maor, G., A.Yearim, and G. Ast, The alternative role of DNA methylation in splicing regulation. Trends Genet, 2015. 31(5): p. 274-80.
14. Yearim, A., et al., HP1 is involved in regulating the global impact of DNA methylation on alternative splicing. Cell Rep, 2015. 10(7): p. 1122-34.
15. Elkon, R., A.P.Ugalde, and R. Agami, Alternative cleavage and polyadenylation: extent, regulation and function. Nat Rev Genet, 2013. 14(7): p. 496-506.
16. Ji, Z., et al., Tranional activity regulates alternative cleavage and polyadenylation. Mol Syst Biol, 2011. 7: p. 534.
17. Mathieu, O. andN. Bouche, Interplay between chromatin and RNA processing. Curr Opin Plant Biol, 2014. 18: p. 60-5.
- 本文固定链接: https://maimengkong.com/learn/797.html
- 转载请注明: : 萌小白 2021年11月6日 于 卖萌控的博客 发表
- 百度已收录
