什么是泛基因组?
泛基因组(pan-genome)作为一个概念出现的时间并不长,相应的研究文献也并不多。但它作为基因组学研究积累到一定阶段而出现,在更大尺度上进行的研究,甫一面世便受到了研究者广泛的关注。
泛基因组(pan-genome)指一个群体中所有基因组的总和,其中前缀pan来源于希腊语“παν”(指全部)。泛基因组通过对一个物种中多个个体或是一个分类群体中多个物种的基因组信息进行分析,以进行功能、进化方面的研究。泛基因组包含一个群体中所有个体均共享的核心基因信息,也包含部分个体所特有的非必须基因信息。
目前泛基因组研究已经在水稻、小麦、鸟类等物种中展开,而最近发表于Nature Genetics上的论文The tomato pan-genome uncovers new genes and a rare allele regulating fruit flavor阐述了番茄的泛基因组研究结果,将番茄这种重要作物的研究提高到了一个新的高度,论文的通讯作者是美国康奈尔大学的James J. Giovannoni教授和费章君教授。

研究背景
番茄是世界上最广泛种植和食用的蔬菜之一,2017年全球年产量1.82亿吨,价值600亿美元。番茄原产于南美洲西部和中美洲(厄瓜多尔,秘鲁等地),16世纪由西班牙人带回欧洲,并逐渐传入世界各地。
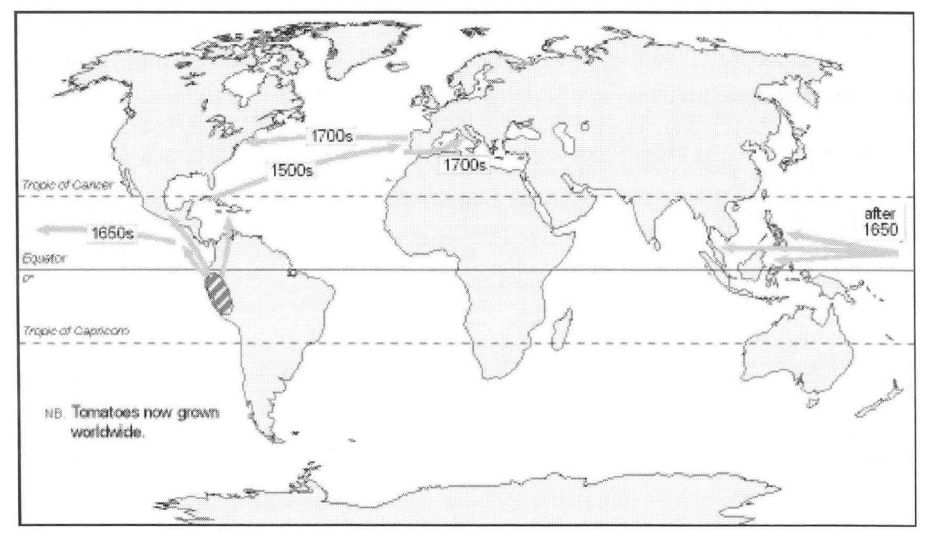
图1 番茄进化路线图
番茄在种植驯化过程中经历过严重的瓶颈事件,所以导致了现在的栽培番茄品种基因多样性较低。但是番茄在经历了多年栽培驯化的过程之后,仍然在不同品系之间显示出表型和代谢等方面的巨大差异。各个品系可能都含有各自独特的序列和基因,并且在表型上有所体现。

图2 不同表型的番茄
泛基因组构建
构建番茄泛基因组使用了以下4类番茄的序列:大果型栽培番茄(S. lycopersicum var. lycopersicum, SLL)372个品种,小果型栽培番茄(S. lycopersicum var. cerasiforme, SLC)267个品种,栽培番茄的近缘物种(S. pimpinellifolium, SP)78个品种,栽培番茄的近缘物种(S. cheesmaniae and S. galapagense, SCG)8个品种。其中98个品种测序超过20×,242个品种超过10×。对每个品种的序列进行了初步组装,得到306Gb长度超过500bp的contig,与参考基因组(Heinz 1706)进行比对后,得到4.87Gb与参考基因组相似程度低于90%的序列。去冗余后得到351Mb不存在于参考基因组中的序列。
对构建得到的番茄泛基因组进行了基因注释和分析,并和参考基因组进行比对,找出了不存在于参考基因组中的4873个基因。但是部分基因可以被参考基因组的reads比对上,可能是在Heinz 1706基因组中包含但未被组装出来。同时,对参考基因组的分析发现,参考基因组中含有35768个基因,但其中272个被判断为可能的组装错误或污染基因而移除。综合来看,整个番茄泛基因组共包含40396个基因,其中74.2%是核心基因(即在所有品系中都存在)。并且分析发现,随着组成泛基因组的品种数增加,泛基因组的大小逐渐增加而核心基因的数量逐渐减少。
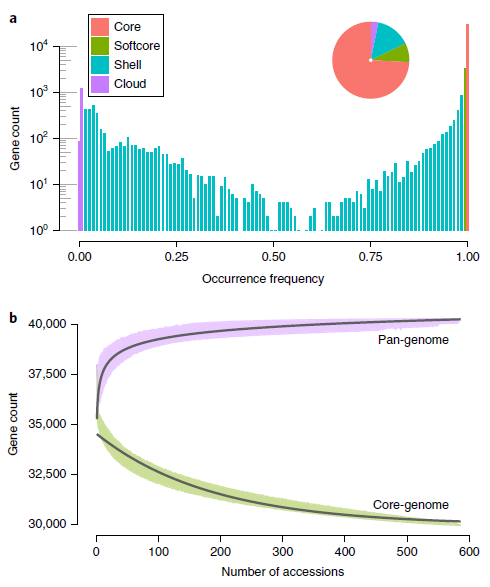
图3 番茄泛基因组基本信息
基因PAV分析
PAV(presence/absence variations)分析通过统计群体中每个个体的基因组包含哪些基因,对个体基因组进行分型,并进行后续的进化分析。在群体中各个个体相对差异较大时,使用PAV分析比使用SNP等变异信息来分析更加能够体现群体内部的差异。而在本研究中,通过对野生、早期栽培和现代栽培的番茄进行PAV比较,可以揭示驯化种植过程中番茄基因组发生变化的过程。野生番茄品系(SP和SCG)包含的基因显著较多,显示番茄驯化中存在基因丢失过程。
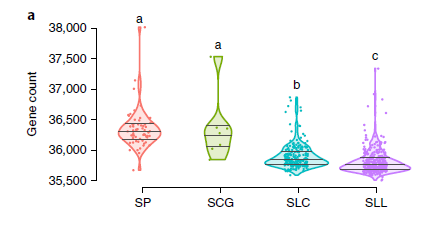
图4 4类番茄基因数量的小提琴图
研究利用PAV数据对番茄各个品系进行了进化分析。从主成分分析的结果来看,SLL与SLC两个栽培品种大致能分离但存在一定的重叠,体现出番茄驯化中的演化进程。野生种群大致聚在一起,但是SP2(SP中一个来源于厄瓜多尔北部海岸的小群体)显示了一定差异,可能是对特殊环境的适应性进化。系统进化树的结果也从侧面支持了这一推论。SLC从厄瓜多尔/秘鲁到中美洲呈现出基因多样性减少的趋势,体现了番茄驯化的地理轨迹。
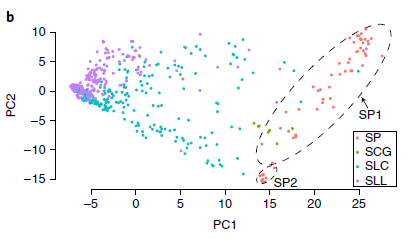
图5 基于PAV数据的主成分分析结果
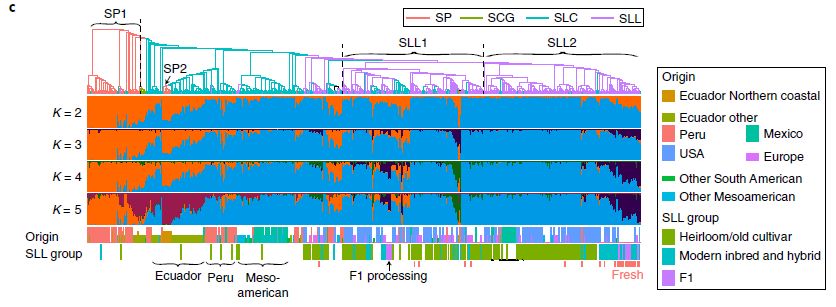
图6 基于PAV数据的系统进化树和structure结果
对SLC vs SP(分析从野生种到栽培番茄驯化历史)和SLC vs SLL传代品种(分析从大果番茄到小果番茄的改良历史)的基因出现频率(occurrence frequency)进行比较,以判断驯化和改良历史中有利(favorable)和不利(unfavorable)的基因。
在这两个过程中,绝大部分基因(94.9%)显现出相同的趋势,即在驯化和改良过程中均呈现不利或有利的相同趋势。而对这些基因进行GO功能富集分析,显示防御反应是在不利基因中最富集的功能类型,尤其是与细胞壁加厚相关的功能。
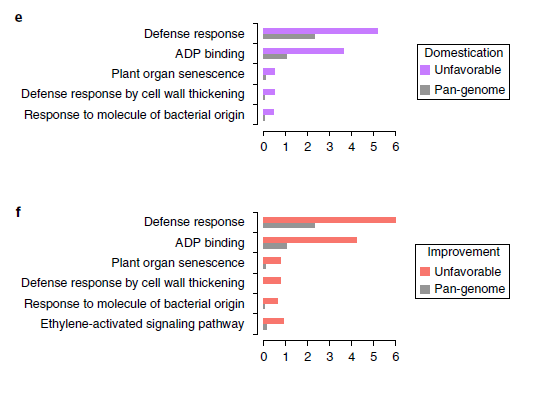
图7 驯化和改良过程中的“不利基因”与整个泛基因组的的GO富集结果比较
对启动子区域(基因上游1kb位置的序列)同样进行了PAV分析,并且得到了与基因PAV分析类似的结果。很多启动子序列在番茄驯化过程中受到选择。利用转录组数据对980个受到选择的启动子下游的基因的表达量数据进行确认,其中有240个基因在有/无启动子区域序列时表达量发生了显著差异,说明栽培过程中通过影响调控序列来对番茄的性状进行选择。
TomLoxC启动子的罕见等位基因
对启动子区域的分析发现了在基因TomLoxC上游启动子区域有一个罕见等位基因。TomLoxC启动子的罕见等位基因在驯化过程中受到显著的负选择,但是在现代番茄的改良过程中可能经历了重引入过程导致等位基因频率升高。该启动子在杂合时,基因的表达量最高,而在纯合时表达量无显著差异。
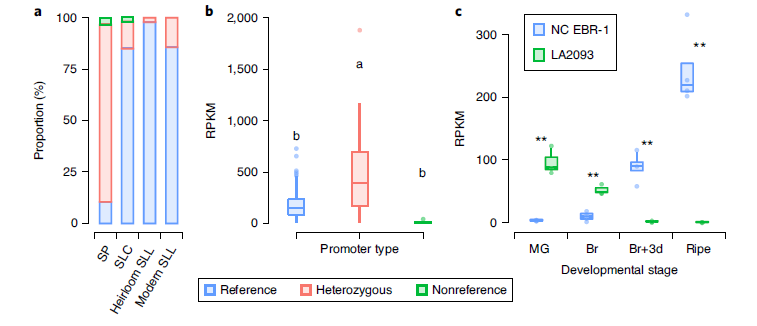
图8 TomLoxC启动子等位基因频率和表达量数据
研究者使用该启动子分别与参考基因组相同和不同的两株纯合亲本NC EBR-1和LA2093杂交进行了QTL定位。两亲本的TomLoxC基因在不同时期的表达模式存在较大差异。QTL定位显示有26种风味物质(脂肪酸来源的挥发性物质和脱辅基类胡萝卜素)含量在基因组上定位到了同一个区间,其中包含TomLoxC基因。在区间包含的所有基因中,TomLoxC基因在亲本之间的表达量差异最大,显示TomLoxC最有可能是性状定位的目标基因,在脱辅基类胡萝卜素的产生过程中扮演重要角色。
研究者通过转基因的方式抑制了一个品种的番茄的TomLoxC基因的表达,发现与原野生型相比,果实中的风味物质含量几乎都有较大的下降。同时在拟南芥中,AtLOX2(TomLoxC基因的近缘基因)出现突变的植株,特定的几种脱辅基类胡萝卜素的含量也出现了明显下降。这些都对TomLoxC基因对番茄风味的影响提供了依据。
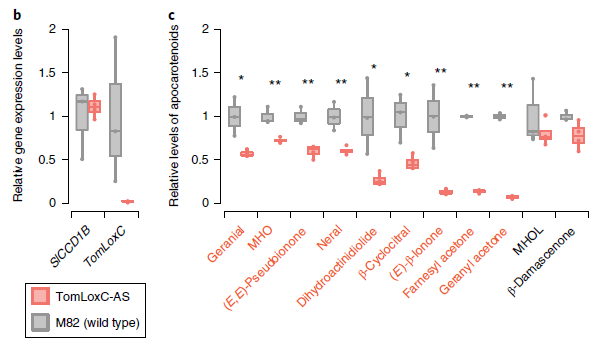
图9 野生型与TomLoxC表达抑制型番茄各类风味物质含量的差异
这类“深度好文”的特点
数据量大:有充足的经费作为基础,并且善于利用之前的研究数据。
样本/数据有代表性:对该领域有足够的了解,对整体和细节均能够把握。
分析思路清晰:对数据的利用方式有清晰的预期,选择最合适的分析方式。
有创造性:利用已有的数据,从不同的角度进行考虑,得到常规分析结果之外的结论。
解决重点关注问题:对学术界重点关注的问题有清楚的概念,能够推进重点关注问题的解决。
行文清楚有逻辑:文章写作逻辑清楚架构合理,主要结果和次要结果区分清晰, 利用图片对结果进行充分的展示和梳理。
参考文献
Zhang JF, Gao L, et al. The tomatopan-genome uncovers new genes and a rare allele regulating fruit flavor[J].Nature genetics, 51, 1044–1051 (2019)
- 本文固定链接: https://maimengkong.com/kyjc/847.html
- 转载请注明: : 萌小白 2022年2月20日 于 卖萌控的博客 发表
- 百度已收录
