首先放一张测序示意图,在DNA片段两端所加的序列即接头序列,也就是我们今天要讲述的主角。
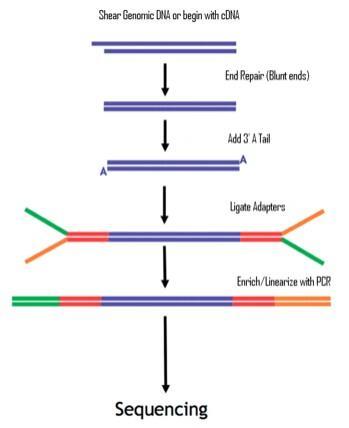 测序示意图
测序示意图
1 基本概念
1.1 adapter
接头,为一段已知的短核苷酸序列,用于链接未知的目标测序片段
1.2 index或barcode
几个碱基组成的寡核苷酸链,用于在混合测序时,区分不同样本
1.3 insert
待测序的目标序列,位于两个adapter之间
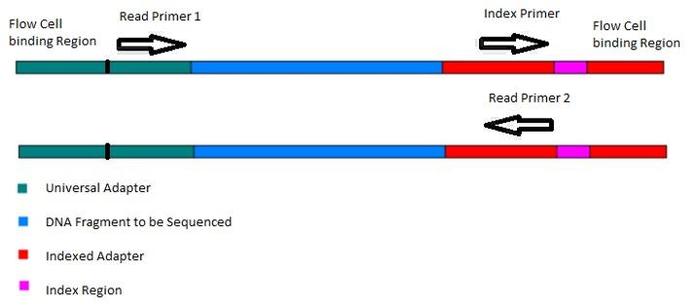 测序片段示意图
测序片段示意图
测序片段包括几个部分:universal_adapter-insert-indexed_adapter。
测序由5'端开始,最开始的几个碱基无法测得,第一个adapter在数据输出时去除,由于测序读长的限制,第二个adapter通常测不到。
但是如果插入片段本身较短,测序会测穿,即会得到 insert-部分adapter 这样的read,这里的adapter便是我们常常提到的需要去除的接头部分。
2 序列信息
2.1 接头序列(示例)
universal adapter:
5’-AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGACGCTCTTCCGATCT-3’
indexed adapter:
5’-GATCGGAAGAGCACACGTCTGAACTCCAGTCAC(barcode)ATCTCGTATGCCGTCTTCTGCTTG-3’
仔细看上面这对接头序列,universal adapter的3'末端的T与待测片段新增的A配对,那么剩余序列的反向互补链为 GATCGGAAGAGCGTCGTGTAGGGAAAGAGTGTAGATCTCGGTGGTCGCCGTATCATT
与 indexed adapter 的前面12个碱基一致 GATCGGAAGAGCACACGTCTGAACTCCAGTCAC,即两段接头序列部分互补,形成Y型的结构。
2.2 index序列
可根据fastq序列中的信息获取
@HWI-ST1276:71:C1162ACXX:1:1101:1208:2458 1:N:0:CGATGT
fastq的格式信息不再赘述,第一行最末的 CGATGT 即本次测序所使用的index。
3 去接头的软件
常见的相关软件包括 Trimmomatic、cutadapt、fastx_toolkit、fastp 等。
关于Trimmomatic的使用,可以参见wangpeng905的文章,介绍很全面。
- 本文固定链接: https://maimengkong.com/kyjc/582.html
- 转载请注明: : 萌小白 2020年1月24日 于 卖萌控的博客 发表
- 百度已收录
