很多在华盈生物完成质谱检测服务后的客户,到了文章发表的阶段都会咨询如何上传原始数据到公共数据库这个问题。对于组学研究来说,文章发表时杂志社一般会要求将原始数据上传到公共数据库,并在正文中提供相应数据库编号供同行下载,以重新进行数据分析和核对。随着蛋白组学的发展,专门供质谱原始数据储存和分享的数据库也出现了,功能也日趋完善。今天,小编将为大家介绍一款目前应用体验度排名靠前的数据库——ProteomeXchange。
ProteomeXchange(简称PX),是应用较为广泛的蛋白质组学质谱数据存储平台(http://www.proteomexchange.org),其旗下包括了PRIDE Archive, MassIVE, PeptideAtlas, jPOST等质谱数据存储平台,接下来我们将一起体验如何一步步上传我们的质谱原始数据到PX上。
一、 ProteomeXchange原始数据的上传
1. 点击下面的网址下载标准的提交工具PX Submission tool,软件基于Java开发环境,请确保电脑已经安装Java。PX Submission tool无需安装,解压文件夹即可直接运行。网址:https://www.ebi.ac.uk/pride/help/archive/submission
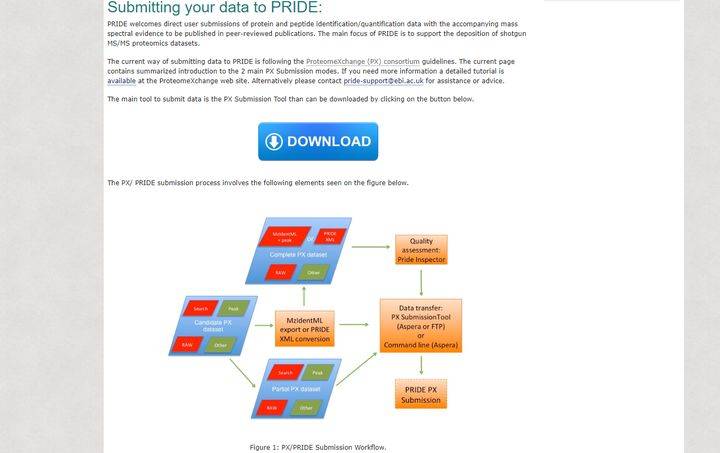
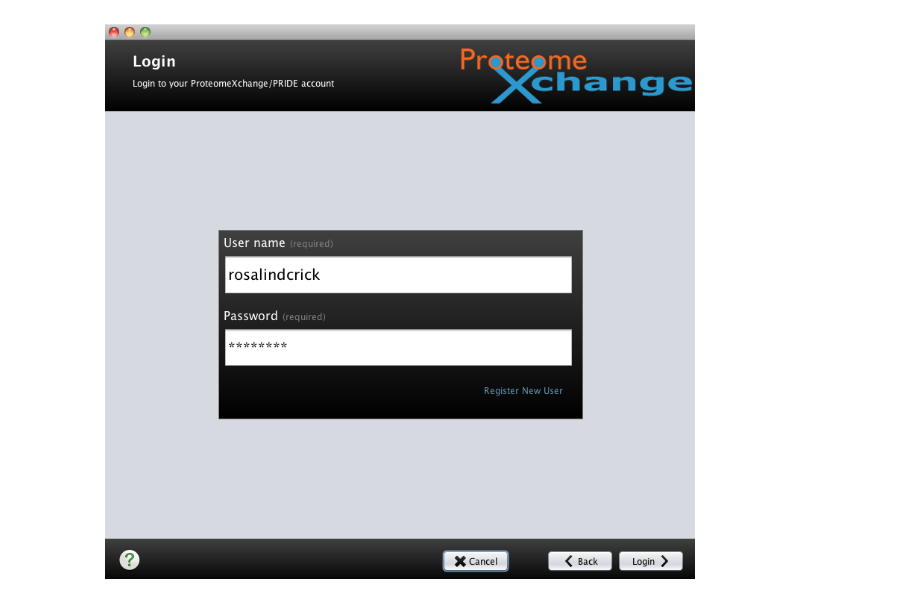
2. 登录账号,若之前未使用的话需要进行注册。
(注册网址:https://www.ebi.ac.uk/pride/archive/register)
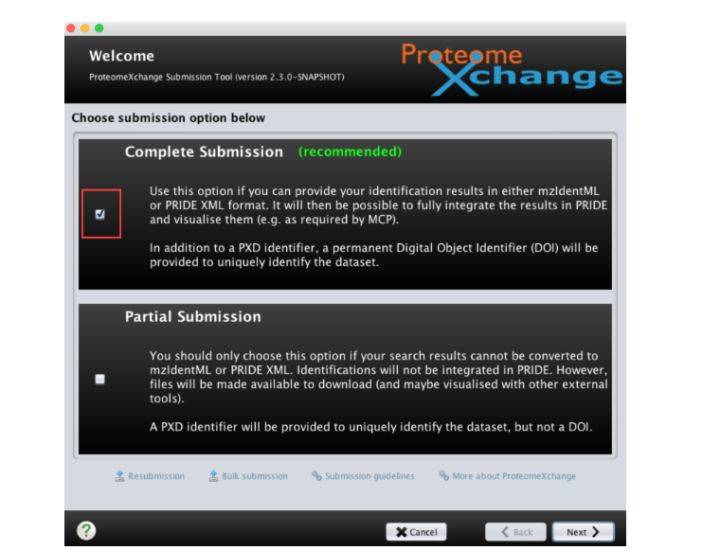
3. 选择提交类型(在实际使用过程中,partial submission为更经常用到的模式)。
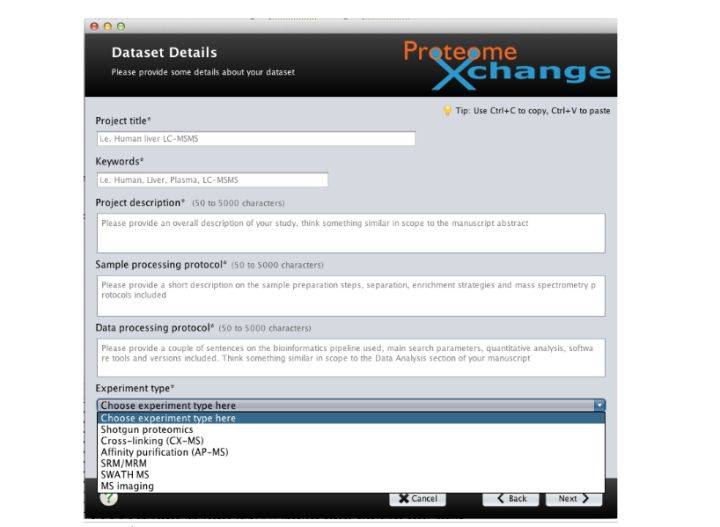
4. 提交原始数据对应的相关信息。
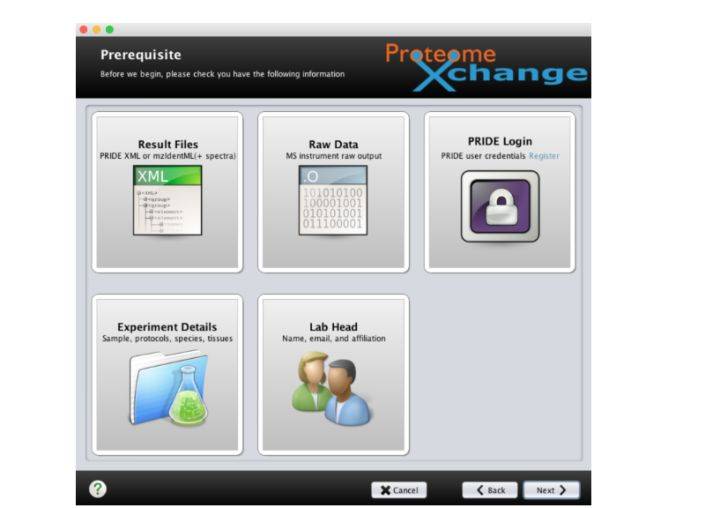
5. 点击Add Files添加项目原始raw文件搜库文件,Fasta序列数据库,搜库结果,项目相关信息等等。若导入的File Type经软件识别类型有误时,可点击对应的文件进行文件类型的修改。
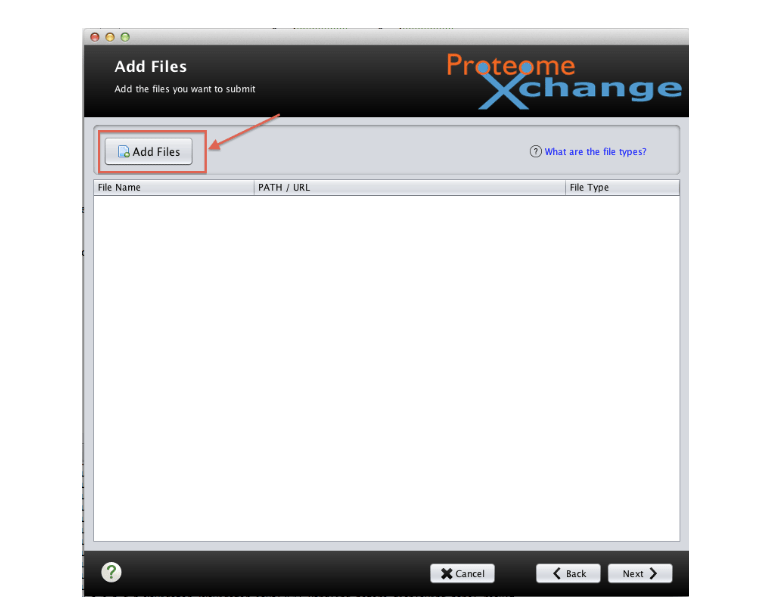
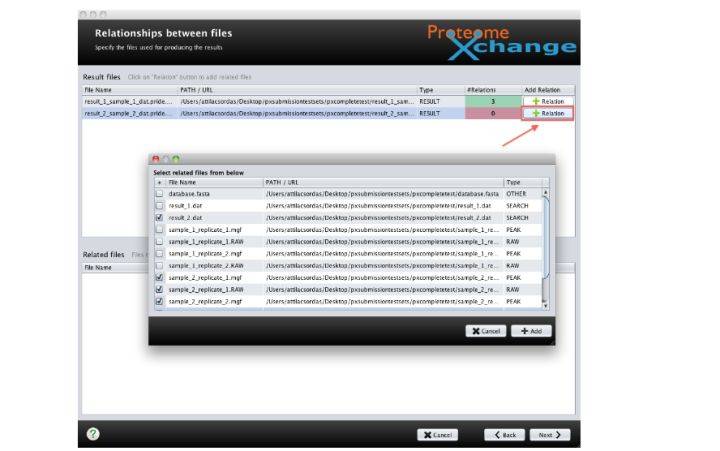
6. 加载进所有文件之后,需要添加Result结果和原始文件之间的相关性。
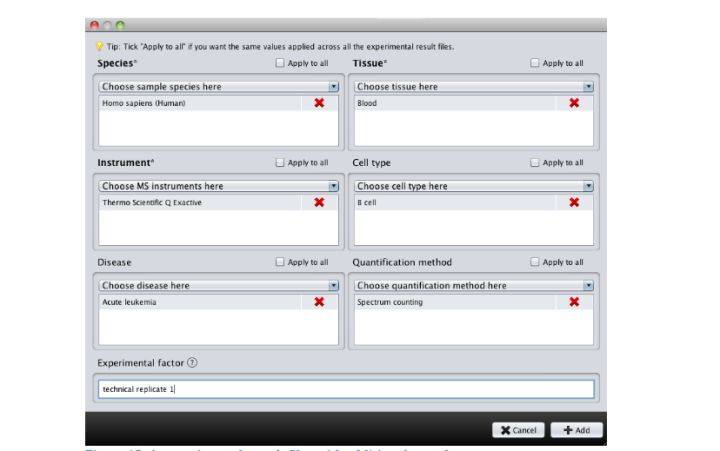
7. 添加物种,样本类型,质谱仪器和修饰等相关信息。(带*号的为必填项)
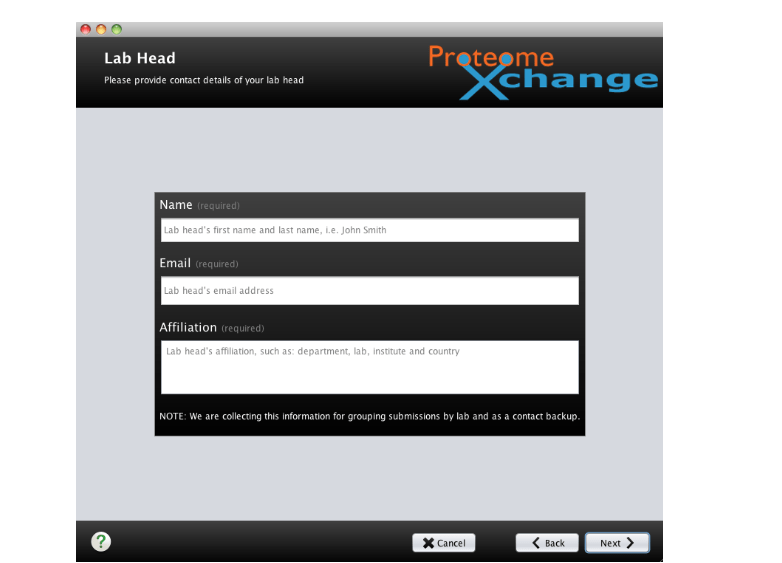
8. 提供实验室负责人或者课题组老师的联系方式等。
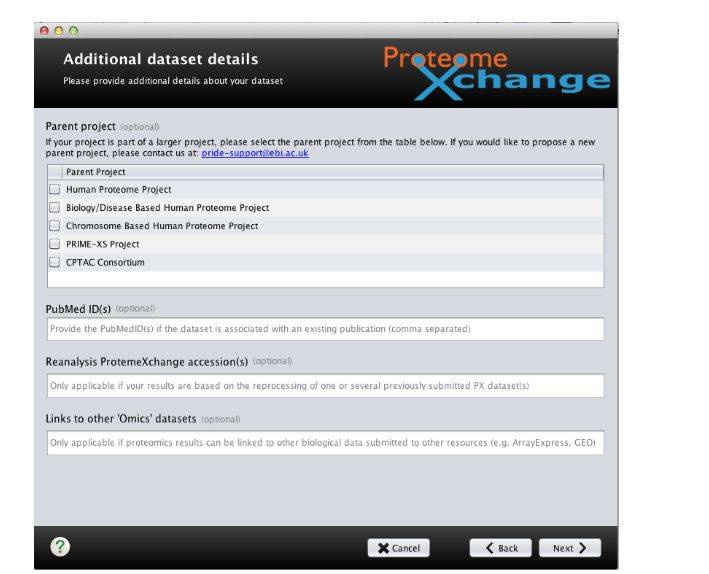
9. 如果本次上传的数据是一个大型研究项目的一部分,或者在数据处理过程中采用了其他发表文献数据或其他数据集数据则需要提供相关信息。
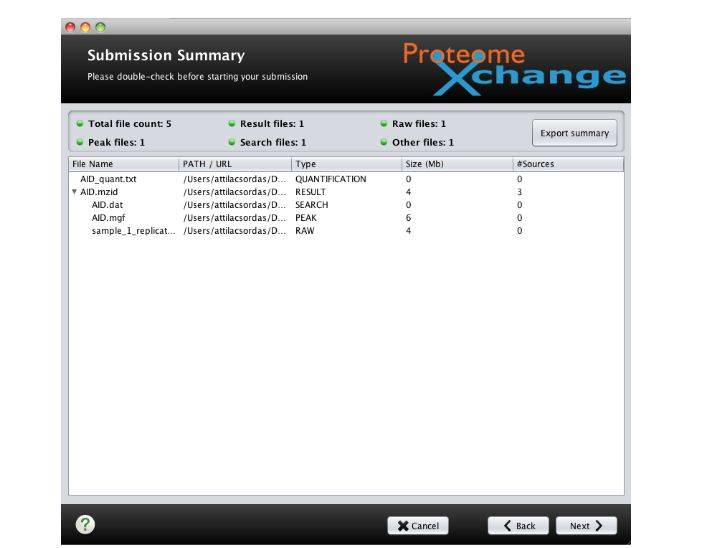
10. 对已上传的数据进行核对,若无误点击Next进行数据提交。
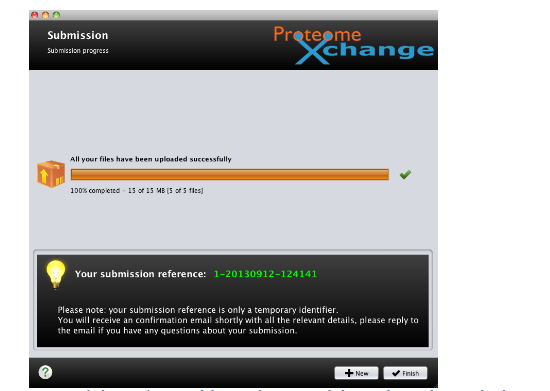
提交成功后,邮箱会收到一条提醒原始数据提交成功的邮件,待数据通过审核之后ProteomeXchange会再次通过邮件提醒Project accession(也可以通过登录网站:https://www.ebi.ac.uk/pride/login进入个人中心查看)。
二、 上传数据内容说明
上传的数据集(dataset)往往包含质谱仪下机的原始文件(Raw data)、搜库完成后的结果文件以及实验信息等。其中质谱原始数据,我们称之为“RAW”,常见为质谱仪直接输出的raw格式文件,或者经过加工后的XML形式文件(mzXML或者mzML);搜库结果文件为ProteinGroups和Peptides蛋白肽段鉴定结果,称之为“RESULTS”。对于微生物等特殊物种,可以选择上传搜库时所比对的DNA测序转换成的氨基酸序列数据文件(一般为.fasta后缀的格式文件)等等。华盈生物目前报告中常用的质谱搜库软件包括Maxquant软件和Proteome Discoverer软件(PD)等。上传数据库时,一般针对Maxquant软件搜库结果,选择上传原始下机Raw文件+搜库结果文件search(可以将table文件夹中所有文件压缩得到压缩包直接上传)。
针对PD软件搜库结果,选择上传原始下机Raw文件+蛋白鉴定列表+搜库searchsummary结果。
转自:华盈生物
- 本文固定链接: https://maimengkong.com/kyjc/1850.html
- 转载请注明: : 萌小白 2024年12月11日 于 卖萌控的博客 发表
- 百度已收录
