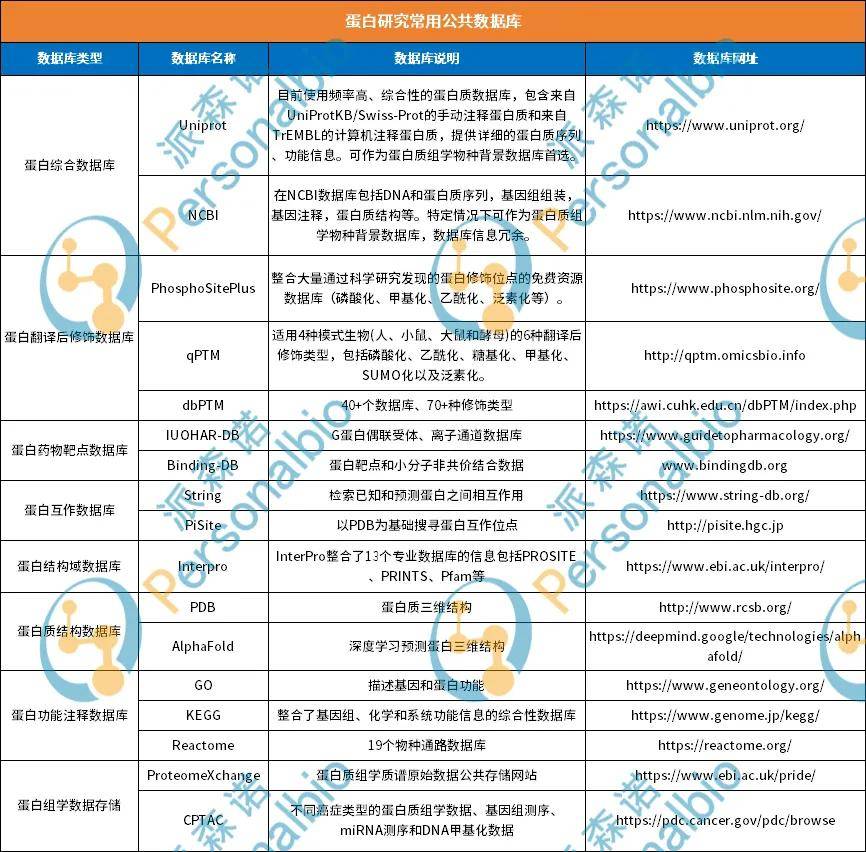
当我们投身于蛋白质的研究时,探索的焦点通常汇聚于蛋白质表达的动态变化、蛋白质发生翻译后修饰的精妙调控、蛋白质间错综复杂的相互作用、蛋白质所参与的生命过程和功能、三维结构的奥秘以及作为药物靶点的潜力。每一个研究方向都蕴含着丰富的知识并彼此交织,共同编织出蛋白质世界的复杂图景。为了应对这一领域的复杂性,我们精心整理了一系列常用的蛋白质研究数据库,旨在为科研工作者提供高效便捷的信息资源,节省宝贵的时间与精力。
一、蛋白综合数据库
1.1 Uniprot
UniProt(Universal Protein Resource,https://www.uniprot.org/)是一个免费开放的综合性蛋白质数据库。该数据库蛋白信息来源于EMBL、GenBank、DDBJ等公共数据库(非冗余蛋白质序列数据库),目前使用频率非常高,包含信息非常全面。该数据库主要提供了蛋白质序列和丰富的功能注释信息,用途多样,是查询蛋白功能和蛋白质组学研究中搜库匹配的首选数据库。应用工具搜索不仅可以进行序列-物种/序列-序列比对,还可以对不同数据来源的ID进行转换。
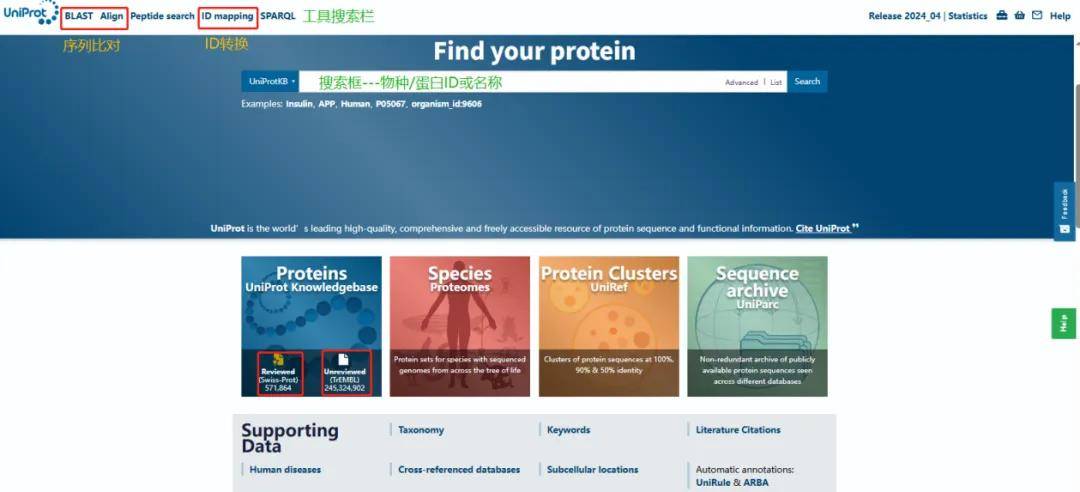 图1.1 Uniprot页面介绍
图1.1 Uniprot页面介绍
常用的UniProtKB由两个子库构成:Swiss-Prot和TrEMBL。其中Swiss-Prot通常来源于已发表的文献,是经过人工验证和注释的高质量和可靠的非冗余蛋白质注释数据,人工注释这些数据效率较低。基于基因组序列由机器自动翻译和预测的蛋白质序列数据库TrEMBL建立弥补了人工注释的不足,并提供了大量新蛋白质信息,但其注释程度不如Swiss-Prot高。
当我们做蛋白质组学涉及以上两个子库的选择问题时,如果对鉴定的准确度要求更高,可以选择下载Swiss-Prot数据库进行搜库,常规物种做蛋白质组学通常选择Swiss-Prot。如果为了鉴定蛋白更加全面,通常建议选择UniprotKB的总蛋白序列信息进行搜库。
 图1.2 Uniprot子库下载
图1.2 Uniprot子库下载
此外Uniprot数据库包含丰富的功能模块,主要包含:蛋白序列、结构域、亚细胞定位、翻译后修饰、表达情况、蛋白互作等,可以直接输入蛋白质ID或者名称进行查询该蛋白参与的生物学过程。
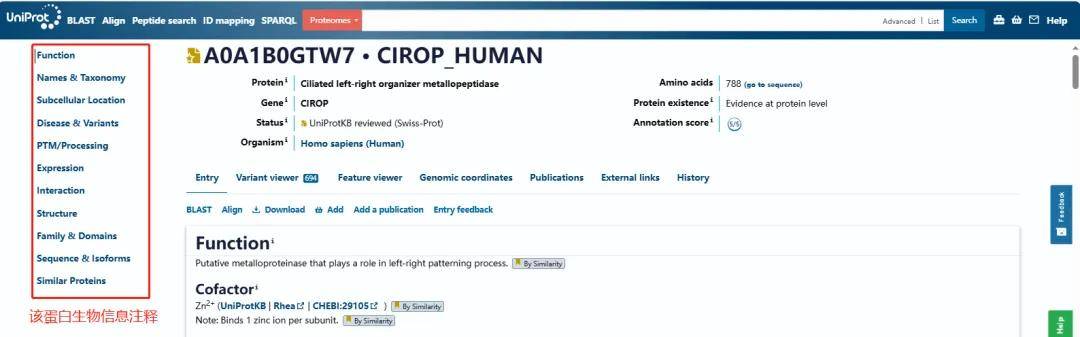 图1.3 Uniprot 蛋白功能注释信息
图1.3 Uniprot 蛋白功能注释信息
1.2 NCBI
NCBI(National Center for Biotechnology Information,美国国家生物技术信息中心,https://www.ncbi.nlm.nih.gov/)数据库包含了大量关于基因、蛋白质、核酸序列、疾病、药物、解剖学、文献等多个方面的信息,收录全世界所有实验室检测信息,是一个综合性数据库,NCBI的数据库内容更为广泛和综合,可以提供36种不同的数据检索及分析工具。
NCBI也可以作为蛋白质组学的物种背景数据库,搜索物种信息即可得到RefSeq蛋白信息,但是会有很多冗余的蛋白信息。因此同一个物种,NCBI的蛋白比UniProt多,假阳性也会随之升高。
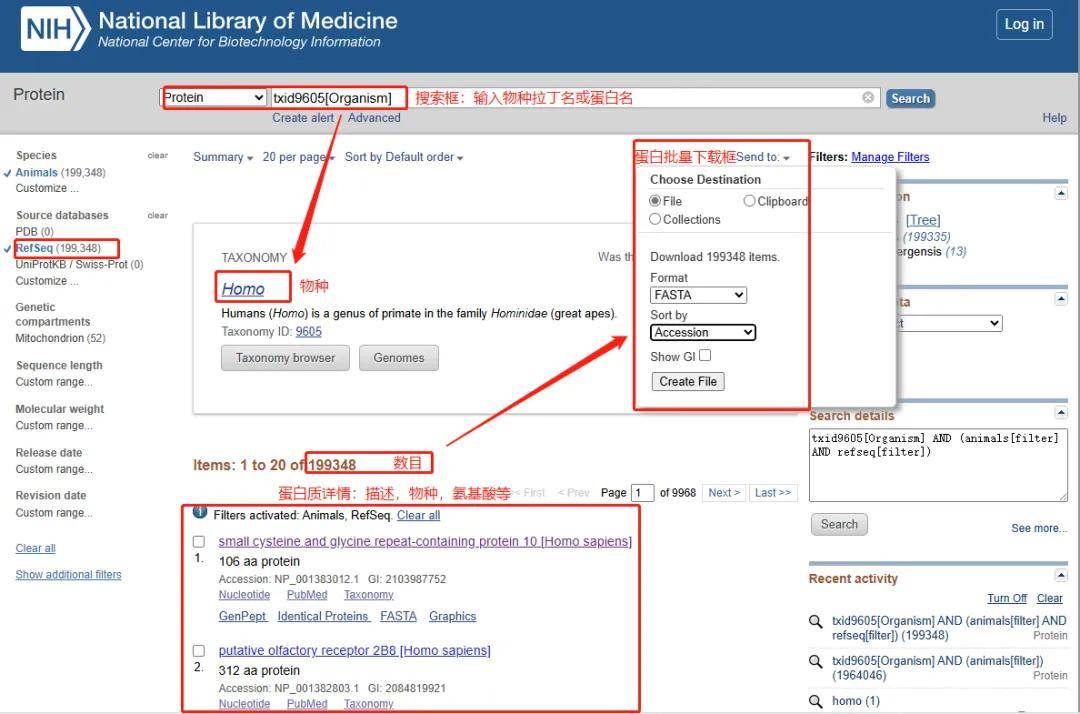 图1.4 NCBI 蛋白使用下载
图1.4 NCBI 蛋白使用下载
关于蛋白质组学搜库数据库的选择(NCBI vs Uniprot),通常建议优先使用Uniprot数据库,若该物种Uniprot数据库蛋白较少,可使用NCBI数据库进行搜库。若特定物种在Uniprot和NCBI中均没有蛋白数据,优先考虑基因组或转录组测序序列翻译成的蛋白质数据库,也可以使用上一级或者近缘物种的蛋白质数据库作为备选数据库。
二
蛋白翻译后修饰数据库
蛋白质翻译后修饰(Post-translational modification,PTM)是指在翻译后的蛋白质氨基酸残基上通过添加或移除特定的基团进行化学修饰,从而调节蛋白质的活性、定位、以及蛋白与其他生物大分子间相互作用。
2.1 PhosphoSitePlus
PhosphoSitePlus数据库(https://www.phosphosite.org/)是一个由CST和NIH联合开发免费的翻译后修饰预测数据库,整合了大量来自高通量测序预测和科学研究实验验证的结果,为蛋白质翻译后修饰的研究提供了全面的信息和工具。该数据库主要包括磷酸化、甲基化、乙酰化、泛素化等,共收录了59499个蛋白的600798个翻译后修饰位点。通过查询蛋白质可以获得蛋白质基本信息(结构域、亚细胞定位)以及蛋白质发生修饰的类型、修饰位点、抗体、修饰相关疾病,以及激酶底物序列。
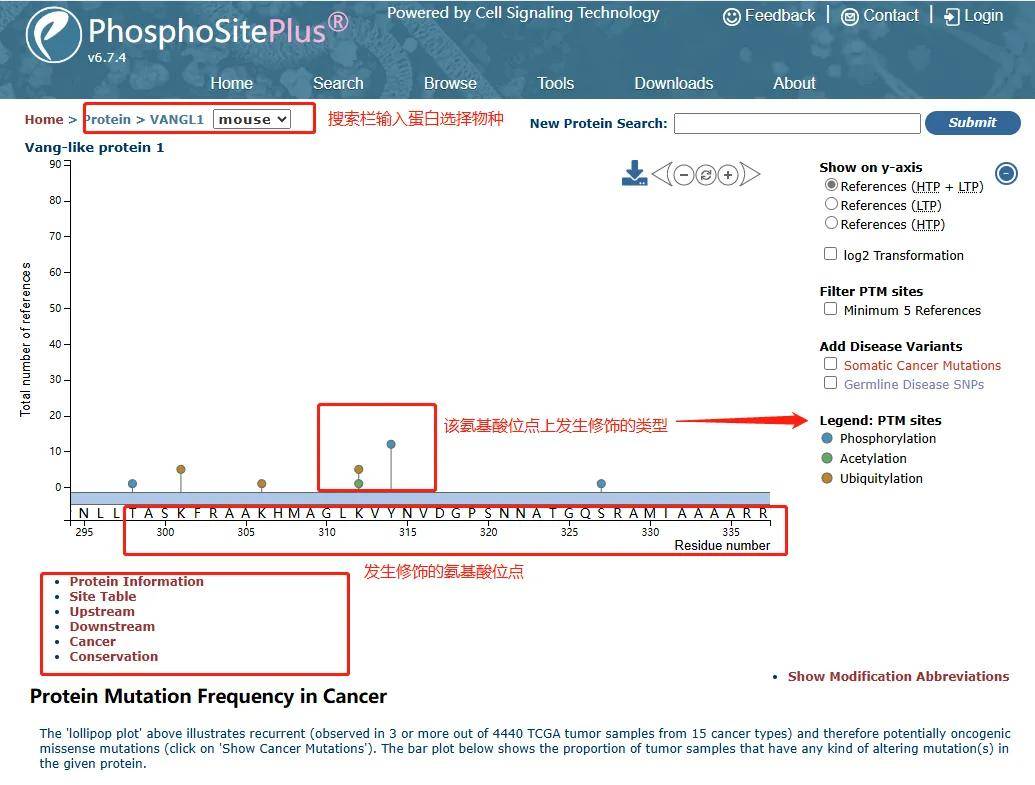 图2.1PhosphoSitePlus数据库使用
图2.1PhosphoSitePlus数据库使用
2.2 qPTM
qPTM(quantification of Post-Translational Modifications,http://qptm.omicsbio.info)是中山大学肿瘤防治中心刘泽先教授团队收集并整合PTMs文献的数据库,涉及从600多个已发表研究中收集的四种不同生物体(人、大鼠、小鼠、酵母)中40728个蛋白质在2596种条件下的660 030个非冗余PTM位点,修饰类型包括6种(磷酸化、乙酰化、糖基化、甲基化、SUMO化以及泛素化修饰)。通过搜索特定物种的蛋白,即可获得前人研究的修饰发生的位点以及实验条件和参考文献。
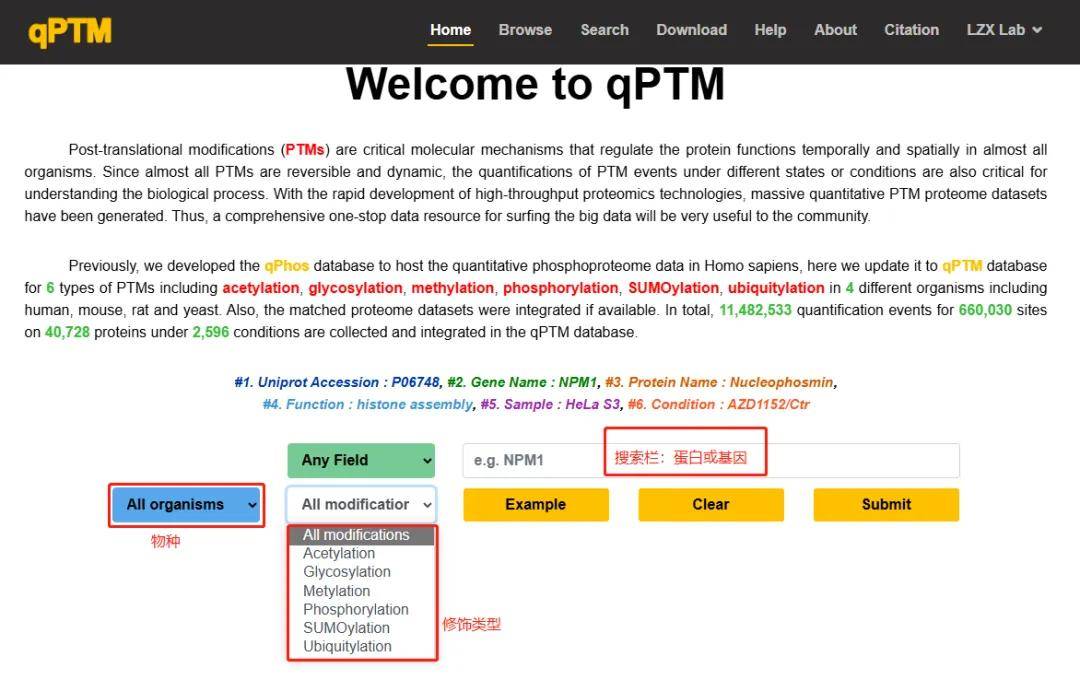
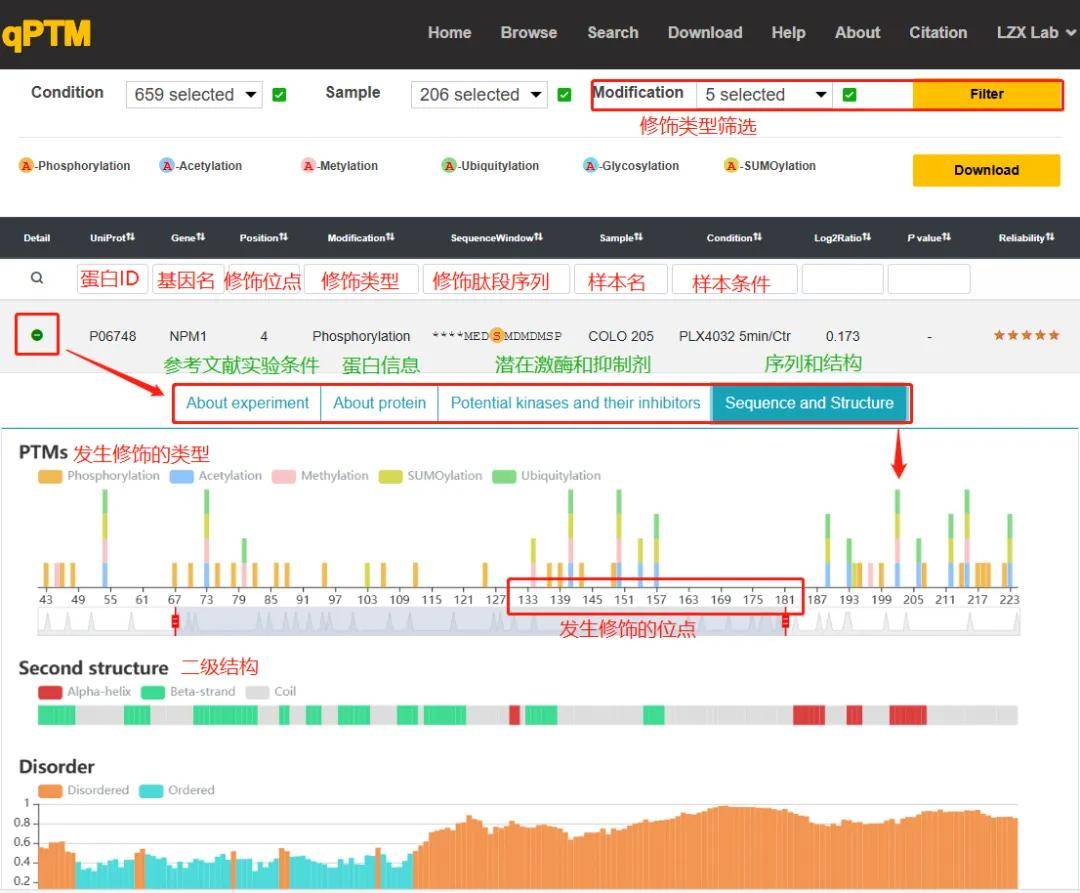 图2.2 qPTM数据库使用
图2.2 qPTM数据库使用
2.3 dbPTM
dbPTM(https://awi.cuhk.edu.cn/dbPTM/index.php)是蛋白质翻译后修饰 (PTM) 的综合资源,整合来自40+数据库、70+种修饰类型、已经被实验/文献证实的PTM位点和预测位点共2235664个,其中重点修饰类型包括磷酸化、糖基化和硫修饰。通过搜索蛋白可获得蛋白二级结构、修饰位点信息、上游调节蛋白、位点功能以及疾病相关信息。
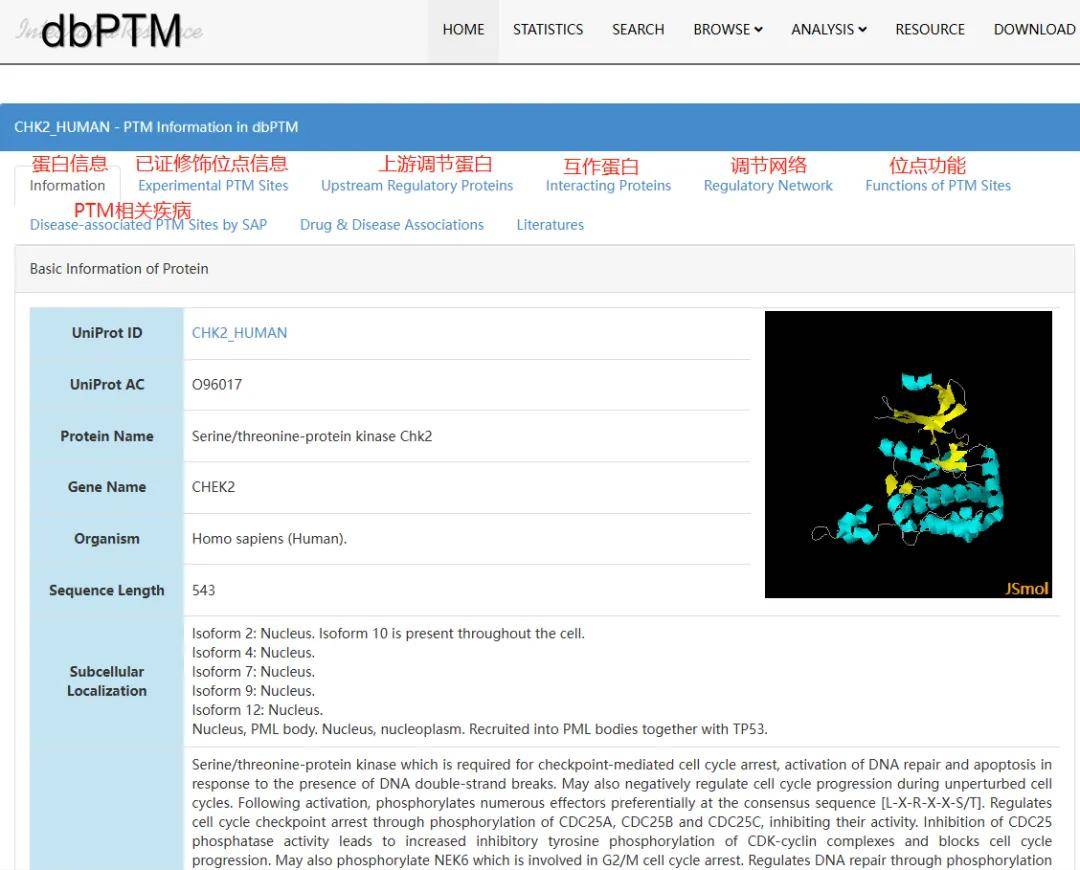 图2.3dbPTM数据库使用
图2.3dbPTM数据库使用
2.4 Plant PTM Viewer
Plant PTM Viewer(http://www.psb.ugent.be/PlantPTMViewer)是植物蛋白翻译后修饰数据库,包含8种不同植物(拟南芥、水稻、大豆、小立碗藓、番茄、玉米、小麦、莱茵衣藻)大约128920个蛋白334255个PTM位点的33种蛋白质修饰。通过该网站我们可以检索目的蛋白在植物中的修饰情况,此外还可以搜索同源序列中的保守翻译后修饰位点。
 图2.4Plant PTM Viewer数据库使用
图2.4Plant PTM Viewer数据库使用
三、蛋白质互作数据库
一般情况下蛋白质很难单独发挥作用,都是由多个蛋白质分子的相互协调共同实现复杂的细胞功能。对于已知蛋白与哪些未知蛋白具有结合作用,我们可通过Co-IP结合质谱鉴定技术(运用蛋白质相互作用数据库)对研究有更深入的了解。
3.1 String
STRING(Search Tool for Retrieval of Interacting Genes/Proteins,https://www.string-db.org/)数据库整合了多个数据源的PPI信息,包括实验数据、文献挖掘和计算预测。它提供了广泛的物种覆盖和功能注释,可用于PPI网络构建和功能分析,涵盖了5090种生物的两千四百多万种蛋白质,是目前蛋白质互作数据库中覆盖物种和互作信息尤其全面的一个数据库。主要可以进行对已知蛋白与之互作的未知蛋白进行分析,结果互作网络图可根据Score值评估互作,Score分越高,互作可能性越大。
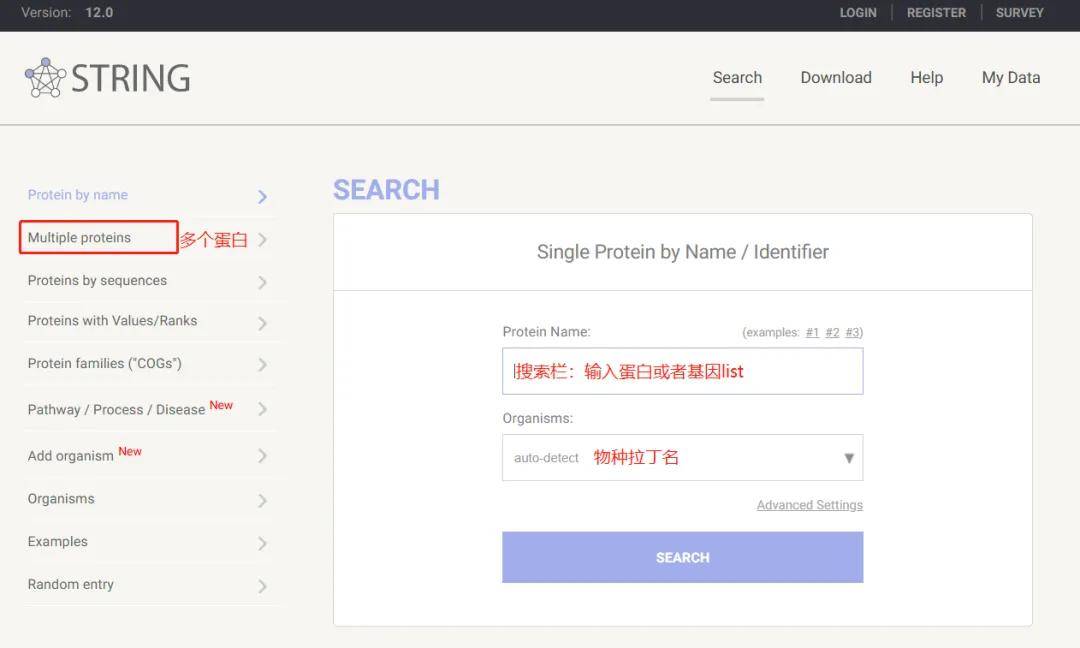 图3.1STRING数据库使用
图3.1STRING数据库使用
3.2 PiSite
PiSite(Database of Protein Interaction Sites,http://pisite.hgc.jp)通过基于大量实验和计算方法中获取的蛋白质结构数据,分析不同的PDB条目来识别蛋白质链上的结合位点。它整合各种蛋白质相互作用位点的信息,包括氨基酸残基相互作用、结合能力以及结构特征等,来构建一个全面的数据库。
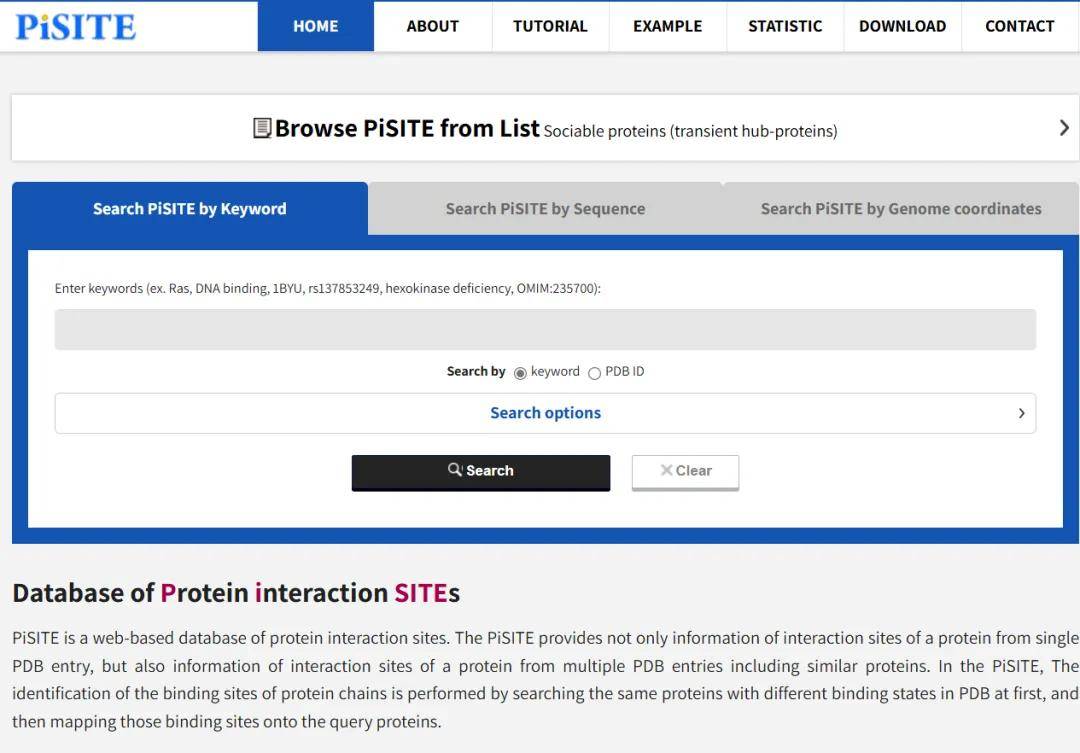 图3.2PiSite数据库使用
图3.2PiSite数据库使用
四、蛋白药物靶点数据库
4.1 IUOHAR-DB
IUOHAR-DB(https://www.guidetopharmacology.org/)是G蛋白偶联受体、离子通道数据库,提供这些蛋白的基因、功能、结构、配体、表达图谱、信号转导机制、多样性等数据。可以用于药物靶点查找,可以按照免疫过程信号通路查询或者在不同细胞特异表达查询或者根据蛋白激酶、离子通道分类进行查询。
 图4.1IUOHAR-DB数据库使用
图4.1IUOHAR-DB数据库使用
4.2 Binding-DB
Binding-DB(Bindind Database),加州大学圣地亚哥分校 Michael K.Gilson实验室发布的一个可公开访问的主要收集药物靶点蛋白质和类药小分子之间相互作用亲和力的数据库。BindingDB的数据来自相关文献报道数据、专利信息、PubChem BioAssays 数据和 ChEMBL 记录数据。BindingDB 收录了110万个化合物与8800个靶点之间的250万个相互作用数据。
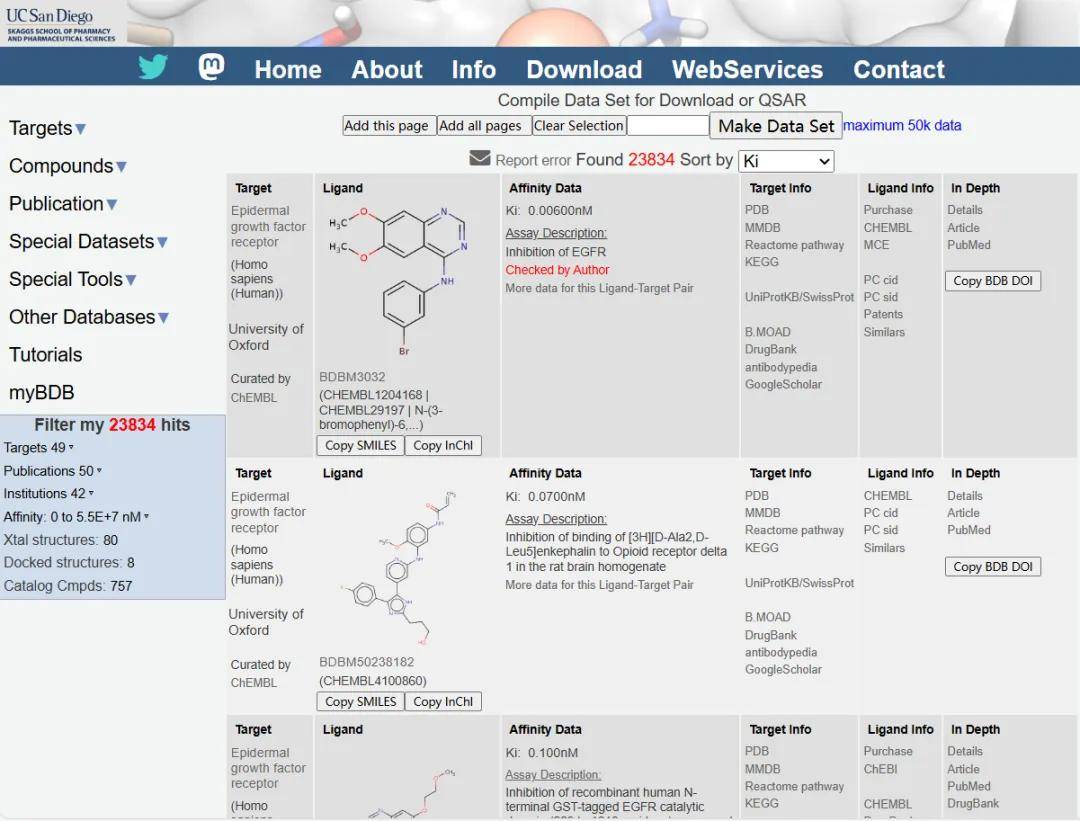 图4.2Binding-DB数据库使用
图4.2Binding-DB数据库使用
五、蛋白结构域数据库
每一种蛋白质都有其独特的功能和结构,这构成了生物多样性的一部分。为了揭示这种多样性需要对成千上万的蛋白质进行分类和功能预测。
5.1 InterPro
InterPro(https://www.ebi.ac.uk/interpro/)将13个蛋白质特征数据库合并为一个集中资源,包括Coils、Gene3D、Pfam、PRINTS、ProSitePatterns、ProSiteProfiles、SMART、SUPERFAMILY、TIGRFAM、ProDom、PIR等数据库。可以直接搜索蛋白序列或者结构域ID获得结构域结果。
 图5.1InterPro数据库使用
图5.1InterPro数据库使用
六、蛋白三维结构数据库
蛋白质的生物活性不仅决定于蛋白质分子的一级结构,而且与其特定的空间结构密切相关。异常的蛋白质空间结构很可能导致其生物活性的降低、丧失。在功能和结构细节上阐明关于蛋白质折叠的过程将对相关疾病的预防和治疗有重要意义。
6.1 PDB
PDB(Protein Data Bank,http://www.rcsb.org/),是美国Brookhaven国家实验室于1971年创建的,通过X射线单晶衍射、核磁共振、电子衍射等实验手段确定的蛋白质、多糖、核酸、病毒等生物大分子的三维结构数据库,通过搜索蛋白质可以获得蛋白质结构的三维可视化(如果有配体相互作用)和结构质量指标。
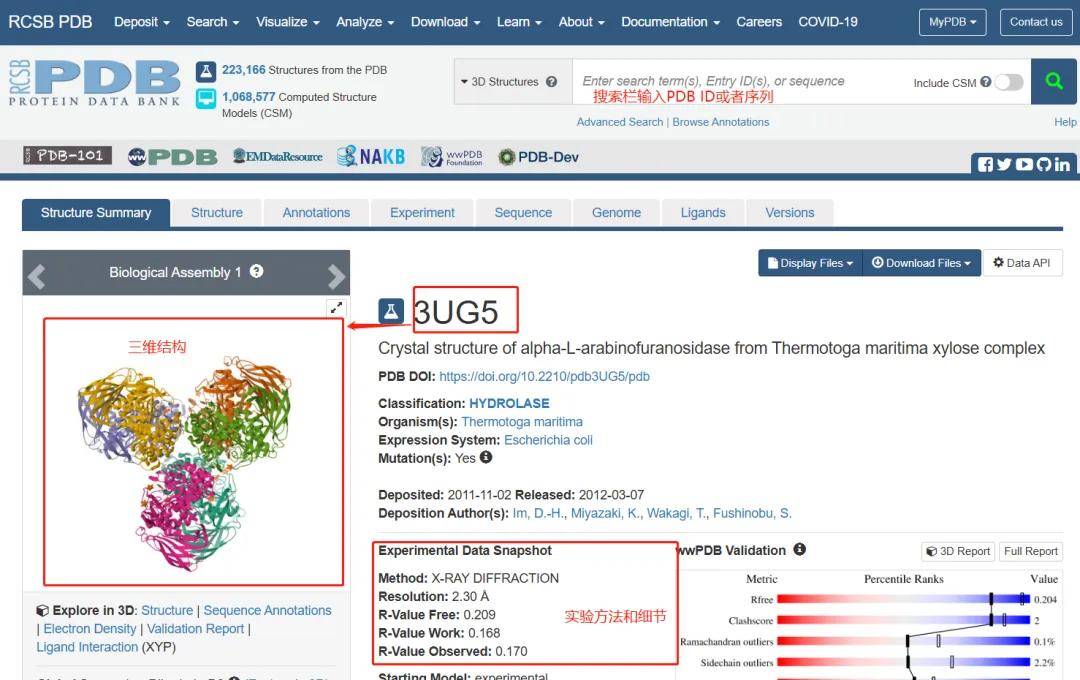 图6.1PDB数据库使用
图6.1PDB数据库使用
6.2 AlphaFold
AlphaFold(https://deepmind.google/technologies/alphafold/)是由谷歌DeepMind开发的一款蛋白质结构预测程序,它采用 AI 和深度学习技术仅根据其基因序列就能预测蛋白质的3D结构,仅需数日内可识别蛋白质的形状,从而找到药物靶点。
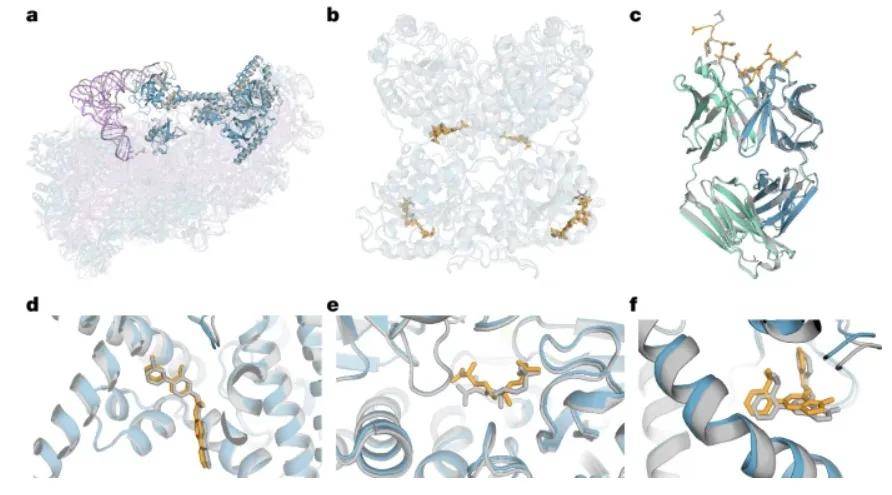 图6.2AlphaFold预测蛋白
图6.2AlphaFold预测蛋白
七、功能注释数据库
7.1 KEGG
KEGG(https://www.genome.jp/kegg/)是一个整合了基因组、化学和系统功能信息的综合性数据库,连接已知分子间相互作用的信息网络,如代谢通路、复合物、生化反应。KEGG途径主要包括:代谢、遗传信息处理、环境信息处理、细胞过程、人类疾病、药物开发等。KEGG包含多个子数据库,有代谢通路、基因信息、化合物、酶、药物等等,均包含大量有用的信息,经常使用的是Pathway查询与分析 。
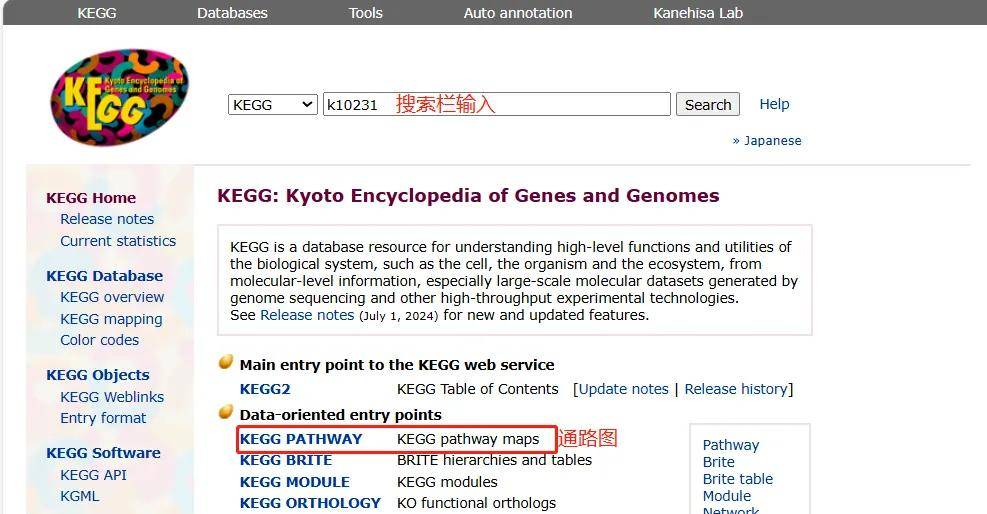 图7.1KEGG数据库使用
图7.1KEGG数据库使用
7.2 Reactome
Reactome(https://reactome.org/)是一个免费、开源、数据经过手动筛选和同行评审的生物分子通路知识数据库。目前该库覆盖了19个物种的通路研究,包括经典的代谢通路、信号转导、基因转录调控、细胞凋亡与疾病。输入蛋白或者基因搜索即可得到相关通路信息,并可以对感兴趣的通路进行富集分析。
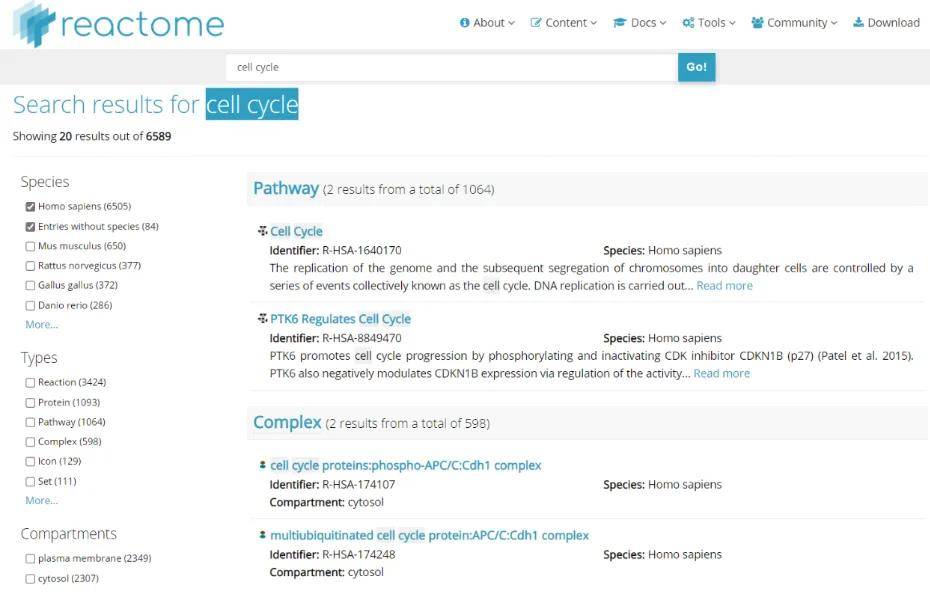
 图7.2Reactome数据库使用
图7.2Reactome数据库使用
7.3 GO
GO(Gene Ontology,https://www.geneontology.org/)数据库为统一基因功能描述而建立,Gene Ontology把描述基因和基因产物功能的术语归纳到三个不同维度的本体中(BP-生物过程、CC-细胞组分、MF-分子功能)。搜索感兴趣的蛋白或者基因ID获取相关信息。
八、Human Protein Atlas
Human Protein Atlas-人类蛋白质图谱(HPA,https://www.proteinatlas.org)瑞典 Knut & Alice Wallenberg基金会创建,利用各种技术,包括基于抗体的成像技术、基于质谱的蛋白质组学、转录组和系统生物学等,绘制细胞、组织和器官中的人类蛋白质图谱。人类蛋白质图谱由十二个独立的部分组成,包括组织图谱、脑图谱、单细胞图谱、组织细胞类图谱、病理图谱、疾病血液图谱、免疫细胞图谱、血液蛋白图谱、亚细胞图谱、细胞系图谱、结构图谱、相互作用图谱。通过搜索蛋白获得蛋白质在多种人类正常组织、肿瘤组织、细胞系和血液细胞内的分布和表达情况。
九、蛋白组学数据存储网站
9.1 ProteomeXchange
ProteomeXchange(https://www.ebi.ac.uk/pride/)是专门用来储存蛋白检测数据的网站。例如通过检索特定癌症,可以在 ProteomeXchange 得到某类癌症有关的蛋白质组学数据集,并可以详细看到每一个数据集的基本信息,包括研究的物种、疾病、使用的蛋白质组学方法以及仪器等信息,并提供该数据集的原始文件以供研究者下载。

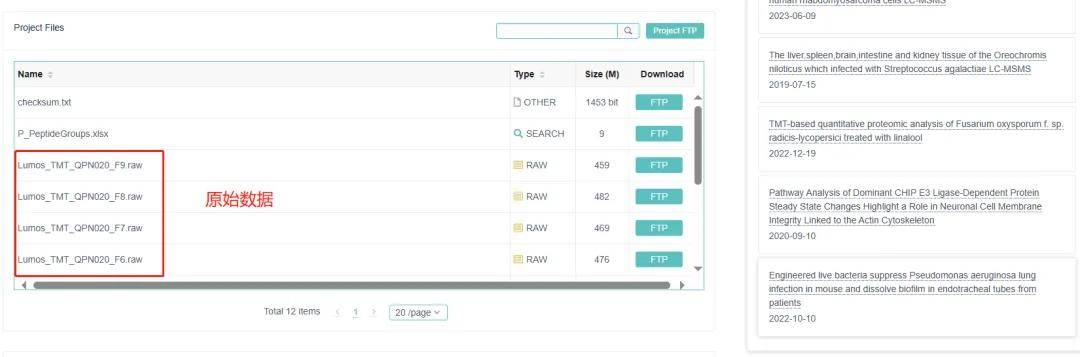 图9.1ProteomeXchange网站时使用
图9.1ProteomeXchange网站时使用
9.2 CPTAC
CPTAC(Clinical Proteomic Tumor Analysis Consortium,https://pdc.cancer.gov/pdc/browse)是由美国国家癌症研究所(NCI)资助建立的一个综合性数据库,CPTAC数据库主要提供了临床队列不同癌症类型的蛋白质组学数据,此外还包含基因组测序、miRNA测序和DNA甲基化数据,希望通过应用大规模蛋白质组学和基因组分析(proteogenomics)来加速对癌症分子基础的理解。在首页搜索栏输入蛋白、基因或者疾病可以获得数据集。
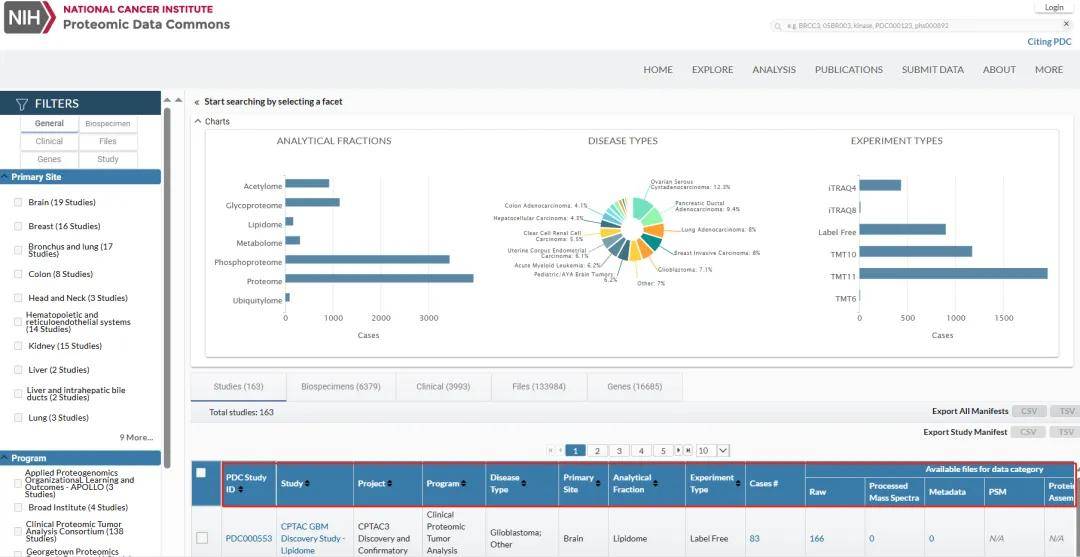 图9.2CPTAC数据使用
图9.2CPTAC数据使用- 本文固定链接: https://maimengkong.com/kyjc/1759.html
- 转载请注明: : 萌小白 2024年8月8日 于 卖萌控的博客 发表
- 百度已收录
