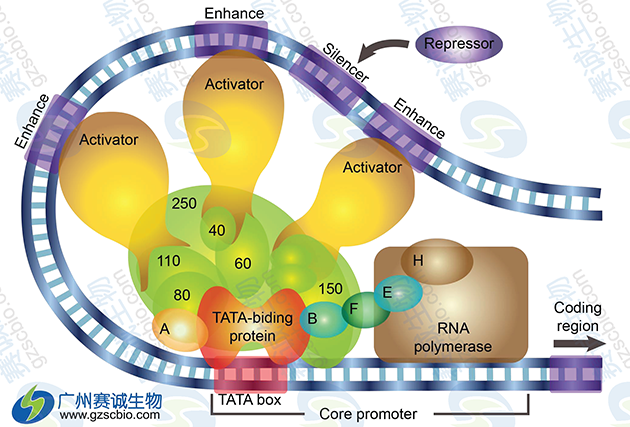
图1 真核生物转录调控原理
转录因子结合位点(Transcription factor binding site,TFBS)是与转录因子结合的DNA片断,长度通常在5~20 bp范围内,它们与转录因子相互作用调控基因的转录过程。确定 TFBS 是理解转录调控机制 , 建立转录调控网络的关键问题。
一个转录因子往往同时调控若干个基因,而它在不同基因上的结合位点具有一定的保守性,又不完全相同。较短的DNA片段在规模较大基因组中重复出现的次数很多,另外TFBS又允许一定的可变性,这给识别TFBS的工作带来了困难,使得预测TFBS的算法普遍存在假阳性率偏高的问题。
结合位点序列目前主要有3类描述模型:1)串模型:最常用的是共有序列模型2)位置频率矩阵:是一种用统计学方法对转录因子与DNA的相互作用进行建模的方法。3)使用信息论中熵的知识,用图形方式来形象、直观的表示结合位点。
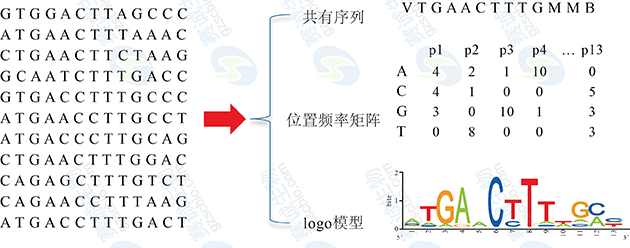
图2 转录因子结合位点的表示
在UCSC Genenome Browser数据库里面Regulation调控卡ENCODE TBBS里面有1000多套转录因子的数据。基本可以满足医学科研的需求。
基因转录调控是一个非常复杂的网络体系,在in vitro和in vivo的实验可能有不同的结果,而生物细胞生理状态以及环境因素的不同也可能导致不同的实验结果。只有通过各种数据的融合和相互校正,才能挖掘出可靠的转录调控关系和TFBS。比如说,将基因表达数据和序列数据进行融合分析,既保证TF与该基因的调控序列有相互结合,又保证该TF对该基因的表达有影响,从而能够确信二者之间的调控关系。有效的利用生物信息学工具分析实验结果,从而产生出可以验证的生物学假设,会使TFBS的预测及鉴定更加准确和高效。
传统上,TFBS识别方法主要可分为两大类:一类是基于字串的方法。这种方法主要是通过对多联核普酸短序列的计数和频率的统计来识别,其中最常用的方法是共有序列模型(ConsensuSModel)。第二类是基于概率序列模型的方法,如期望最大化(ExpeetationMaximization,EM)和吉布斯采样(Gibbssampling)等方法。随着实验方法的改进,尤其是高通量实验方法的出现,近两年出现了一些针对CHIP一CHIP以及CHIP一SEQ实验数据的处理方法。微阵列试验方法成为使用广泛,技术相对成熟的一种TFBS识别方法。
TFBS的生物信息学领域有以下几个方面可以进行深入研究:
(1)根据已知的TFBS模型,在基因组中预测TFBS的各种算法普遍存在假阳性率偏高的问题,降低预测中的假阳性是今后研究的重要目标。引起假阳性的一个主要原因是,基因组中存在很多与TFBS序列相同但没有转录因子结合功能的短串。随着人们对转录调控过程的了解的深入,可以考虑增加新的信息,比如将染色体结构信息,即核小体在基因组中的分布情况,或TFBS与转录起始位点之间距离的分布特征,以此作为先验信息,提高TFBS预测的准确率。另外, 转录调控通常需要多个转录因子的合作,它们的TFBS之间距离较近,组成相应的“顺式调控模块”(Cis-Regulatory Module,CRM),有CRM的区域比只有单个TFBS的区域更有可能是真正的转录调控区域。因此,预测CRM从而推断TFBS的分布,也能大大提高TFBS预测的准确率。
(2)目前的TFBS研究多为从DNA中提取信息、构造模型、设计算法,而忽略了转录因子本身能提供的信息,可以尝试将具有相同DNA结合域的转录因子家族作为一个整体进行研究。
(3)目前已有一些工作考虑转录因子与TFBS结合能的高低对下游基因转录的mRNA表达量的定量关系,而目前对TFBS模型的评介标准主要是“能否准确判别某DNA序列是否为TFBS”,对于模型能否准确描述转录因子与TFBS结合强度的定量关系缺乏评价,此类定量模型也有待开发。近年来,一些研究发现TFBS的丢失和获得在转录调控网络的进化中起重要作用,TFBS在调控网络进化中的作用机制还有待进一步揭示。我们相信实验技术的进步以及对转录调控机制的深入理解必将为TFBS的生物信息学研究注入新的生命力, 生物信息学与实验相互结合相互促进,人们对转录调控的认识将更加系统深入。
- 本文固定链接: https://maimengkong.com/kyjc/1701.html
- 转载请注明: : 萌小白 2024年3月29日 于 卖萌控的博客 发表
- 百度已收录
