肿瘤突变的类型丰富多样,包括点突变,杂合性缺失,缺失突变,倒位等等。如下图所示:
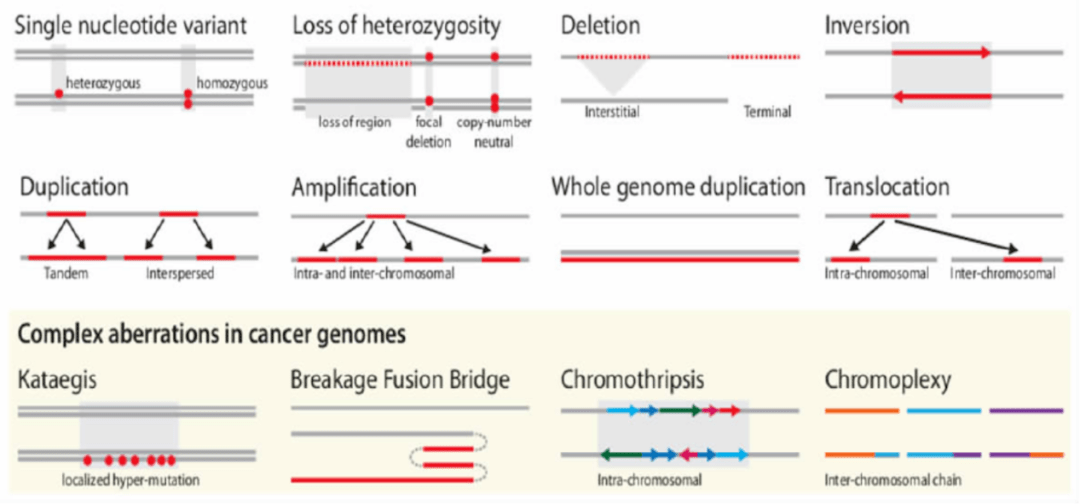
小师妹最近在学习肿瘤克隆进化的过程中,发现人类的点突变的分类与农学里的差异较大,为了厘清这里边的关系,小师妹觉得非常有必要做一个系统的梳理。
农学中的点突变,之前在Nature Genetics|从GWAS结果里挖掘候选基因方法微文中有描述,在GWAS定位的LD block中,将所有的SNP分为5类,再一次摘抄下来如下:
I:引起氨基酸编码变化和可变剪切位点(位于内含子start或者end处的GT或者AG)的显著关联SNP;
II:位于基因5’端的显著关联SNP(比如起始密码子ATG上游2Kb内的SNP);
III:不满足以上I和II标准的显著关联SNP(比如位于编码区,但是并未引起氨基酸变化的内含子区或者3’端非编码区);
IV:编码区(coding regions)外的显著关联SNP;
V:不显著关联的SNP。
这里边最常见的分类就是基因区同义突变和非同义突变,然后就是promoter区的SNP突变。另外,农学中取样的植物组织往往是叶片,也不存在体细胞这一说。医学相关研究中对突变的分类更加细致,什么是体细胞突变(somatic mutation)?|肿瘤全外一文也阐述了体细胞突变(somatic mutation)和胚系突变(germline mutation)的概念。在肿瘤全外中,一般使用安捷伦V6捕获探针,该探针虽然也有一部分的内含子和基因间区的探针,但是真正在分析的时候,重点还是关注基因外显子上的突变。根据突变的DNA序列长度,突变可分为点突变(point mutation)和片段突变(fragment mutation)。当然还有其他的分类方式,比如根据突变类型可将突变分为四类:替换(substitutions)、缺失(deletion)、插入(insertions)、倒位(inversion)。本期微信我们重点看点突变。点突变在农学研究中常被称为SNP(single nucleotide polymorphisms),即重点关注不同材料之间的碱基多态性。而在肿瘤全外研究中,点突变被称为SNV(single-nucleotide variants),即单碱基变异,重点在变异。
根据点突变对基因转录和蛋白质翻译产生的影响定性,可以把点突变分为同义突变(synonymous mutation)和非同义突变(non-synonymous mutation)二种。
同义突变,碱基替换不引起氨基酸改变,则称为又称为中性突变(neutral mutation)或沉默突变(silent mutation)。同义突变的原因是点突变发生在密码子的第三个核苷酸,由于遗传密码的简并性,这个突变后的密码子恰恰同突变前的密码子编码同一氨基酸,这样,突变不会改变蛋白质中的氨基酸序列。
引起编码氨基酸改变的点突变又称为非同义突变/替换。非同义突变,又进一步分为错义突变(missense mutation)和无义突变(nonsense mutation)。错义突变是指受到影响的密码子变成另一种新密码子,编码一个新的氨基酸,使氨基酸序列发生变化。错义突变大多发生在密码子的第一位或第二位核苷酸。无义突变是指一个编码氨基酸的密码子,在点突变后变成了一个终止密码子,使多肽合成提前中止,产生了缺失原有羧基端片段的缩短的肽链。在多数情况下,这种被截短的蛋白质片段是没有活性的。
另外,对于非同义突变后产生的影响(是否为有害突变),有不同的软件方法进行相关的评估。比如PolyPhen2,SIFT,MutationTaster以及PMut均可实现。
在肿瘤全外相关研究中,还有另外一种突变,即拷贝数突变/替换(somatic copy number alterations,SCNA),这类突变的定义和鉴定,小师妹下次为您揭晓。
转自:联川生物
- 本文固定链接: https://maimengkong.com/kyjc/1474.html
- 转载请注明: : 萌小白 2023年4月24日 于 卖萌控的博客 发表
- 百度已收录
