#单细胞测序的优势#
近年来,快速发展的高通量测序,已经广泛应用于生命科学的各个领域。传统的混合细胞测序(bulk cells sequencing)检测的是数以万计的细胞群体平均后的表达水平,无法反应每个细胞的真实情况,这使得局部/微小组织的功能研究有了极高的门槛。同时,细胞异质性也成了研究组织中特殊种类的细胞功能作用时无法跨越的鸿沟。
单细胞测序技术通过对单个细胞的RNA进行测序,精准得到单个细胞的基因结构与表达情况,从一定程度上解决了上述这些问题。
关于单细胞测序详细的内容可以看往期文章→#为什么要做单细胞测序 。
#单细胞转录组简述#
单细胞转录组是单细胞测序中最火的一个组学,通过检测单个细胞内所有mRNA总表达量,可以得到区别于普通转录组“分辨率”更高的数据。
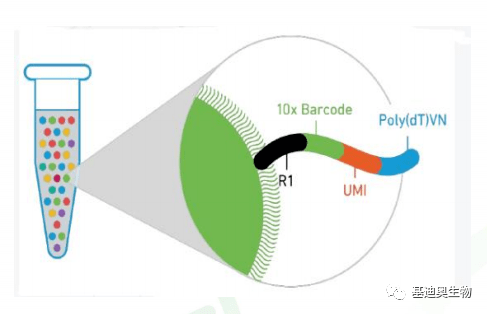
图1 微凝胶珠单细胞标记
测序原理是通过油包水GEM结构将每个细胞包裹分开,同时每个GEM会包裹一个微磁珠,根据微磁珠上不同的捕获序列为每一个细胞进行标记以便于后续的分析(Barcode:区分细胞;UMI:Unique molecular index,区分分子)。
单细胞转录组最重要的基础数据处理便是细胞聚类分群。其流程主要有四个内容:数据合并(批次效应矫正)、细胞过滤(除去“多胞”和破损、应激细胞数据)、亚群聚类(高变基因筛选、PCA降维、聚类分群)以及可视化。
检测细胞数的增加会导致数据分析复杂度的大大提高。因此,细胞分群的处理、分析和解读,对单细胞转录组测序就显得尤为重要。
关于数据合并中的批次效应,在之前#单细胞测序批次效应的产生和规避 #的文章中已经详细讲过。今天我们主要聊一聊细胞的降维与聚类。
#单细胞转录组中的降维与聚类#
在以往常规转录组测序中,由于实验生物学重复,同组内测序得到的数据是接近的,通过简单的线性降维PCA主成分分析法,即可得到想要的可视化结果(组内样本聚集,组间样本分离)。
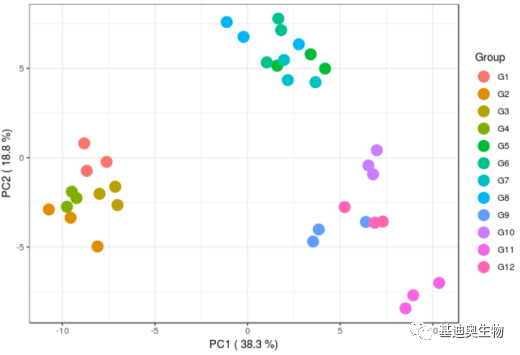
图2 PCA样本分析图
此时,我们想要的实验结果就是组内有极高的相似性,与其说是聚类,不如说其实只是通过降维图将本来就“应该在一起”的数据可视化,以此来判断是否有离群样本需要剔除,是否组间有差异可进行后续差异分析。而这就让人产生了一个误解,聚类就是基于降维图把“在一起”的点聚为一类。但是,实际上聚类和降维是两个独立的过程。
在单细胞转录组测序中,一个项目可以得到上万个细胞样本,基因数量通常也是几万个,这样高纬度的数据集不适合人直接进行阅读和解析细胞之间的功能差异。因此,我们需要通过聚类来简化细胞数量,通过降维来可视化数据。
细胞的降维与聚类就好比是“摘叶子”,而整份单细胞转录组的数据则好比是一株巨大的树。这棵树很高很大,看着很累,所以我们需要降维来让树更容易观测;这棵树的叶子(细胞)很多,所以我们需要聚类来将叶子进行分类。这样,我们就可以通过相对简单的方式来阅读这一份数据了。

#什么是降维?为何我们需要降维?#
降维就是复杂的高维数据信息简化为更容易阅读的低维数据信息,就像素描把三维的立体图形绘制在二维的平面上,而我们可以通过绘制而成的二维平面来了解到三维物体本身是什么,却不用我们到作者的绘画现场去看画的东西到底是什么样。我们需要通过对单细胞转录组数据降维,来获得更容易阅读的低维数据信息。
这里用三种摘叶子的方法来对应说明三种常见的可视化降维方法。
摇树(PCA)
让树叶以近乎垂直的方式掉落在地面(低纬度)。这种方法虽然“简单粗暴”,但可以使所有叶子快速地落在地面上。虽然会一定程度上丢失树叶生长高度的信息,但能够还原大部分真实的生长位置信息。
这种方法其实就与线性降维PCA的原理相似,将高维特征收敛到低维上。而低维的各个维度(正交特征)也被称为主成分PC,是在原有高维特征的基础上重新构造出来的低维特征。
关于PCA的原理算法,详细的介绍→#你是否真的了解PCA? 中有讲述。
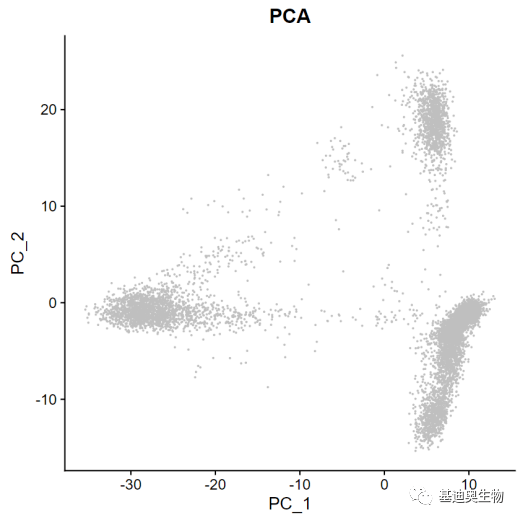
图3 PCA降维后可视化
搭梯子摘(tSNE)
以一个随机的位置作为起始点,架个梯子爬上去摘,每次摘一部分就得爬到树下放叶子,然后再爬上梯子继续摘,那整个过程就会变得十分漫长,耗时耗力,但优点是能有效地还原一部分树生长高度的位置信息,并能将生长在一起的叶子放在同一摞。
这种方法与之对应的降维法便是非线性降维tSNE算法,由于tSNE的计算过程较为复杂且耗时长,往往需要先通过PCA进行预降维处理。PCA降维过的数据再进行tSNE降维(降至二维或三维)实现可视化。而tSNE算法其实主要就是通过将临近的相似点距离收缩,较远的(非相似)点距离增大将各集群边界分开。
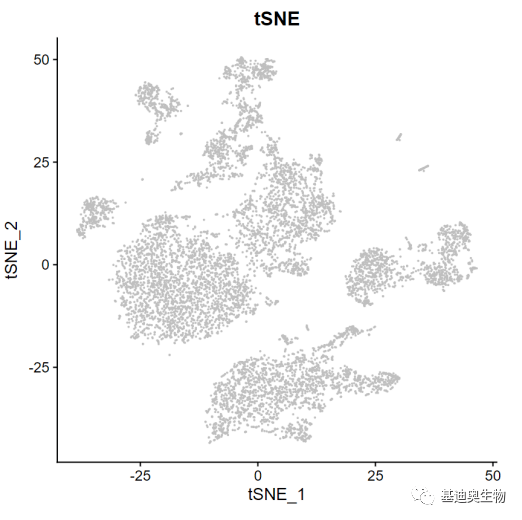
图4 tSNE降维后可视化
请专人搭梯子摘(UMAP)
第三种,请一个经验老道的园艺工帮忙摘。(上一种的假设是一个没有经验的人的摘叶子方式)由于园艺工经验老道,虽然摘叶子的方法是差不多的,但是速度要快上许多。在摘的过程中,还能够下意识地有选择性地把相近相似的叶子摘下来,同时,还能够将摘下来地每一摞叶子都简单地打个包压缩空间,并把不同时候摘的相似叶子都放在一块。
最后的这种方法便是现在炙手可热的可视化工具UMAP。在可视化质量方面,UMAP算法与tSNE具有竞争优势。由于对嵌入维度没有计算限制,使得其在高维数据分析中不仅可以比tSNE有更快的计算处理速度(对PCA预降维的需求度降低),还能更有效地保留了更多全局结构,可以通过可视化结果看出具有相关性集群大多相近。
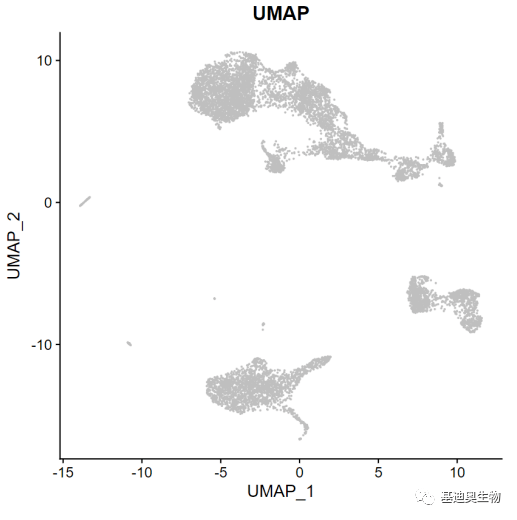
图5 UMAP降维后可视化
#什么是聚类?为何我们需要聚类?#
聚类就是归类,按照一定的标准来进行归类就是聚类。我们得到降维后的数据后,就能根据聚类算法对细胞进行分群聚类,通过可视化图来呈现更直观的效果。具体的详细介绍可以看#你知道scRNA-Seq细胞聚类的算法原理嘛? 这里不过多赘述了。
这里,我们还是以树叶比作细胞来方便大家理解。我们把不同年份的叶子比作不同种类的细胞来进行分类。由于上述摘叶子(降维)的方式不同,我们最终呈现出来分叶子(聚类)的可视化情况也会有所不同。
相同或相近年份的叶子会在一定程度上相似(如颜色、大小、叶脉纹路等),就好比我们同类细胞或者相近来源的细胞,他们某些基因表达量是相近的。将不同年份的叶子颜色、大小、叶脉纹路等的差异理解为不同细胞的基因的表达情况和表达量的差异。然后我们人为的设定一个分辨率阈值,将表达量差异小于一定范围的细胞归为一类。
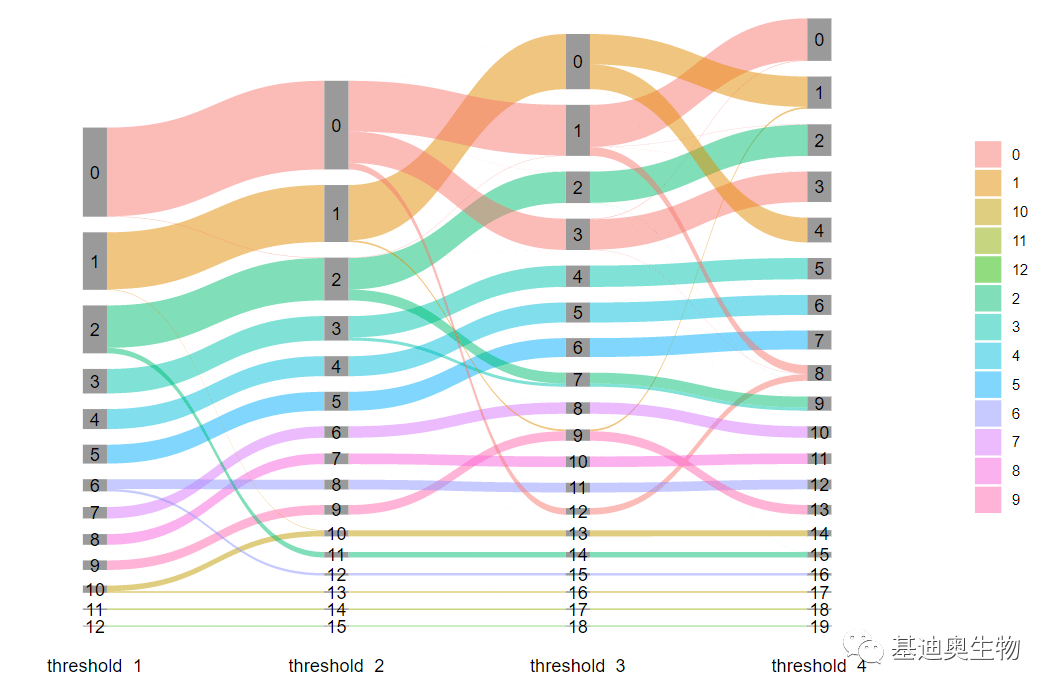
图6 不同的分辨率下的聚类分群
#降维和聚类的关系#
降维和聚类是同时进行的两个独立过程,将两者结果合并才是我们最终得到的可视化结果。但这个合并过程中一定会存在矛盾的地方,这也是影响了可视化图中分群边界与聚集远近的最主要原因。
PCA是经典的线性降维方式,每一个维度代表了数据的一个特征信息,用越多的特征信息去描述一个数据就越接近真实;取用的维度越少,信息的丢失量越多。而在可视化中,我们只能使用“最有特色”的两个或三个主成分去描述数据,这就使得大量的细胞无法被良好地区分开,最终呈现的结果就是不同类型的细胞类型之间的边界不明显 (如图7)。
此外,还需要提及的一点是,并不是所有聚类分出的同种细胞都会降维在一起。在可视化图中相同的细胞类型可能也会有较远的分布。例如图7中,tSNE图的cluster 4和UMAP图的cluster 7。
tSNE的算法使得高维的相近距离在低维观测的时候有一定几率变成较远距离,可视化图上就会看起来不像是同一簇细胞。而UMAP虽然能比tSNE更好地将相似细胞簇聚集,不同类细胞簇分开,但当UMAP的计算距离和聚类的计算距离差异较大时,可视化结果就也会显示同一个细胞聚类被分了开来。
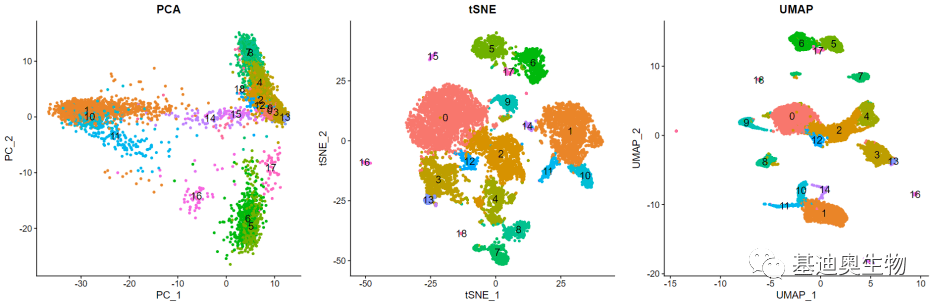
图7 tSNE可视化图中同类细胞来源不同的簇
#小结#
在单细胞转录组测序中,降维和聚类其实是两个独立的过程。会让人容易混淆是因为降维和聚类经常体现出一定的一致性,本质原因是他们都是使用的高维数据特征。
用前面分树叶的例子来说,聚类是把一类相同特征的叶子归为一类,这一类叶子具有相同的叶脉、颜色等等特征;而为什么他们具有这些特征呢,就是因为这些叶子生长在相近的地方(数据在高维空间的距离较近),容易受到相同的光照、水运、激素等的影响,而这些叶子在落下来时,也更容易落在相近的位置(降维时聚在一起)。正是因此,降维和聚类具有相当的一致性。
此外,像前面提及的tSNE,虽然可以保证降维在一个集群内的细胞之间会具有相似性,但各个集群之间的相似性无法得到保证,集群之间的远近也无法代表集群间的相似性。而UMAP也会有一定概率出现同种细胞在可视化图中呈现不同簇的情况。
- 本文固定链接: https://maimengkong.com/kyjc/1145.html
- 转载请注明: : 萌小白 2022年8月18日 于 卖萌控的博客 发表
- 百度已收录
