增强子作为基因组上的顺式作用元件,在调控网络中发挥重要作用。随着研究的不断深入,科学家提出了超级增强子super-enhancer的概念,将基因组上富集了增强子的区域定义为超级增强子。
类似于将富集CpG位点的区域定义为CpG岛,这种将富集某种元件的区域单独定义并进行研究的思想在生物学领域非常常见,体现了不同元件功能层次的多样性,由单个位点到一组位点,这种功能研究的层次性也对应了生命体复杂的调控机制。
和增强子的研究类似,对于超级增强子,也是研究其在不同生物学过程,疾病发生发展等过程中的调控作用。SEdb是一个综合性的超级增强子数据库,文章发表在Nucleic Acids Research上,链接如下
https://academic.oup.com/nar/article/47/D1/D235/5146197
该数据库的网址如下
http://www.licpathway.net/sedb/
采用了H3K27ac这种组蛋白修饰作为增强子区的标记,利用从ENCODE, RoadMap, GEO等公共数据库中下载的H3K27ac chip_seq数据,首选采用MACS识别增强子区,然后采用ROSE这款软件来识别超级增强子区。
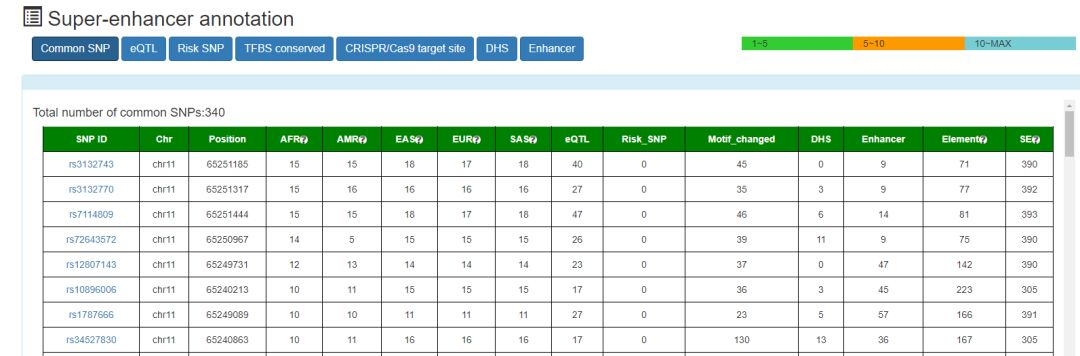
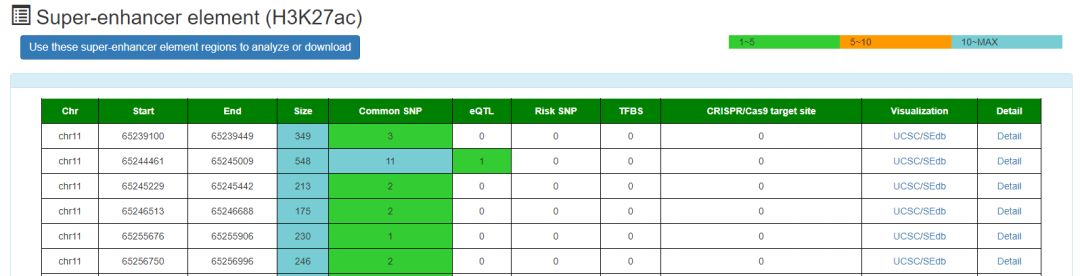
对于识别到的超级增强子区,利用bedtools进行下列注释
-
SNP位点
从dbSNP数据库下载SNP位点,利用1000G的数据对SNP位点进行连锁不平衡分析,同时从GWAS Catalog和GWASdb下载疾病相关的risk SNP位点信息,注释在超级增强子区域内存在的各种SNP位点。 -
DHS
从UCSC和ENCODE下载DNase酶超敏区域信息,进行注释 -
Enhancers
从ENCODE和FANTOM5下载增强子区域信息,进行注释 -
TFBS
从UCSC下载转录因子结合位点的信息,进行注释 -
CRISPR/Case9 target sites
从UCSC下载基因编辑位点信息,进行注释 -
eQTLs
从GTEx, HaploReg和PancanQTL数据库中下载eQTL-gene关系对,进行注释 -
motif change
从TRANSFAC和JASPAR数据库下载转录因子的motif信息,利用atSNP这个R包计算SNP位点对motif的影响
除了对超级增强子进行注释外,还通过6种不同策略对超级增强子的靶基因进行预测,所有的注释信息和靶基因预测结果都可以通过数据库检索进行查询和浏览。
考虑到超级增强子的特异性,所有的数据都是以单个样本的形式进行分析和存储的,示意如下
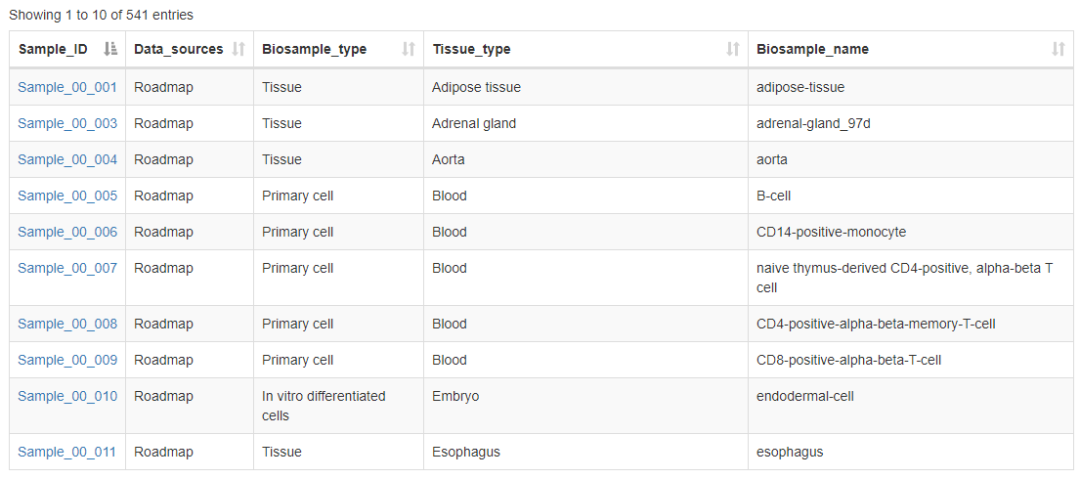
点击样本编号可以查看单个样本的超级增强子信息,首先是样本的基本信息和超级增强子在不同染色体上的分布,示意如下

接下来是超级增强子的列表,示意如下
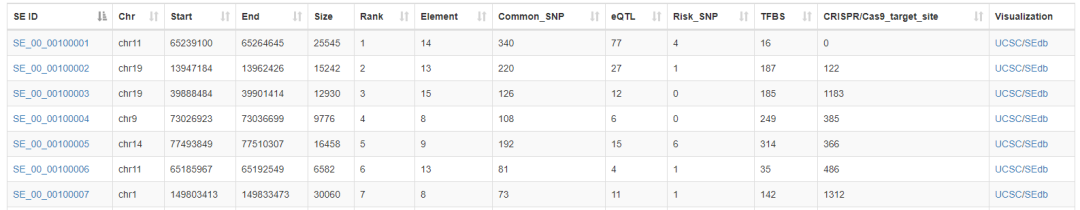
点击第一列的ID,可以查看详细信息
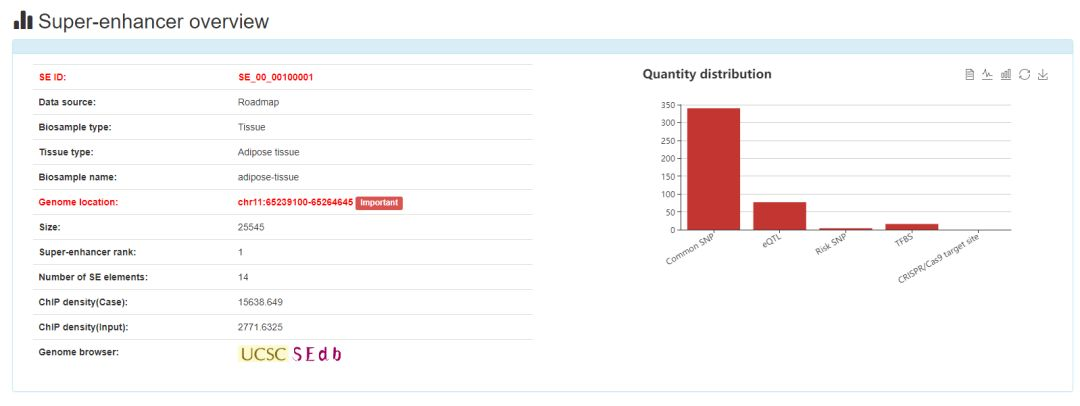
1. Overview
2. Annotation
3. target gene
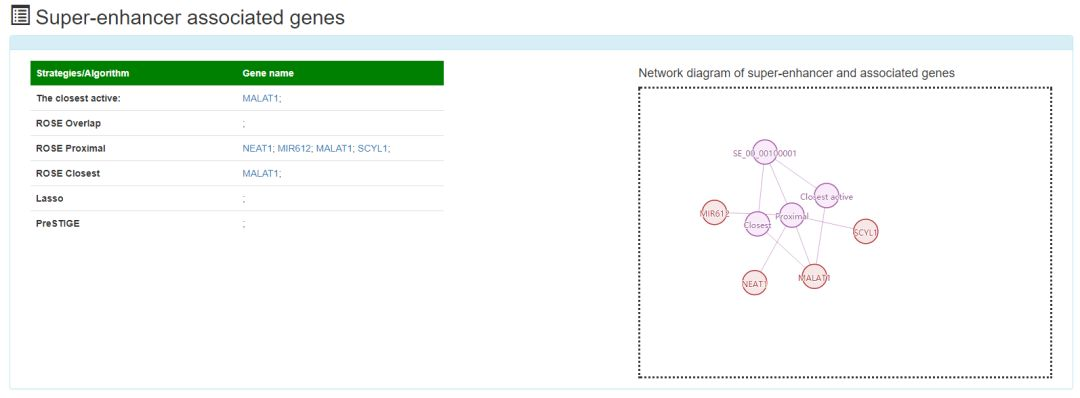
4. enhancer
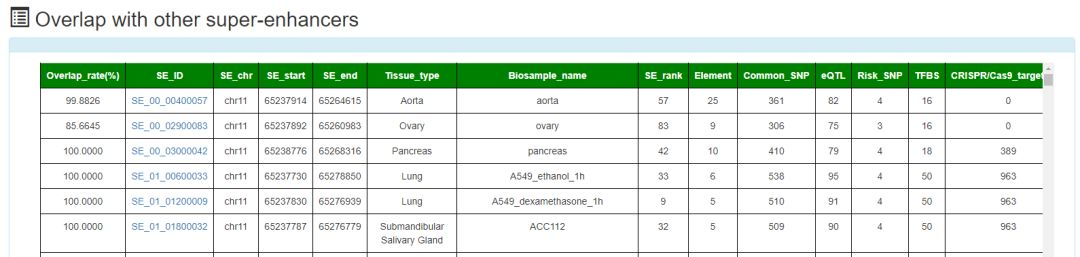
5. overlap with super-hancers
除了基本的检索和浏览功能外,该数据库还提供了很多的在线工具,数据也是开放下载的,更多的用法请参考官网的帮助文档。
·end·
- 本文固定链接: https://maimengkong.com/moreshare/1383.html
- 转载请注明: : 萌小白 2023年3月1日 于 卖萌控的博客 发表
- 百度已收录
