7个常用数据库拿下肿瘤3区文章,你还犹豫什么
最常用的生信分析在线数据库+最简单的生信分析思路=2021年10月发表在Frontiers in genetics(IF:4.772)的肺腺癌生信分析文章——《 RAB11FIP1: An Indicator for Tumor Immune Microenvironment and Prognosis of Lung Adenocarcinoma from a Comprehensive Analysis of Bioinformatics》。
文章共8图4表,以TCGA_LUAD数据集为分析对象,使用Oncomine、HPA、LinkedOmics、cBioPortal、STRING、TIMER与GEPIA等7个高频使用的生信分析数据库,以RAB11FIP1基因为主变量,免疫浸润分析为重点,纯生信分析的手段分析了肺腺癌中RAB11FIP1的异常低表达与肿瘤的预后等关联。今天这篇推文带大家快速领会文献分析套路,5分钟看完全文,手把手教大家一小时学会全文每张图表的完整复现~
题目:RAB11FIP1: An Indicator for Tumor Immune Microenvironment and Prognosis of Lung Adenocarcinoma from a Comprehensive Analysis of Bioinformatics
期刊信息

1.材料与方法
1)疾病:肺腺癌
2)物种:人类
3)数据来源:TCGA LUAD(n=515)
2.图标结果及复现
2.1 使用工具
1)仙桃学术生信工具(https://www.xiantao.love/products)
2)HPA数据库( https://www.proteinatlas.org)
3)LinkedOmics数据库( http://www.linkedomics.org/admin.php)
4)cBioPortal数据库(http://www.cbioportal.org/)
5)STRING数据库(https://cn.string-db.org/)
6)TIMER数据库( https://cibersortx.stanford.edu/)
7)GEPIA数据库(http://gepia.cancer-pku.cn/)
2.2复现任务
Figure 1| RAB11FIP1 expression levels in LUAD.
RAB11FIP1在肺腺癌中的表达水平
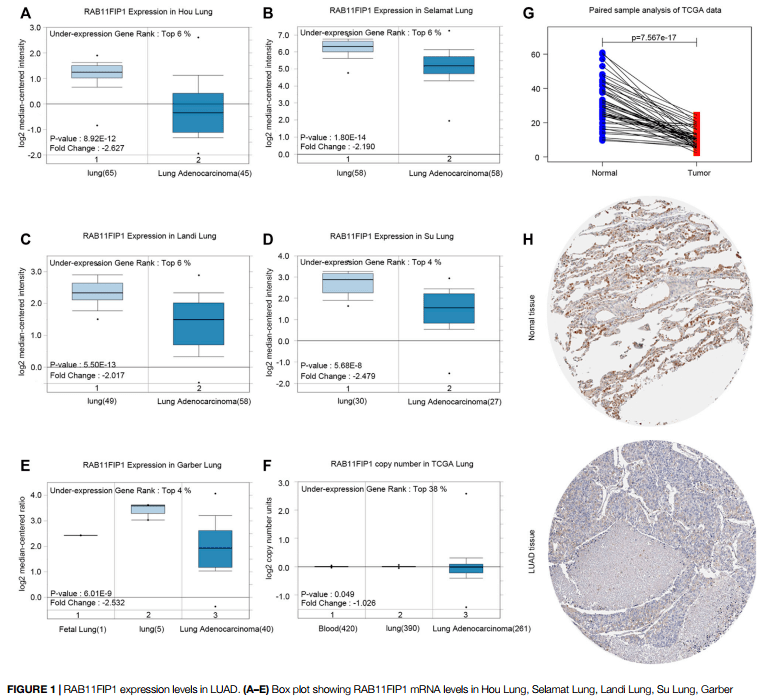
Figure 2| Tranion of RAB11FIP1 stratified by gender, age, and other criteria in a subgroup of patients with lung cancer.
RAB11FIP1在肺腺癌不同亚组中转录水平
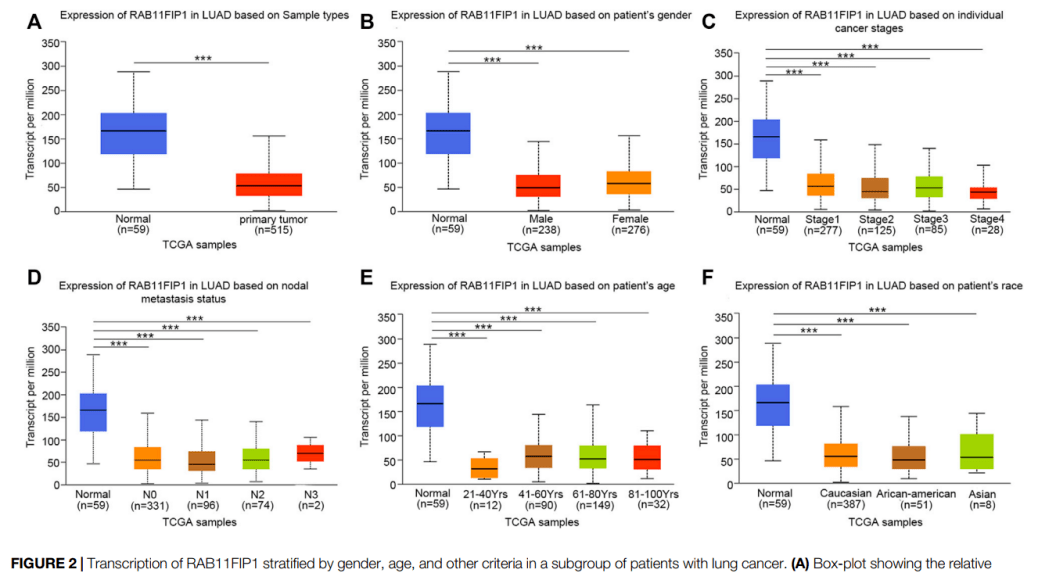
Table 1| Association between RAB11FIP1 expression and clinicopathologic parameters by Logistic regression.
Logistic回归分析RAB11FIP1表达水平与临床病理参数关联
Figure 3| Relationship between RAB11FIP1 and prognosis in LUAD.
RAB11FIP1表达水平与LUAD预后关联
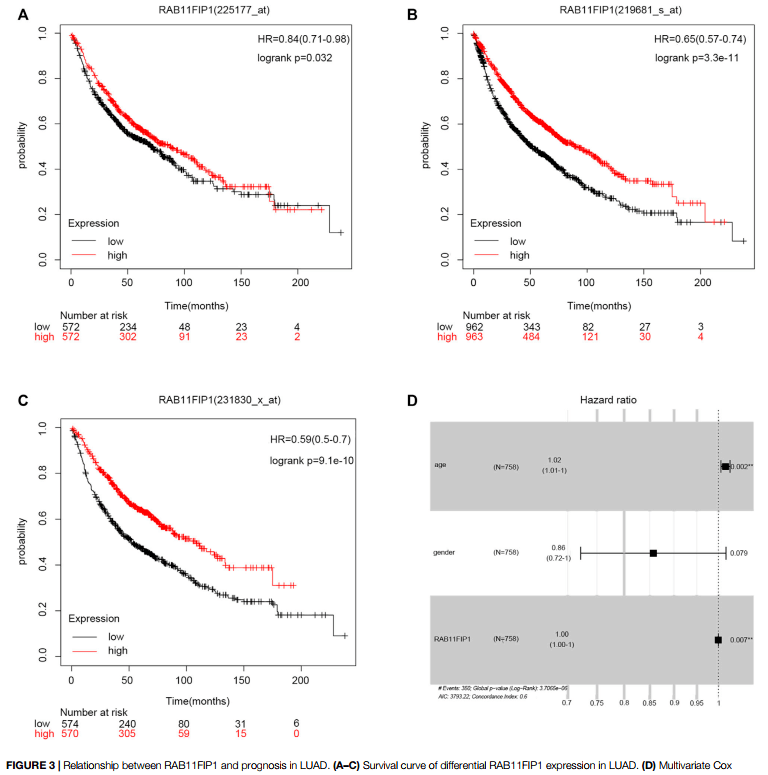
Table 2| Univariate and multivariate Cox analysis of clinicopathological parameters and OS in patients with LUAD.
LUAD患者临床病理参数单、多因素Cox分析表格
Figure 4 | GSEA with GO term and KEGG pathway.
GO条目与KEGG通路GSEA富集分析
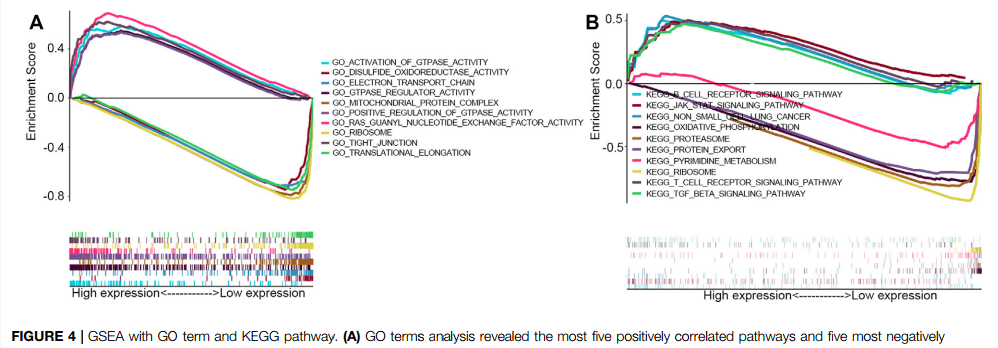
Figure 5|Correlation between gene differential expression and RAB11FIP1 in LUAD中基因差异表达与RAB11FIP1的相关性
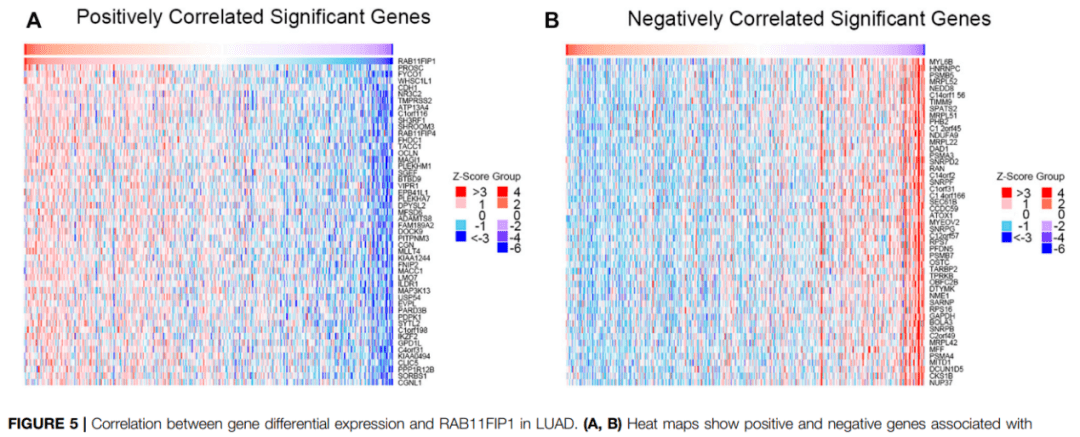
Figure6|GO annotation and KEGG pathway in lung cancer. The GO annotation and KEGG pathway of RAB11FIP1 co-expressed genes in lung tissue were analyzed by GSEA.
GSEA分析LUAD中RAB11FIP1共表达基因的GO注释和KEGG通路
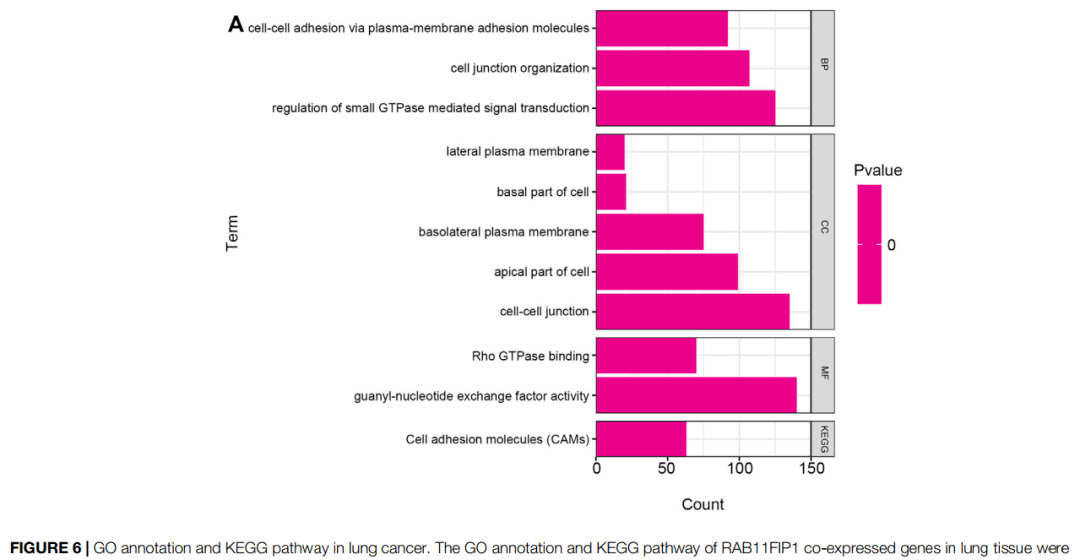
Figure7| Top two modules from the PPI network.
PPI网络的Top2 核心模块
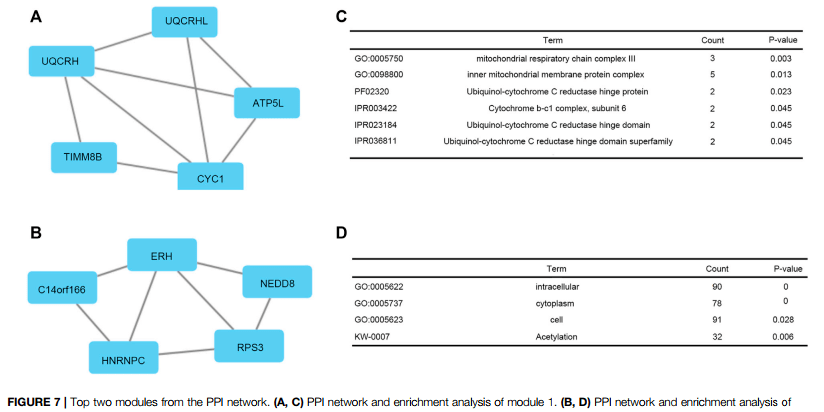
Figure 8 | Relationship between expression of RAB11FIP1 and level of immune invasion in LUAD
LUAD中RAB11FIP1表达与免疫浸润水平的关系
Table3|Correlation analysis between RAB11FIP1 and marker genes of immune cells in TIMER
TIMER数据库分析RAB11FIP1基因与免疫细胞标志基因相关性
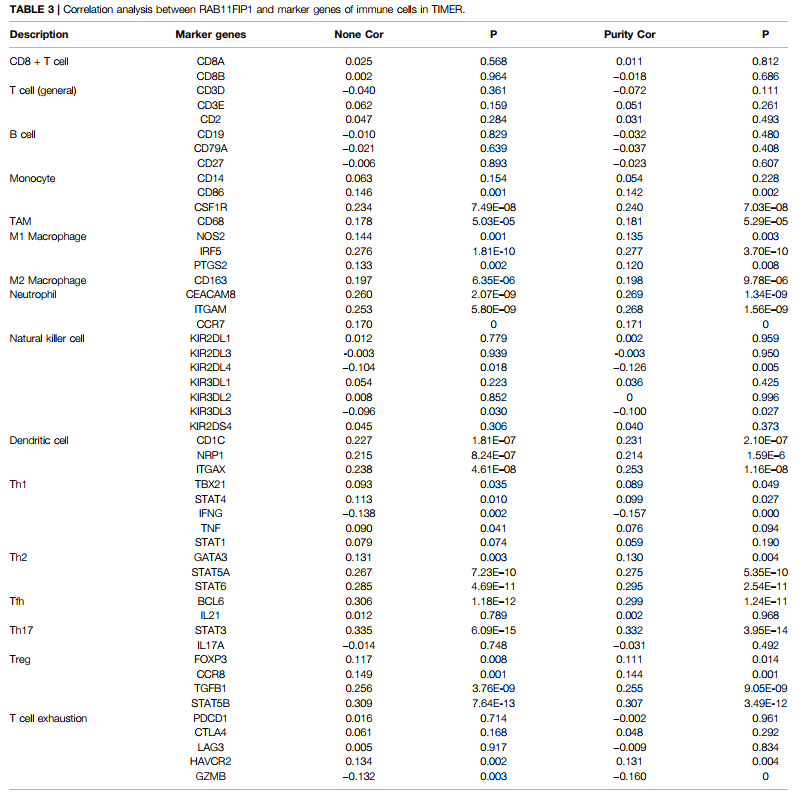
Table 4|Correlation analysis between RAB11FIP1 and TIICs marker genes in GEPIA.
GEPIA数据库中RAB11FIP1和TIICs标记基因的相关性分析
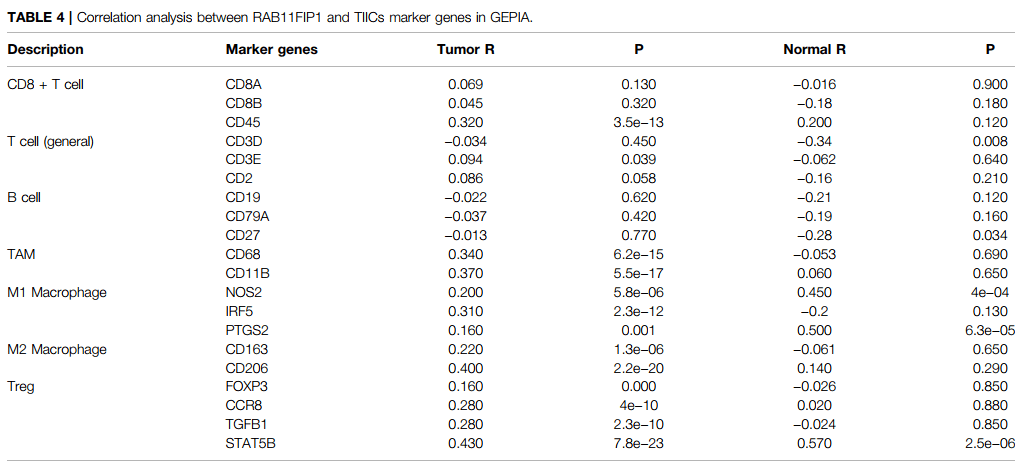
3.复现步骤
3.1 Fig.1 RAB11FIP1在肺腺癌中的表达水平
1)图1 A-F为Oncomine 4.5 数据库中RAB11FIP1在不同数据集中正常肺与肺腺癌中表达比较箱式图,由于Oncomine数据库在2022年1月17日已停止使用,此图无法复现;我们可以看到主变量RAB11FIP1分子在肿瘤组织中表达显著降低
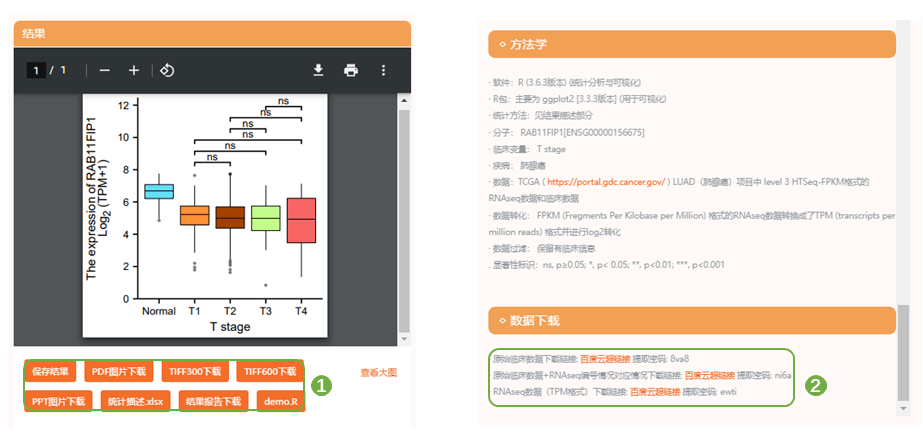
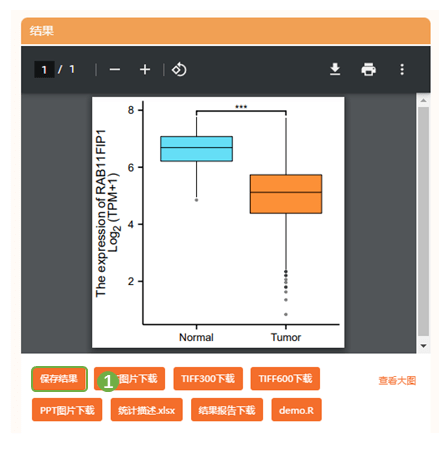
2)图1 G为TCGA数据库中LUAD数据集配对样本表达比较,同样也是肿瘤组织中RAB11FIP1表达显著减少。进入“仙桃学术—生信工具——表达差异——[云]配对样本”,选择“TCGA_LUAD”数据集,右侧的“基因”处输入“RAB11FIP1”,选择对应的分子信息,提交分析,得到原文同款TCGA_LUAD数据集配对样本RAB11FIP1 表达差异比较连线图。图例、图性色彩及透明度等均可实现个性化调整,实现满意的效果后保存结果,下载图片,查看统计描述与分析报告,可看到TCGA_LUAD数据集配对样本中RAB11FIP1在肺腺癌组中表达显著降低
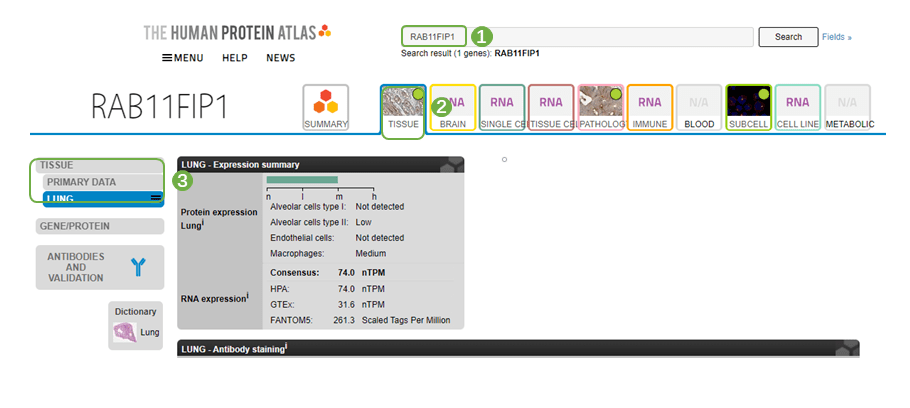
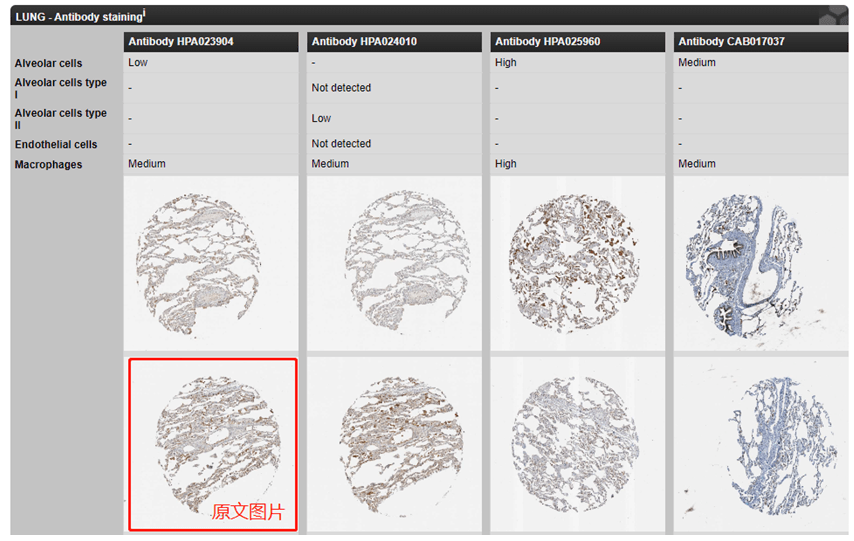
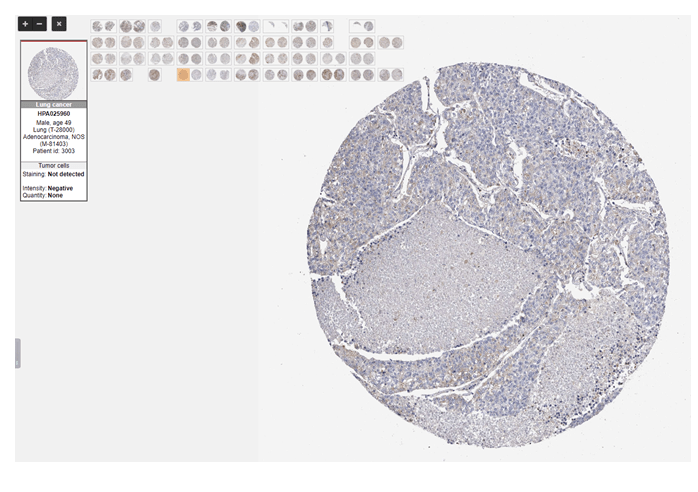
3)图1 H为HPA数据库中RAB11FIP1蛋白在正常肺组织(上)与肺腺癌组织(下)中免疫组化结果图片,HPA数据库在生信文章中作为表达差异实验辅助结果出现的频率非常高,数据库简洁易上手,首先进入HPA数据库官网(https://www.proteinatlas.org),输入目标分子“RAB11FIP1”,选择“TISSUE”,左侧选择组织类型为“LUNG”,即可看到原文中的同款正常肺组织IHC图片,点击进入详情页面,保存图片即可;回到起始页,输入目标分子“RAB11FIP1”,选择“PATHOLOGY”,左侧“PATHOLOGY”选择”LUAD“,就看到了原文中同款肺腺癌染色图片
3.2 Fig.2RAB11FIP1在肺腺癌不同亚组中的转录水平
图2 A-F为使用UALCAN数据库绘制的TCGA数据集中RAB11FIP1分子在正常肺组织与肺腺癌组织、性别亚组、肿瘤T分期亚组、N分期亚组、年龄亚组与人种亚组的表达分析
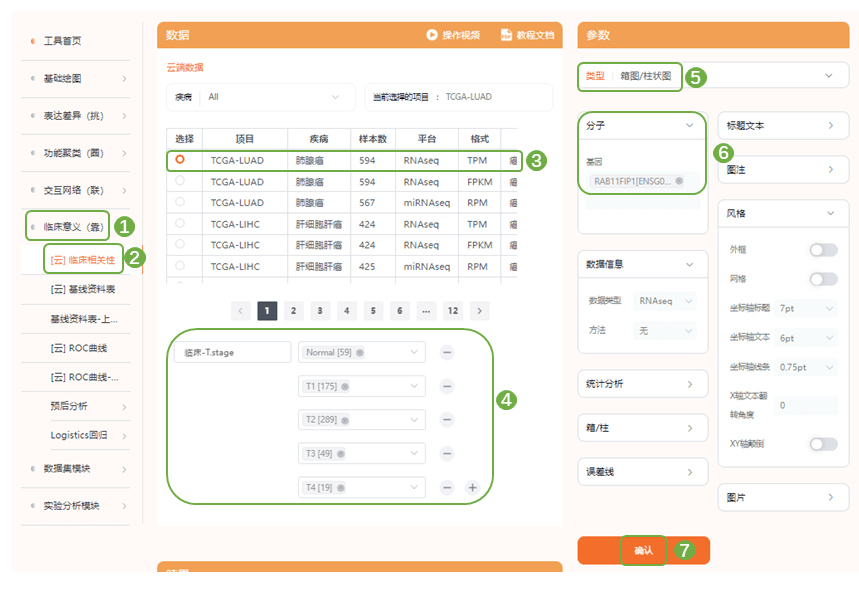
进入“临床意义(靠)——[云]临床相关性“,选择TCGA_LUAD数据集,每次选择一个亚组类型,依次选定亚组内因子顺序,右侧”分子“中选择“RAB11FIP1”,提交分析,得到原文同款亚组比较箱式图;依次完成目标亚组分析,仙桃学术页面拉下,还非常非常友善提供了整理好的TCGA表达谱文件下载链接噢~
3.3 Table1Logistic回归分析RAB11FIP1表达水平与临床病理参数关联
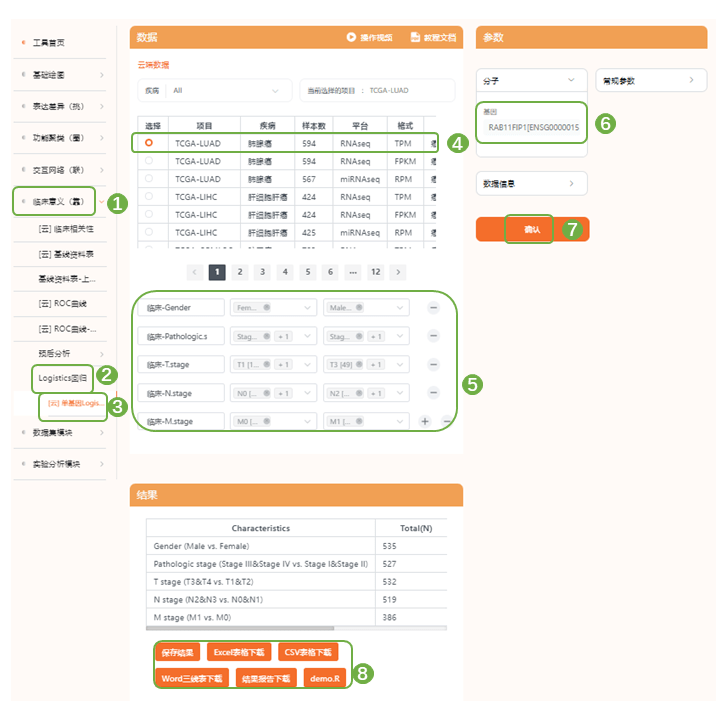
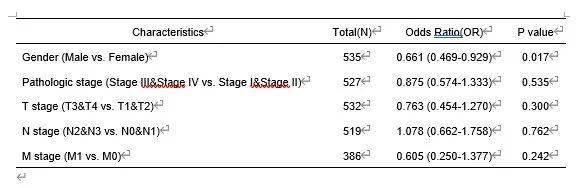
Table1为RAB11FIP1单基因logistic回归分析,即以分类临床变量逐个作为logistic回归方差的因变量,RAB11FIP1基因为自变量,使用logistic回归的方法分析目标基因对于临床变量的推断效能,评估单个基因和临床变量之间的关系
进入“临床意义(靠)——Logistic回归——[云]单基因logistic回归“,选择数据集,右侧为目标分析的单个基因,选择原文中的临床变量:gender、stage(pathologic stage)、tumor size(T stage)、lymph node metastasis(N stage)与distant metastasis(M stage),提交分析,保存结果并下载word三线表
3.4 Fig.3 RAB11FIP1表达水平与LUAD预后关联 & LUAD患者临床病理参数单、多因素Cox分析表格
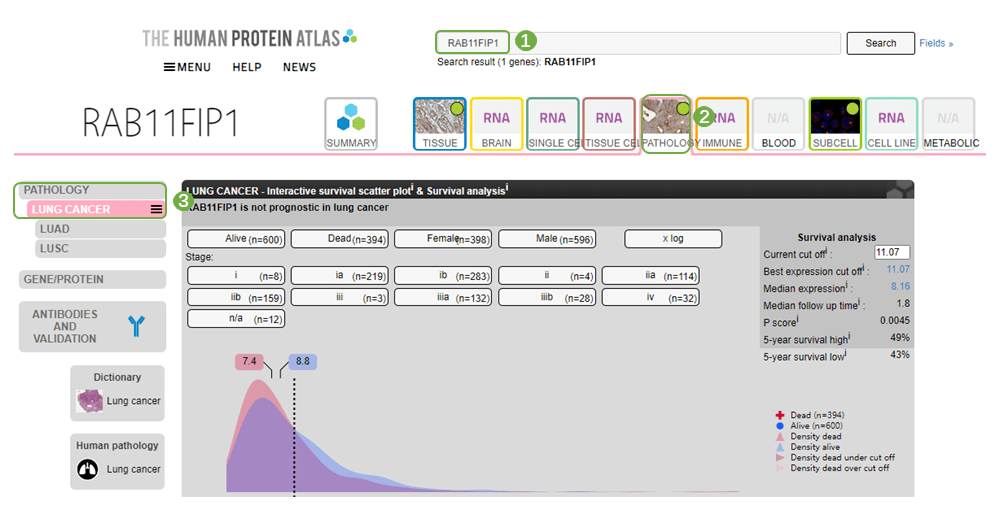
1)图3 A-C为RAB11FIP1分子(3种探针)的表达水平与LUAD生存预后KM曲线图,可以看到RAB11FIP1低表达组预后更差。进入“临床意义(靠)——预后分析——[云]KM曲线图,选择数据集与主变量分子,提交分析,即可得到根据分子表达水平分为高、低组的KM曲线图,图中的“+”表示删失数据,KM曲线下降越缓慢则预后越好
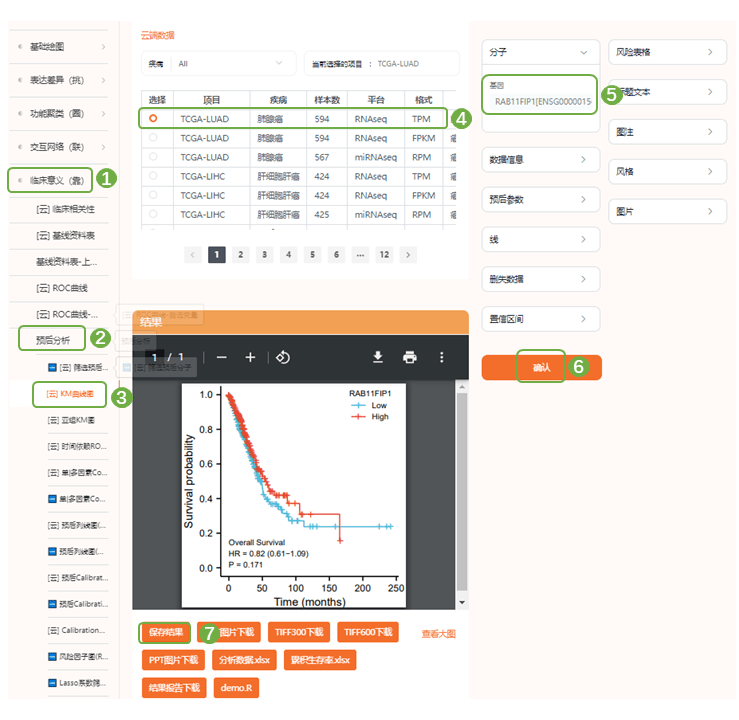
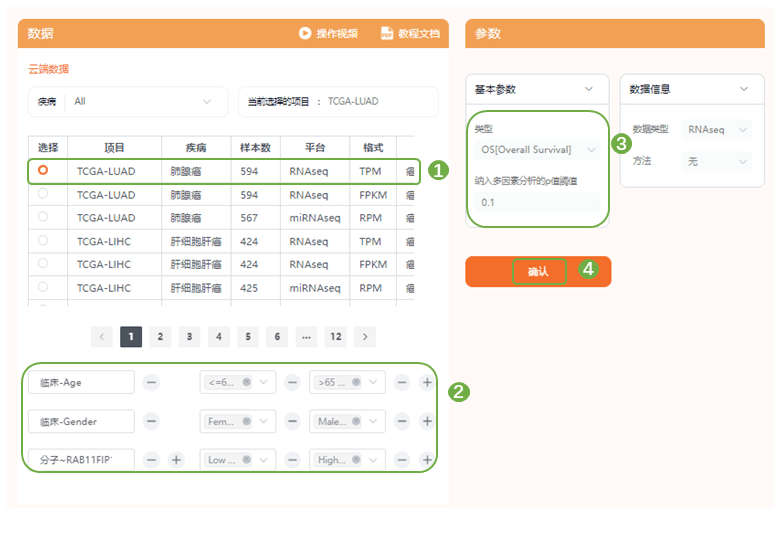
2)图3 D为age、gender与RAB11FIP1 三个变量的多因素Cox回归分析森林图,进入“临床意义(靠)——预后分析——[云]单|多因素Cox回归分析”,选择数据集,以及进行单因素Cox回归分析的变量,右侧选择预后类型参数,仙桃学术可选OS(overall survival,总体生存期)、DSS(disease specific survival,疾病特异性生存期)与PFI(progress free interval,无进展生存期),提交分析,保存结果,并下载excel表格,这里的word三线表即为原文中的Table2啦~

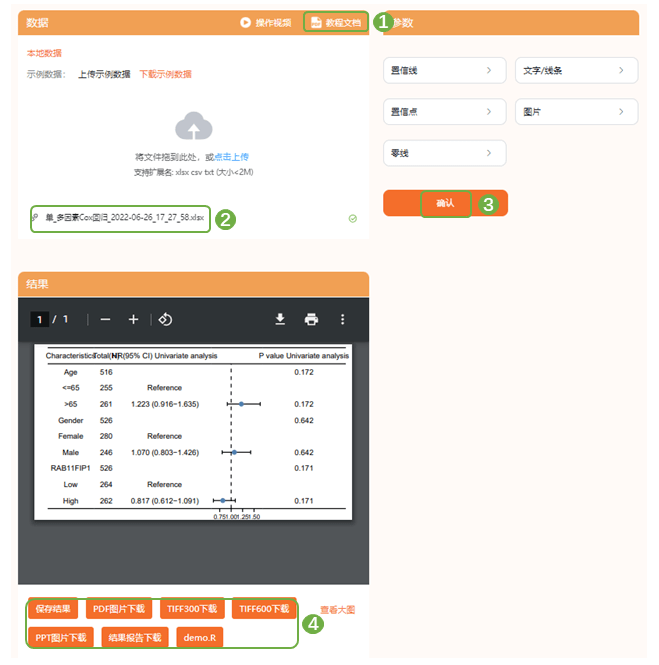
3)进入“基础绘图——森林图“,查看教程文档,按文档要求格式准备上传文件,这里直接上传刚才下载的excel表格即可,提交分析,保存结果,同样地,图片的颜色、标题等均可在右侧功能区进行修改



3 . 5 Fig.4 GO条目与KEGG通路GSEA富集分析
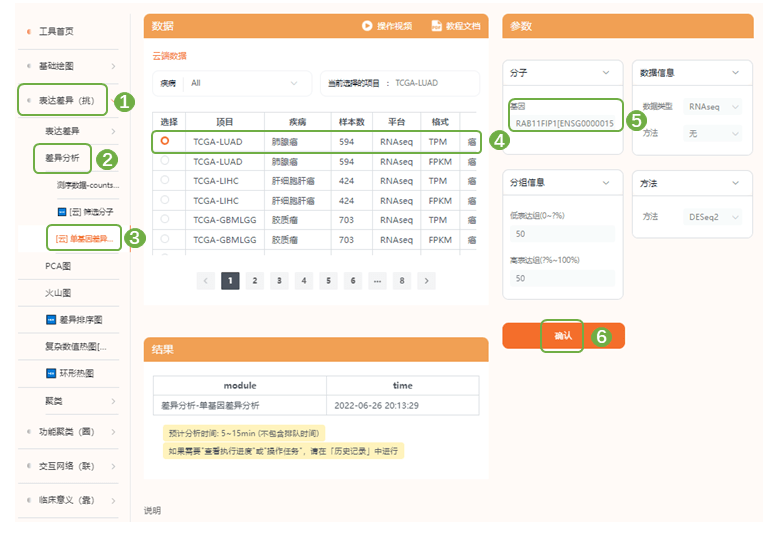
1) 图4为以主变量RAB11FIP1基因表达水平分为2组,对相关基因进行GO及KEGG富集分析 。进入“表达差异(挑)——差异分析——[云]单基因差异分析”,选择TCGA_LUAD数据集,右侧“分子”栏目下选择目标基因,提交分析,稍等1-2min分析完成后在历史记录中下载分析结果表格,保留gene name与logFC 2列数据即可,另存为新文件
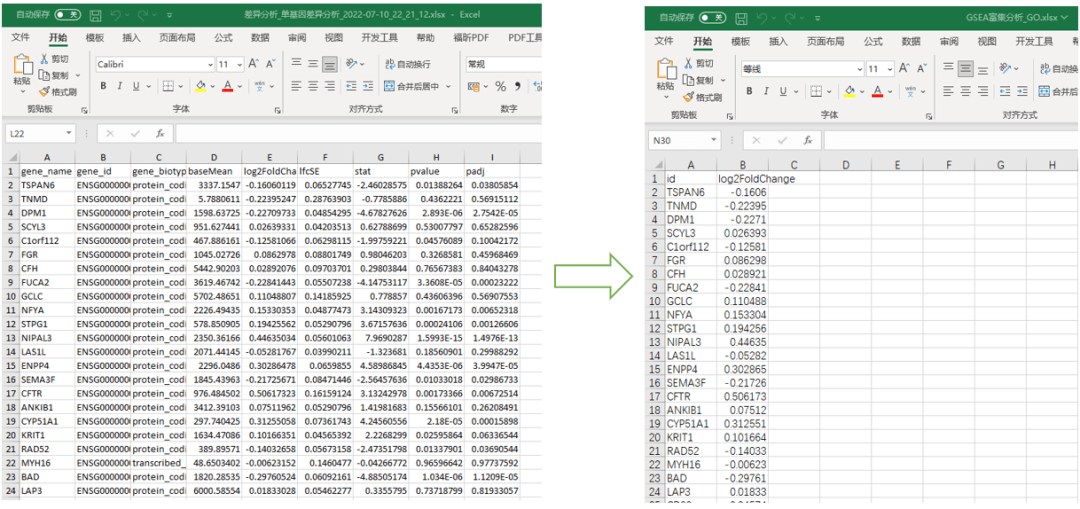
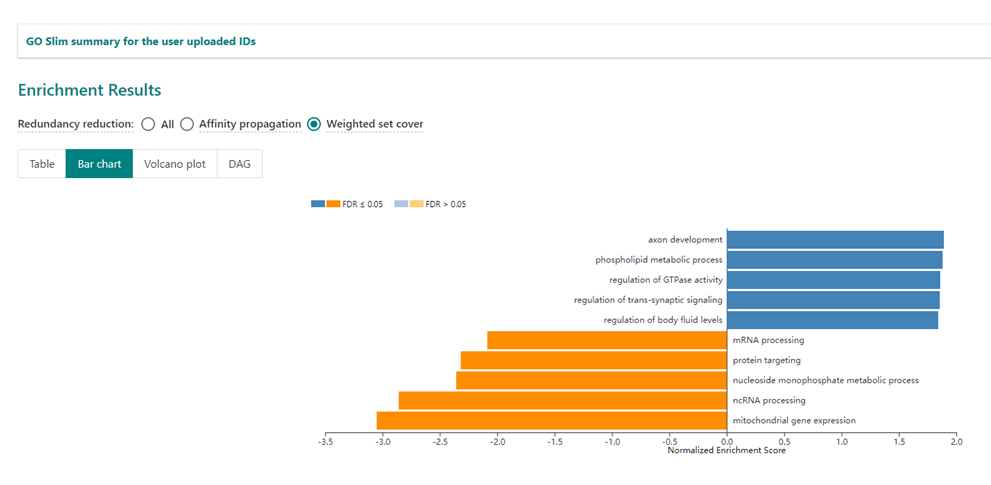
2)进入“功能聚类(圈)——GSEA富集——GSEA分析”,上传整理好的文件,右侧参考数据集依次选择”c5.all.v7.2.symbols.gmt[Gene ontology]”与”c2.cp,v7.2.symbols.gmt[Curated]”,进行GO与KEGG富集分析,依然在“历史记录”中下载分析结果表格,根据p.adj <0.05 & | NES | >1筛选合格的富集结果,可以选择根据NES排序展示top5结果,也可以选择展示自己感兴趣的表型相关富集结果
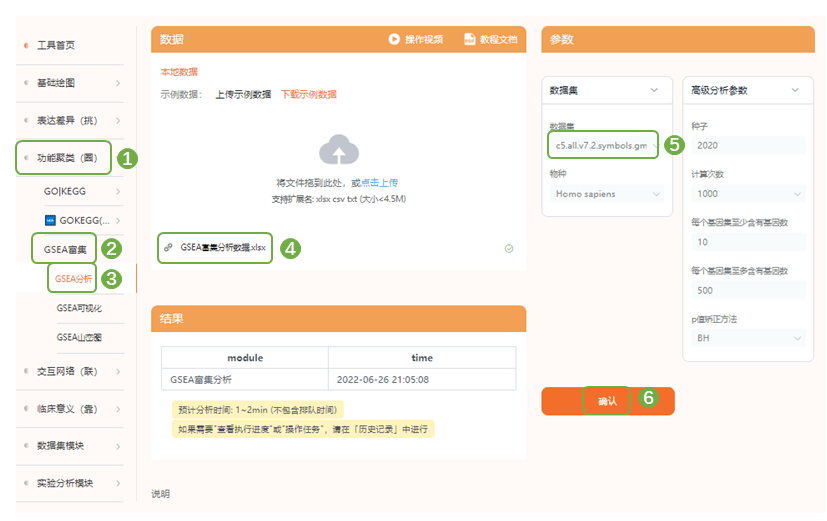
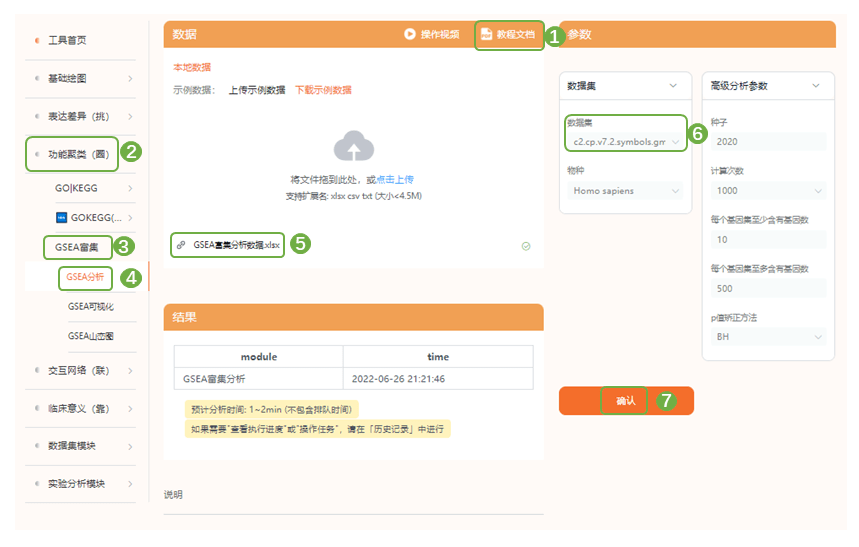
介绍:MSigDB数据库将所有的基因集划分为9个大类:H: hallmark gene sets,包含由多个已知的基因集构成的其他类别的基因集;C1: positional gene sets,包含人类每条染色体上的不同cytoband区域对应的基因集合;C2: curated gene sets包含已知数据库,文献和专家支持的基因集信息,包含2个二级分类:CGP(chemical and genetic pertuibations)、CP(Canonical pathways),其中KEGG、REACTOME属于CP子集;C3: regulatory target gene sets,包含转录因子或microRNA调控潜在靶点的基因集,包括MIR(microRNA targets)和TFT (tranion factor targets)2个子集;C4: computational gene sets:通过挖掘大量肿瘤相关芯片数据定义的基因集,包括CGN(cancer gene neighborhoods)和CM(cancer modules)2个子集;C5: ontology gene sets,包含Gene Ontology(GO)、Human Phenotype Ontology(HPO)对应的基因集合;C6: oncogenic signature gene sets,包含在肿瘤中异常的细胞通路特征基因集;C7: Immunologic signature gene sets, 代表免疫系统内细胞状态和干扰的基因集;C8: cell type signature gene sets,包含人类组织单细胞测序研究中确定的细胞类型的聚类标记基因集。
2)进入“功能聚类(圈)——GSEA富集——GSEA(可视化)”,选择相应的云端数据集,右侧“基因集ID”处支持手动输入感兴趣的信号通路,”绘制部分“有3种选择,上部分为富集评分情况,如果NES为正,则峰出现在左侧(头部富集)(高表达组富集),NES为负反之;中部分每一根竖线代表基因集中一个分子,分子根据给定的值排序,排序后单独提取当前基因集中定义的分子,分子位置即为图中所示;下部分根据上传数据中分子给定的值归一化后进行可视化,一般无需关注下部分;提交分析,目前仙桃学术支持同时绘制5个基因集,伙伴们也可以选择单独展示自己感兴趣的1-2个基因集,可以显示富集分数,pvalue等参数噢~
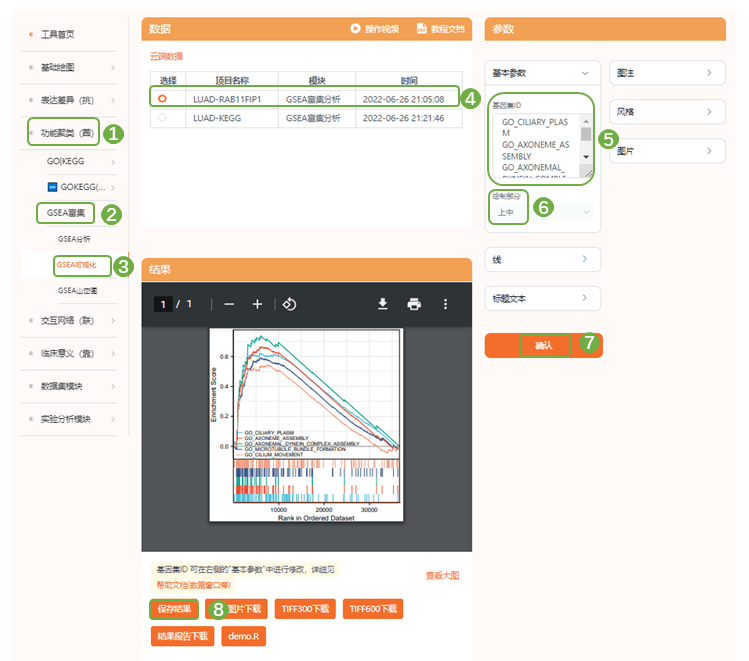
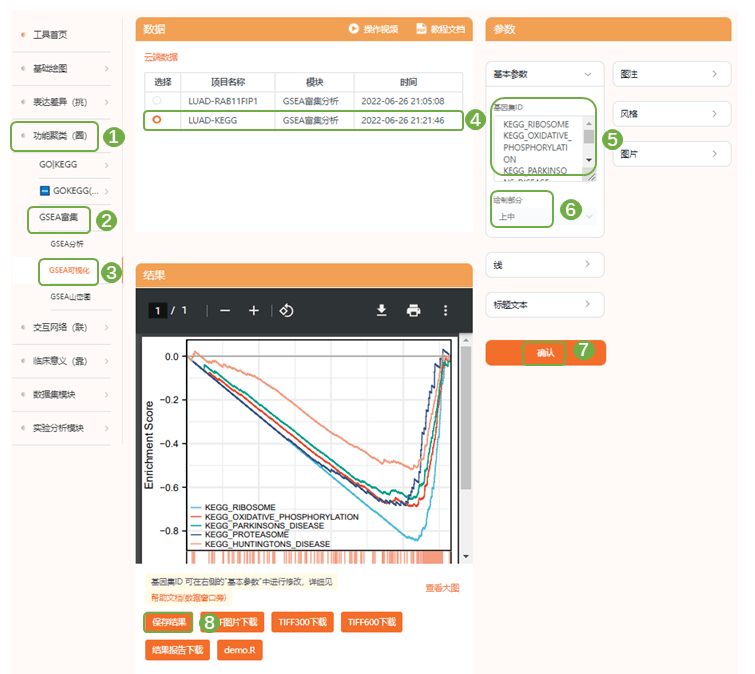
3.6 Fig.5 LUAD中基因差异表达与RAB11FIP1的相关性
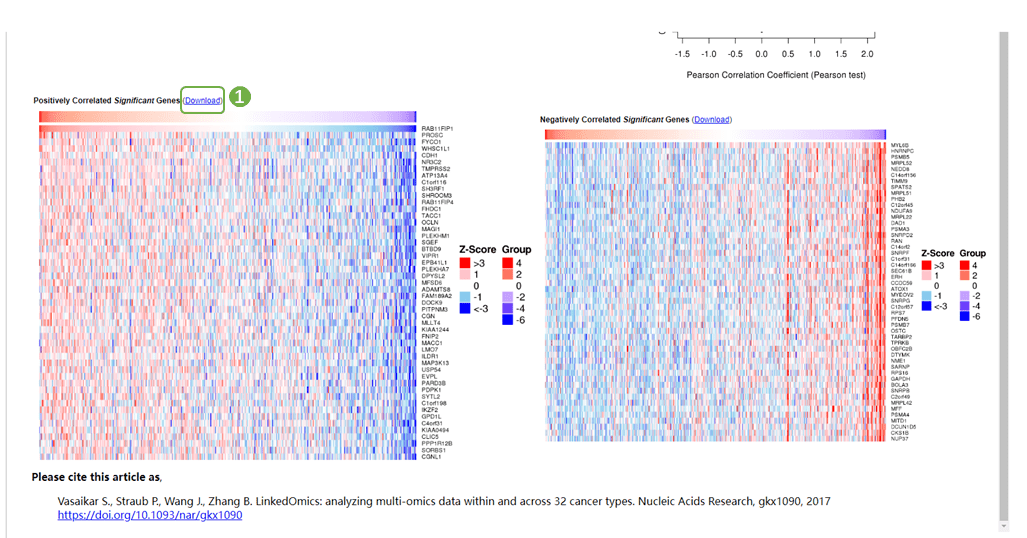
图5为LinkedOmics数据库中LUAD数据集中差异表达基因与RAB11FIP1分子的相关性热图。
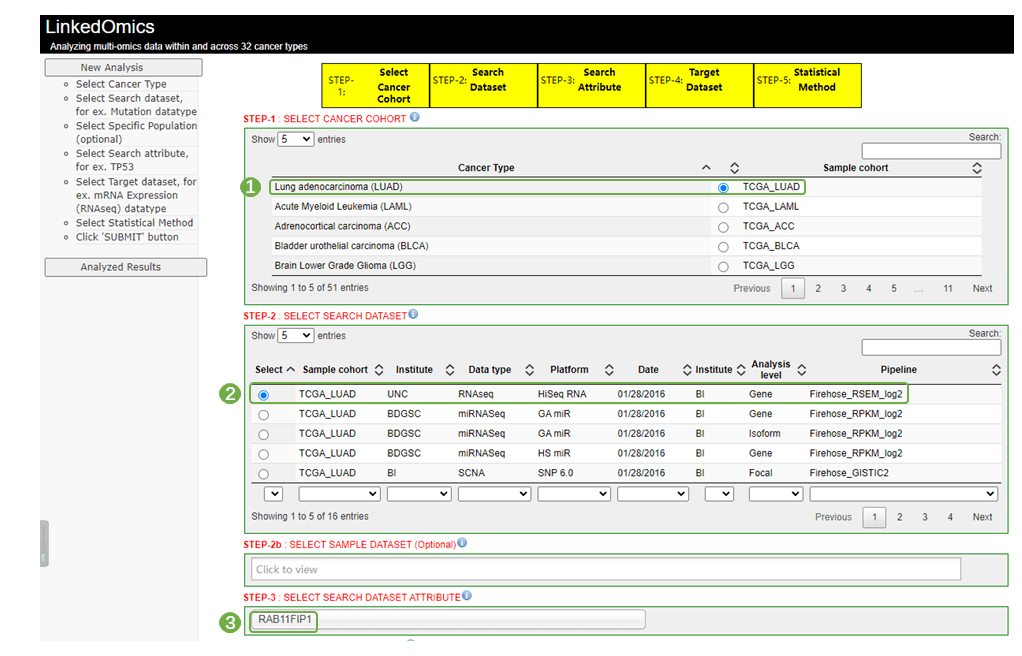
1)进入LinkedOmics数据库(http://www.linkedomics.org/admin.php),LinkedOmics数据库是挖掘TCGA数据的一款经典好用数据库,功能非常强大,数据库使用前使用普通的邮箱注册即可,无需教育邮箱。根据官网首页的step提示,首先STEP1选择分析数据集,在第6页找到LUAD数据集,点击前边的小圆圈选中;STEP2选择数据类型,HiSeq RNA(高通量RNA-seq),STEP 2b可进一步选择样本信息,为可选项,这里我们选择纳入TCGA_LUAD所有样本;STEP3选择目标基因,输入RAB11FIP1;STEP4中依然选择HiSeq RNA数据集,STEP5选择相关性分析方法,这里与原文一致,选择Pearson相关性分析

2)待分析完成后,选择分析结果,在“LinkedFinder”模块下查看分析结果,网站提供了分析结果下载的选项,大家可以根据需要下载分析结果~分析结果下拉,即可看到与原文同款的差异基因与RAB11FIP1表达相关性分析热图
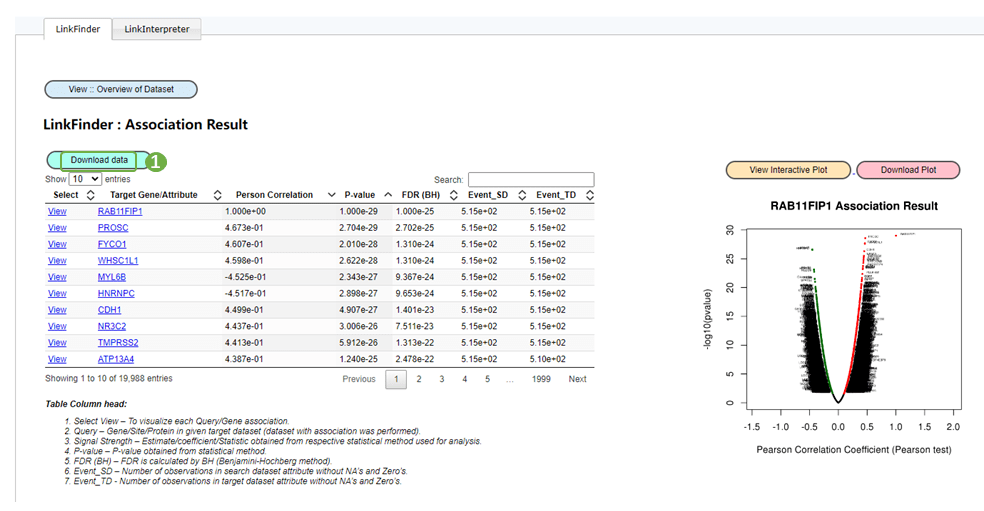
3.7 Fig.6 GSEA分析LUAD中RAB11FIP1共表达基因的GO注释和KEGG通路
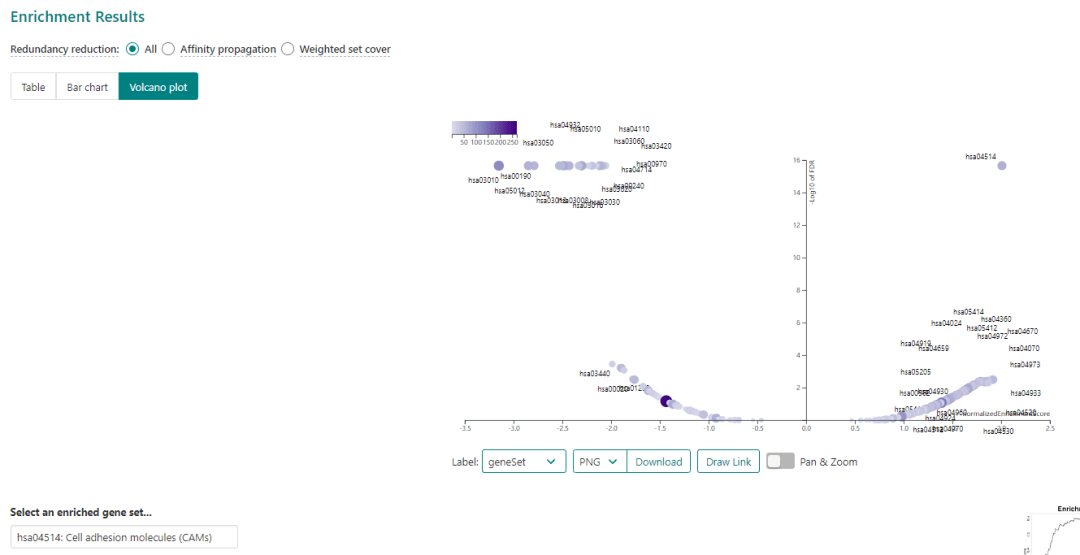
接下来使用相关性基因分析结果进行了GSEA富集分析,进入“LinkInterpreter”模块,“Select Tool”处选择“Gene Set Enrichment Analysis(GSEA)”,“Enrichment Analysis”处可以分别选择GO分析的BP、MF、CC以及KEGG分析,网站提供了Table、条形图与火山图3种展示形式,点击“Download”可下载结果
3.8 Fig.7 PPI网络的Top2 核心模块
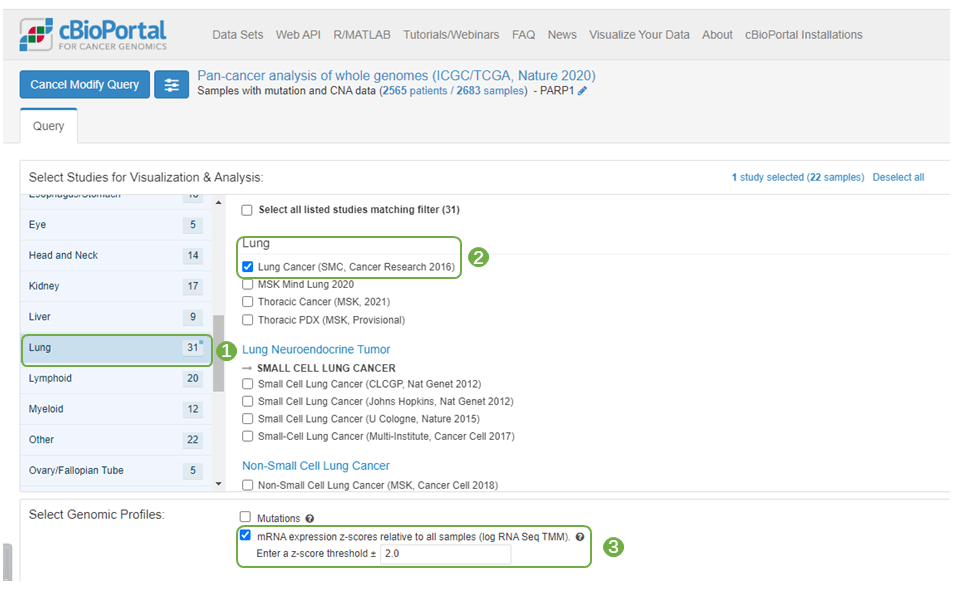
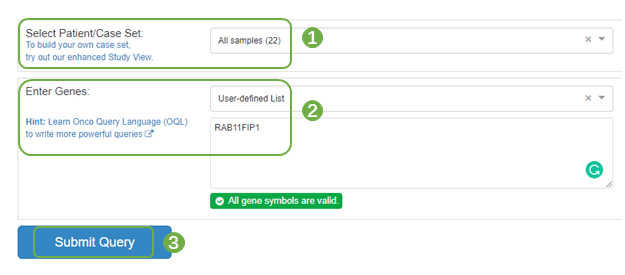
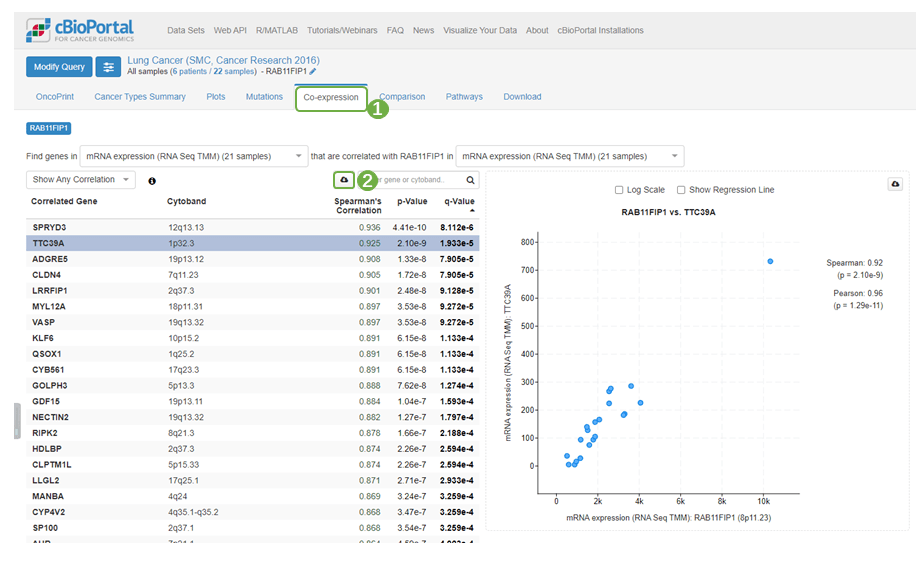
1)图7为RAB11FIP1共表达基因的蛋白互作网络分析。首先进入cBioPortal数据库(http://www.cbioportal.org/)分析LUAD数据集中RAB11FIP1的共表达基因,左侧组织类型选择“Lung“,右侧选择”Lung Cancer“,“mRNA Expression”表达数据,z-score阈值设置默认即可;纳入所有样本,输入目标基因“RAB11FIP1”,提交分析
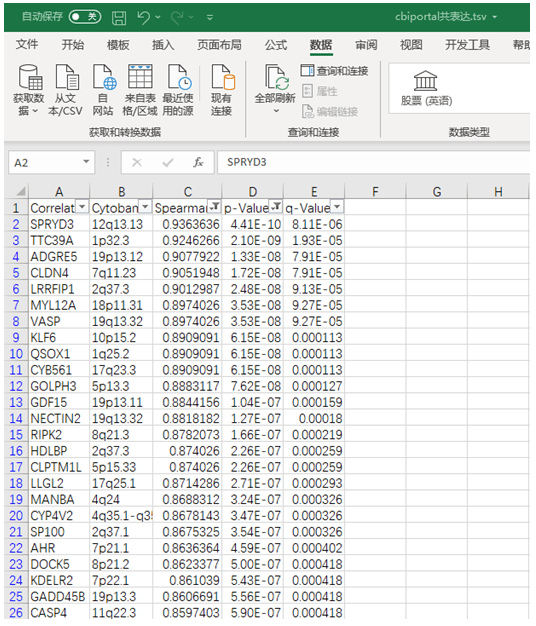
2)上方的导航栏选择“Co-expression”,查看共表达分析结果,点击云朵样图标下载数据,筛选p.value < 0.05的基因,在18407条记录中共有5047个基因符合筛选条件,基因数目还是过多,文章中对于PPI网络分析的具体参数缺乏相关阈值,这里我们再选择Spearman's Correlation > 0.8的基因再进行一次筛选,可以看到共有137个基因满足条件
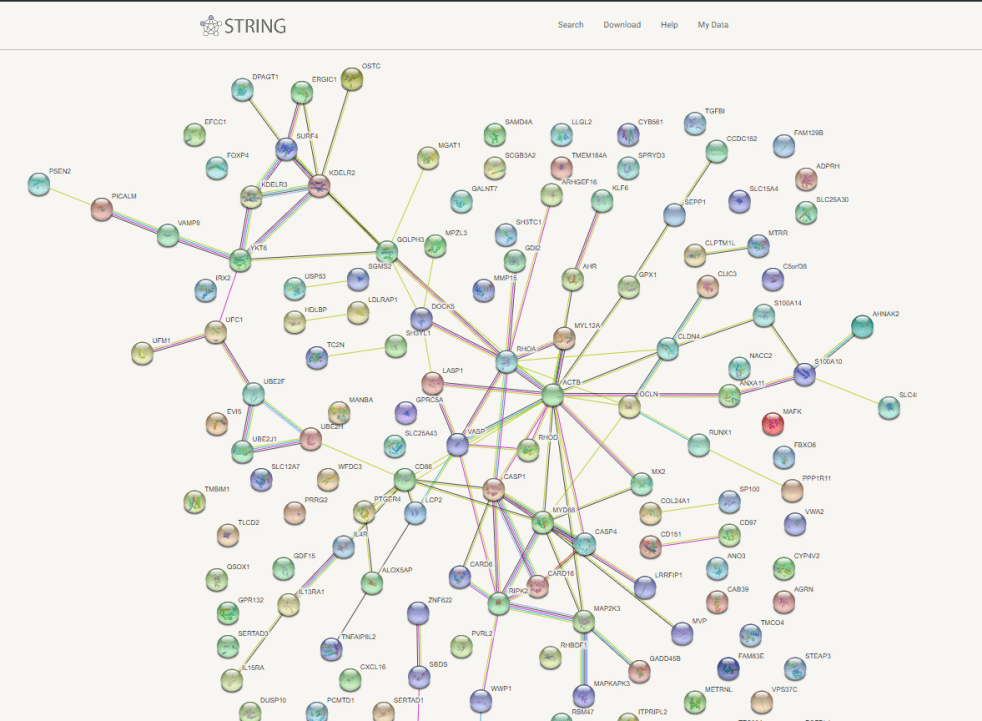
3)进入STRING数据库(https://cn.string-db.org/),点击官网首页“SEARCH”进入分析界面,左侧功能栏选择“Multiple proteins”,粘贴刚才筛选后的共表达基因,物种选择“Homo sapiens”,提交分析,之后点击“Continue”,得到蛋白-蛋白互作网络图,将网络导出至Cytoscape软件

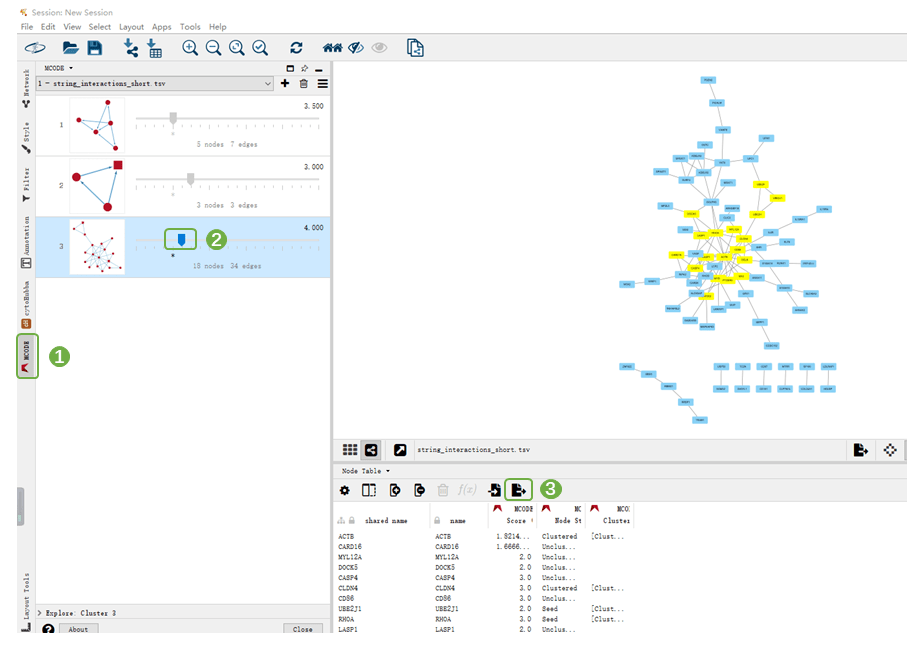
5)图7C、D为模块基因的富集分析结果表格,下载模块分析表格,“selected“中筛选”TRUE“,复制18个核心基因,进入仙桃学术”功能聚类(圈)——GO|KEGG——GO|KEGG富集分析“,右侧分子列表粘贴刚才的18个核心基因,富集分析条目可选择GO+KEGG、全部GO条目或KEGG等类型,提交分析,保存结果,下载结果三线表,即得到发表级分析结果
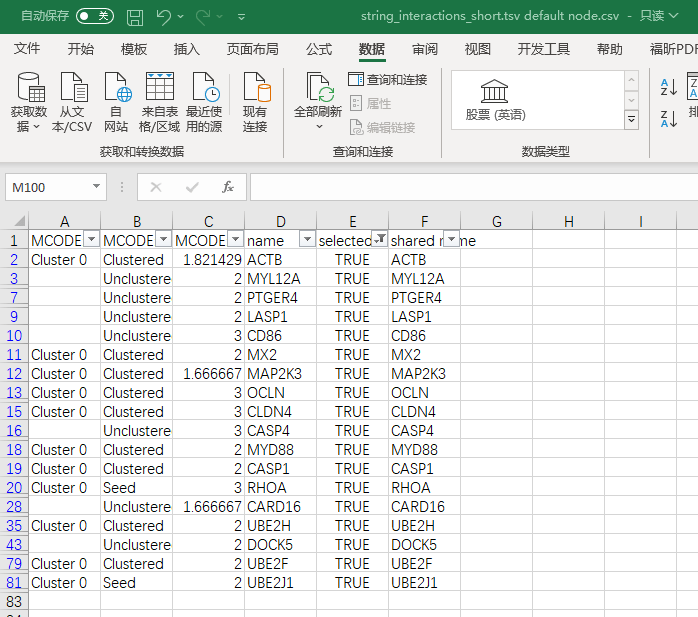
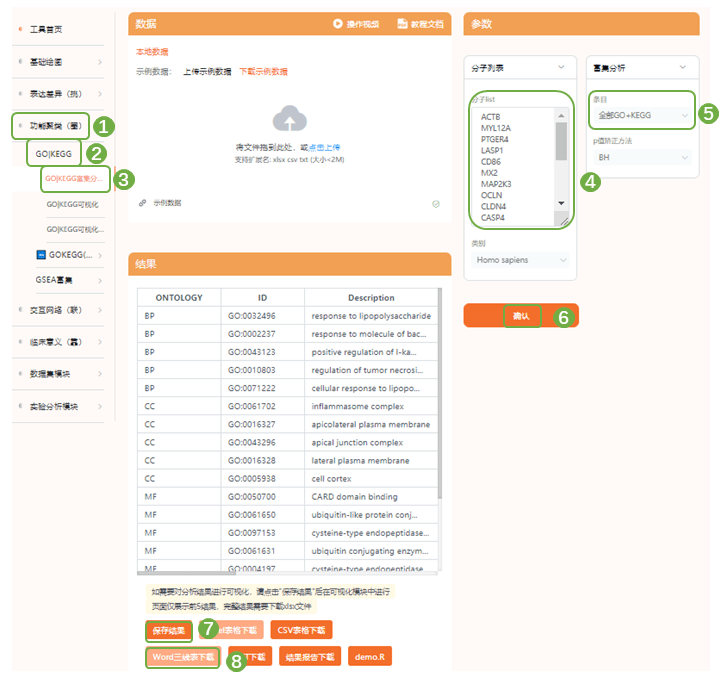
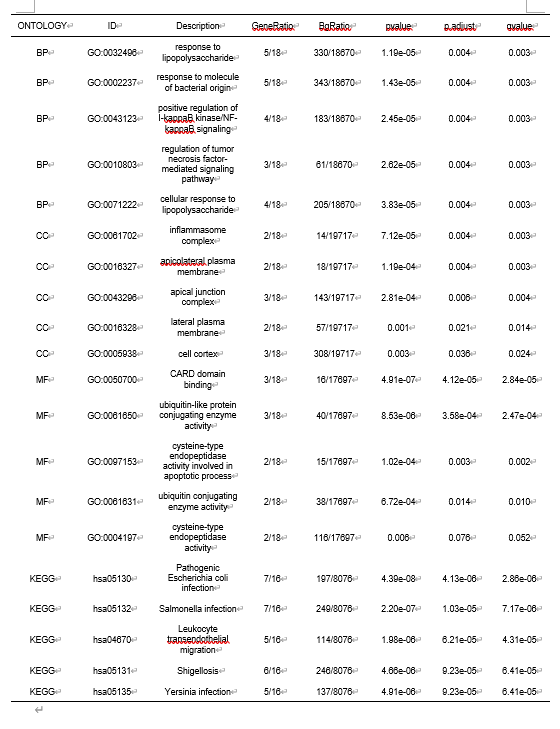
3.9 Fig.8 LUAD中RAB11FIP1表达与免疫浸润水平的关系
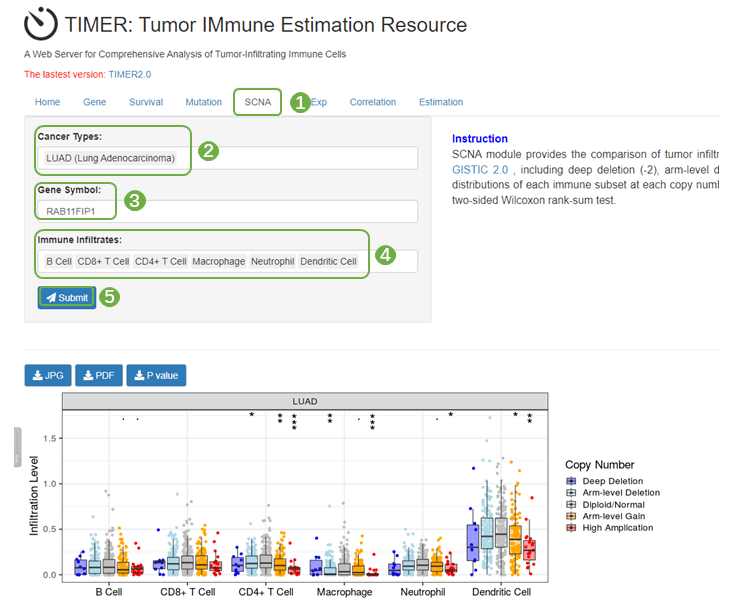
图8 A为TIMER数据库(https://cibersortx.stanford.edu/)中RAB11FIP1拷贝数变异与免疫细胞浸润的相关性分析箱式图与散点图组合
1)进入TIMER数据库,上方的功能模块中选择“SCNA“,肿瘤类型处选择“LUAD”,目标基因为”RAB11FIP1”,免疫浸润细胞与原文一致,即默认的6种细胞类型与顺序,提交分析,下载分析结果图片即可
2)图8 B为RAB11FIP1高、低表达两组种28种免疫细胞浸润箱式图,进入仙桃学术“交互网络(联)——免疫浸润——[云]分组比较图”,选择LUAD数据集,右侧“分子“输入RAB11FIP1,”算法参数“中”细胞“仙桃学术提供了aDC、B cells、CD8 T cells、Cytotoxic cells、DC、Eosinophils、iDC、Macrophages、Mast cells、Neutrophils、NK CD56bright cells、NK CD56dim cells、NK cells、pDC、T cells、 T helper cells、Tcm、Tem、TFH、Tgd、Th1 cells、Th17 cells、 Th2 cells与Treg等20余种免疫细胞类型,”箱/柱“中可对图片配色方案等进行个性化调整,设置坐标轴文本旋转30°,提交分析,得到原文Figure 8B同款图片
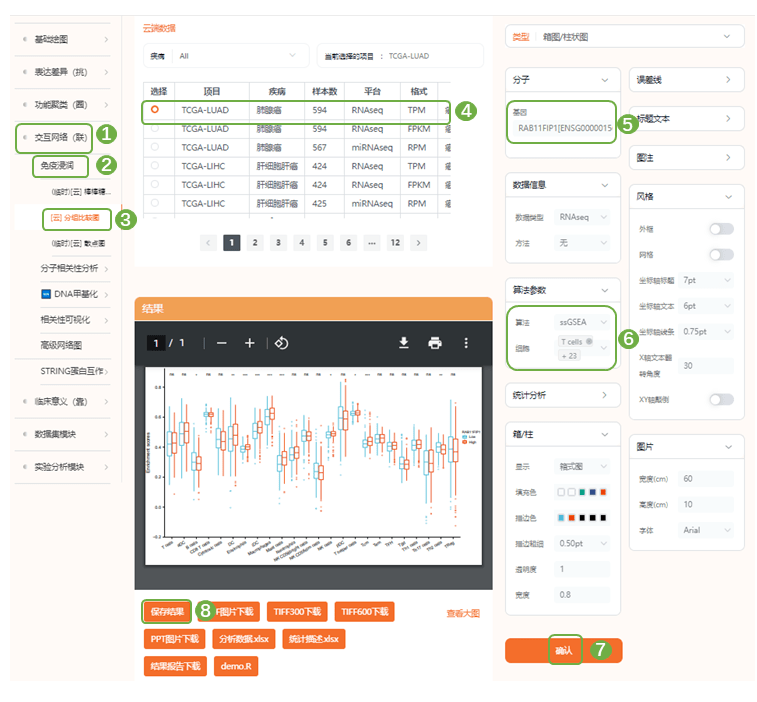
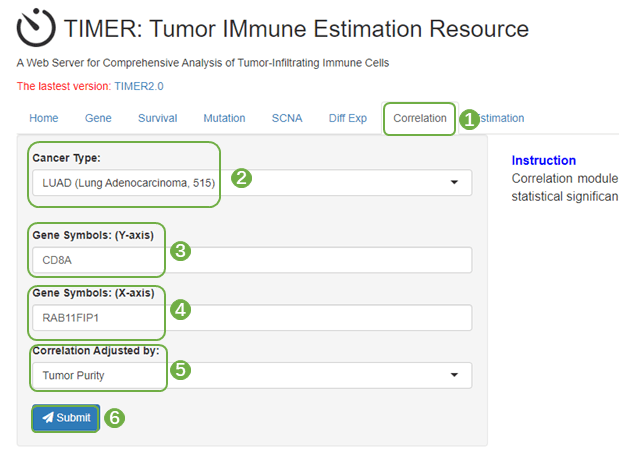
3.10 Table3TIMER数据库分析RAB11FIP1基因与免疫细胞标志基因相关性
表3为TIMER数据库分析RAB11FIP1基因与免疫细胞marker gene的相关性分析结果表格
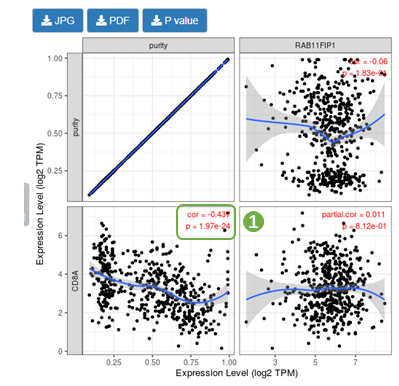
进入TIMER数据库,上方的功能模块中选择“Correlation“,肿瘤类型选择”LUAD“,x轴基因为RAB11FIP1,y轴基因分别为各类免疫细胞的marker gene,根据”tumor purity“进行校正,提交分析,得到分析结果图,将图中的参数结果整理为word三线表即可
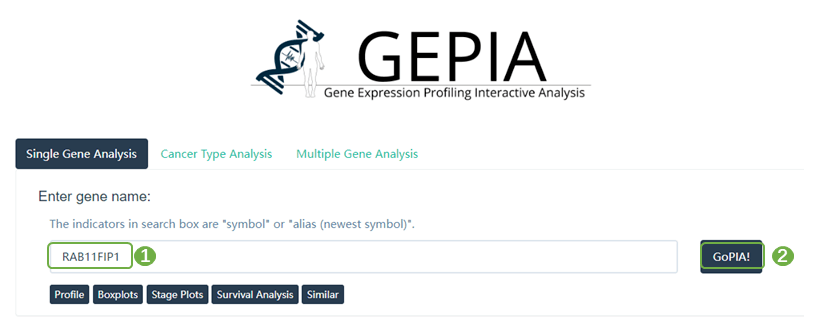
3.11 Table4GEPIA数据库中RAB11FIP1和TIICs标记基因的相关性分析
表4为GEPIA数据库分析RAB11FIP1基因与TIICs (tumor-infiltrating immune cells,肿瘤浸润免疫细胞)marker gene的相关性分析结果表格
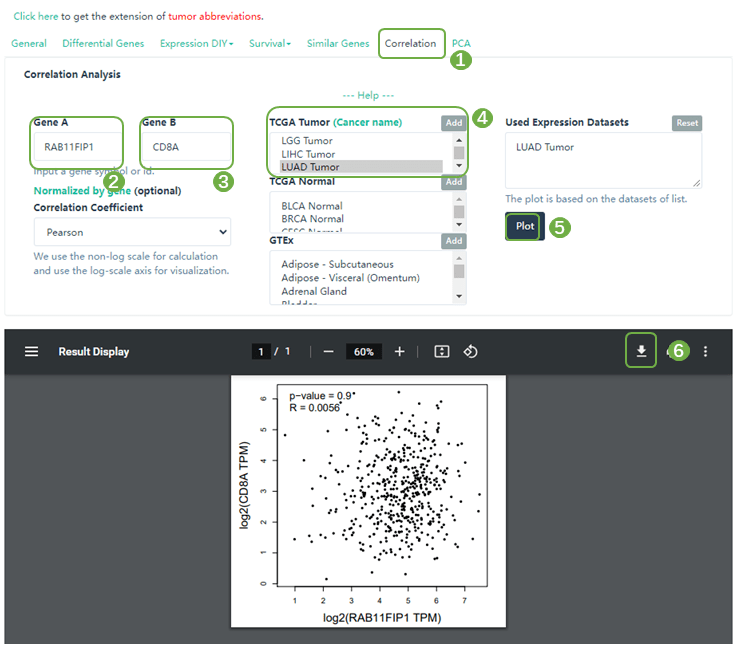
进入GEPIA数据库(http://gepia.cancer-pku.cn/),选择“Single Gene Analysis”,输入目标基因“RAB11FIP1”,开始分析。上方的功能模块中选择“Correlation”分析,x轴基因,即 ”Gene A” 选择 ”RAB11FIP1“,y轴基因,即 ”Gene B“ 为TIICs的marker gene,“TCGA Tumor”选择“LUAD“,提交分析,得到相关性分析的pvalue与相关系数R,整理为word三线表即可
现在文章中所有的图表都复现完毕啦~ 最后依然是我们的保留节目——全文总结
4.文章思路总结
挑
RAB11FIP1在肺腺癌中的表达水平(Figure1)
RAB11FIP1在肺腺癌不同亚组中转录水平(Figure2)
LUAD中基因差异表达与RAB11FIP1的相关性(Figure5)
圈
GO条目与KEGG通路GSEA富集分析(Figure5)
GSEA分析LUAD中RAB11FIP1共表达基因的GO注释和KEGG通路(Figure6)
联
PPI网络的Top2 核心模块(Figure7)
LUAD中RAB11FIP1表达与免疫浸润水平的关系(Figure8)
TIMER数据库分析RAB11FIP1基因与免疫细胞标志基因相关性(Table3)
GEPIA数据库中RAB11FIP1和TIICs标记基因的相关性分析(Table4)
靠
Logistic回归分析RAB11FIP1表达水平与临床病理参数关联(Table1)
RAB11FIP1表达水平与LUAD预后关联(Figure3)
LUAD患者临床病理参数单、多因素Cox分析表格(Table2)
5.全文总结
1
本文以RAB11FIP1基因为主变量,以纯生信分析的手段分析了肺腺癌中RAB11FIP1的异常低表达与肿瘤的预后、生存等临床关联。主变量为其他肿瘤中首次报道,但尚未有肺腺癌中的研究,属于变量移植类研究
2
本篇文章生信分析内容可由7个在线数据库全部复现完成,复现难度偏低,零成本低耗时发到接近5分的3区文章,不比在实验室摸爬滚打,和各种有毒有害试剂打交道、甚至还有危险操作可能的基础文章香太多
3
原文“挑圈联靠”四字箴言面面俱到,由TIMER数据库分析了主变量拷贝数变异与免疫细胞浸润的关系,此处还可扩展延申,如分析LUAD中RAB11FIP1表达显著降低与基因突变、抑癌相关生物学效应的异常改变,以及主变量RAB11FIP1是否具有临床诊断、预后相关预测价值。还可增加外部数据集,如GEO数据库中肺腺癌数据集补充验证;文章若有继续深入研究打算,可再挖掘RAB11FIP1异常低表达的下游生物学效应及其作用靶点,以及异常表达的上游机制;若有较好结果,还可在通过伦理审批后收集临床相关标本进行检测,增加实际应用转化价值。
欢迎大家关注解螺旋生信频道-挑圈联靠公号~
— END—
- 本文固定链接: https://maimengkong.com/morejc/1374.html
- 转载请注明: : 萌小白 2023年2月5日 于 卖萌控的博客 发表
- 百度已收录
