生信分析中我们经常会做KEGG和GO富集分析,一般拿到结果后就想到用柱状图展示pathway的富集情况。比如下面这样:
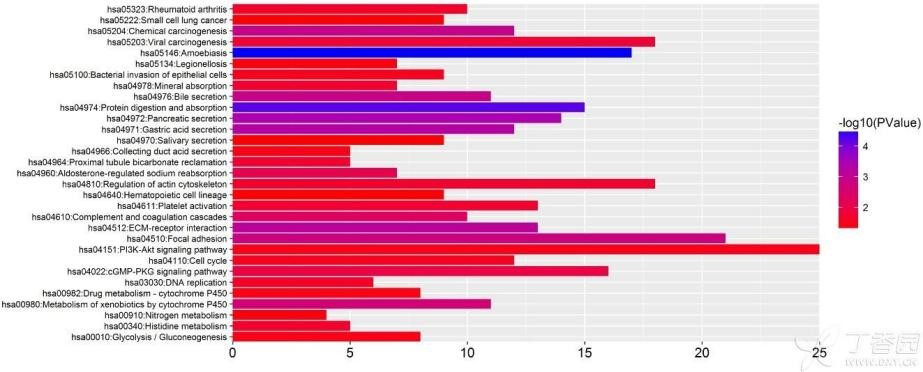
如果学会使用Cytoscape中的ClueGO+CluePedia插件,可以帮我们更形象地展示pathway之间的关系,以及gene在pathway中的富集情况,论文插图瞬间提示一个档次,比如像这样:
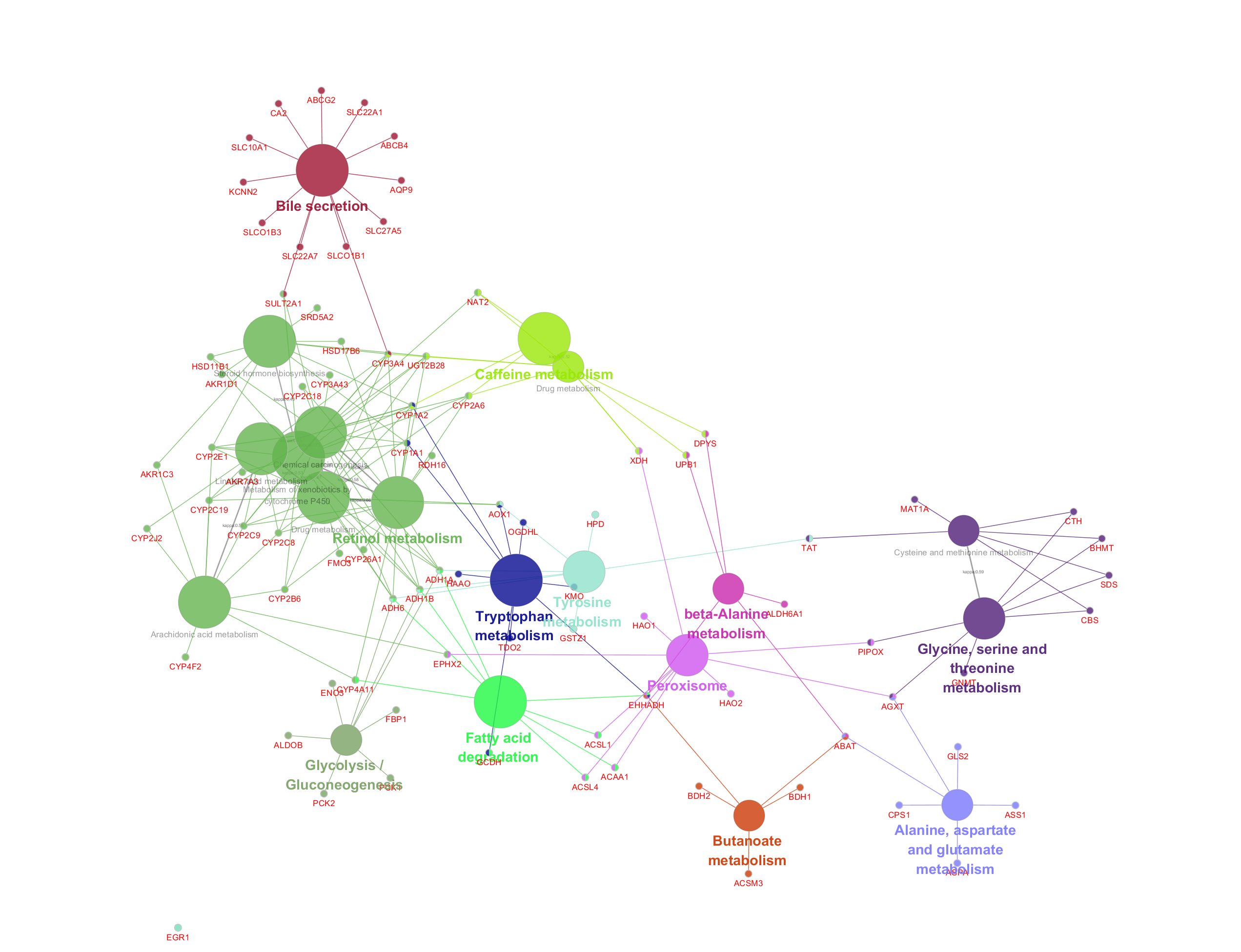
下面我们就来看一下如何使用Cytoscape中的ClueGO和CluePedia插件。
Step1:安装Cytoscape
Cytoscape依赖于最新版本的java,安装前需要先安装好java8。
Cytpscape软件可以在官网下载:http://www.cytoscape.org/download.php。
下载完成后双击图标既可以按照提示完成安装,就不具体介绍了。
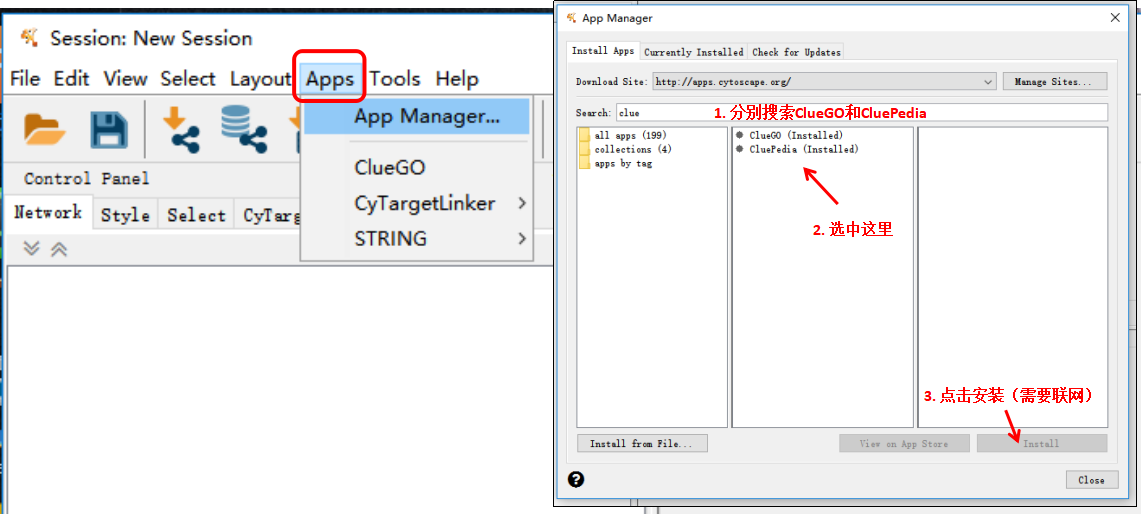
Step2:安装ClueGO和CluePedia插件
安装完成后,打开Cytoscape。在主界面中选择Apps-App Manager,在弹出的对话框中分别搜素ClueGo和CluePedia并安装这两个插件,安装过程需要联网。
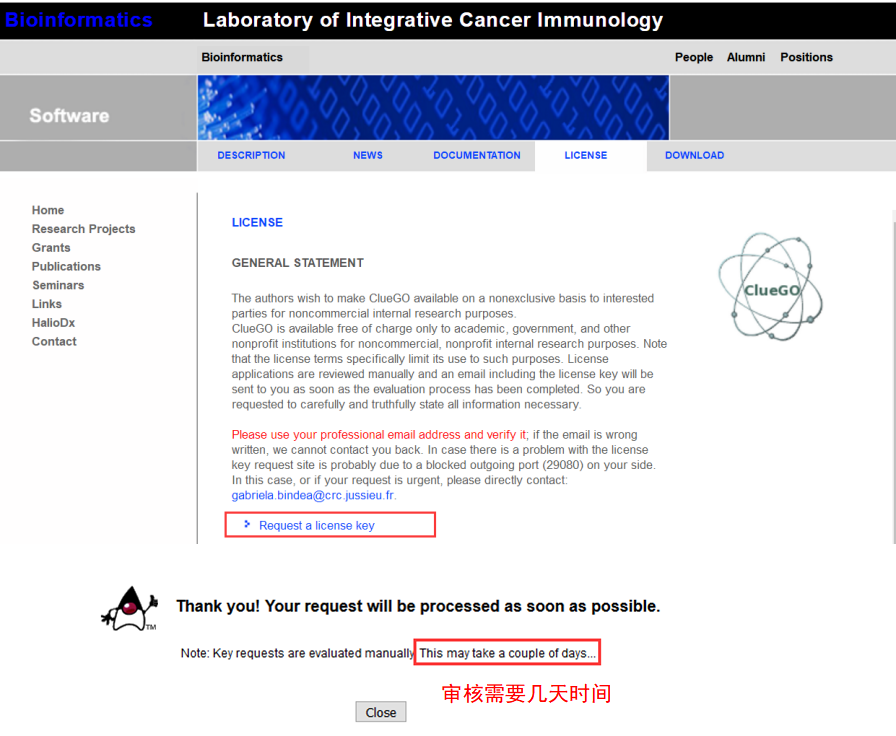
ClueGO第一次使用还需要按照提示申请license,申请网址为;
http://www.ici.upmc.fr/cluego/cluegoLicense.shtml。点击Request a license
key,填写基本信息之后提交审核,审核时间可能需要几天。拿到license之后点击App-ClueGO,在弹出的提示框中输入license即可激活ClueGO。
激活ClueGO
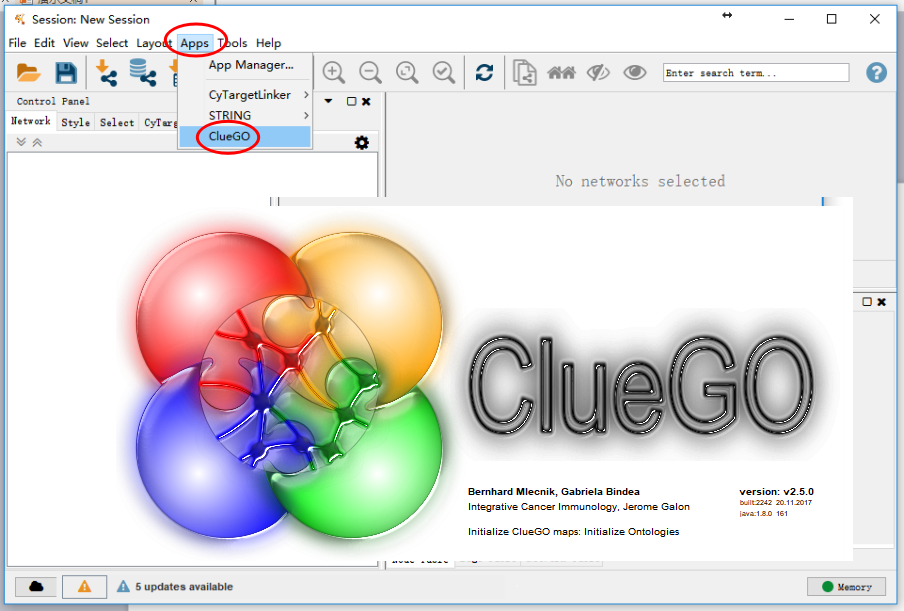
Step3:使用Cytoscape进行pathway富集分析及可视化
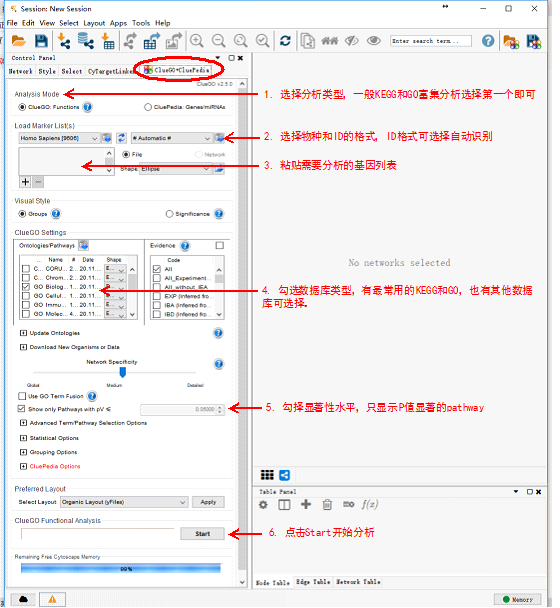
如下图所示,在ClueGO+CluePedia面板中选择相应的参数,并将需要分析的基因列表粘贴到3的方框中,点击start开始分析。
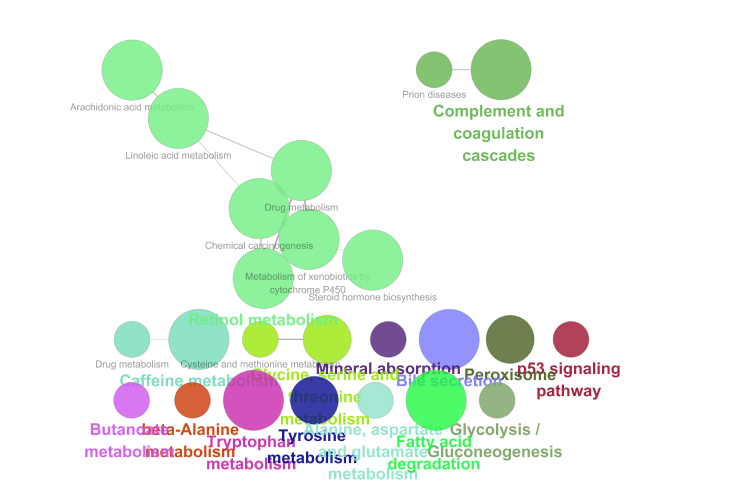
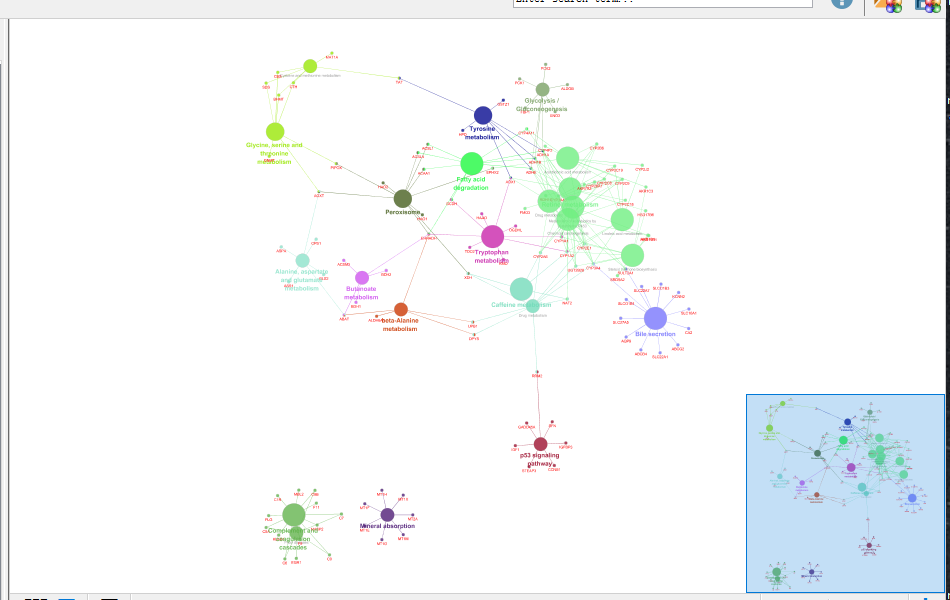
右上角的面板中会显示下图这样的pathway关系图,展示富集出来的显著差异的pathway及其之间的关系,右下角的面板会展示pathway的P值信息:
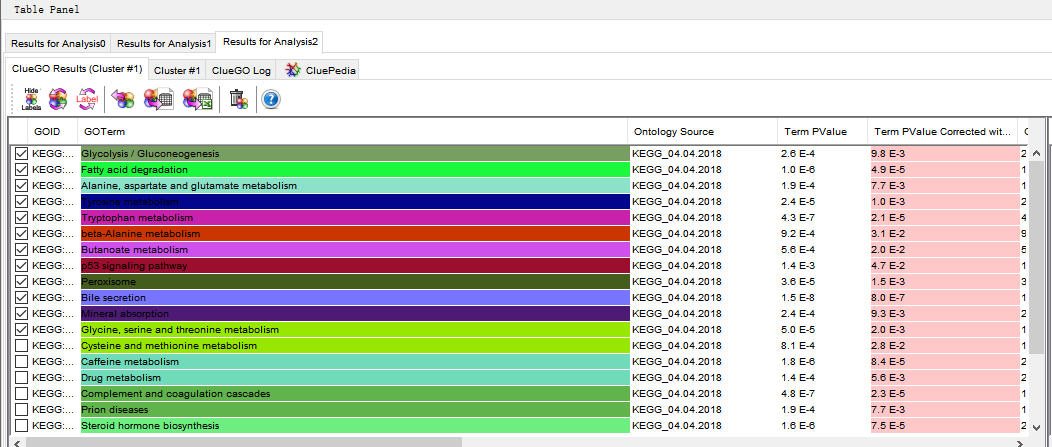
下面,我们还需要用CluePedia对上面得到的图进行优化调整,并且显著基因在pathway中的富集情况。在右下角的面板中点击CluePedia选项,下面有一行选项可以改变网络的展示形式,并且显著基因与pathway之间的联系。之后便可以得到包含基因的pathway图。可以使用鼠标对edge进行微调,也可以在Layout中进一步调整图片展示形式。
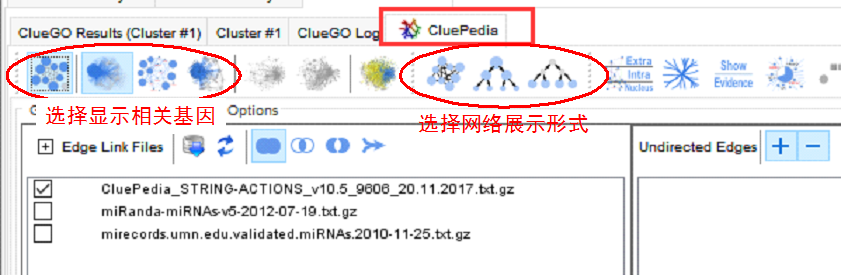
Step4:导出结果
在左上角的选项中,可以选择导出图片和表格,分辨率可以选择最大。
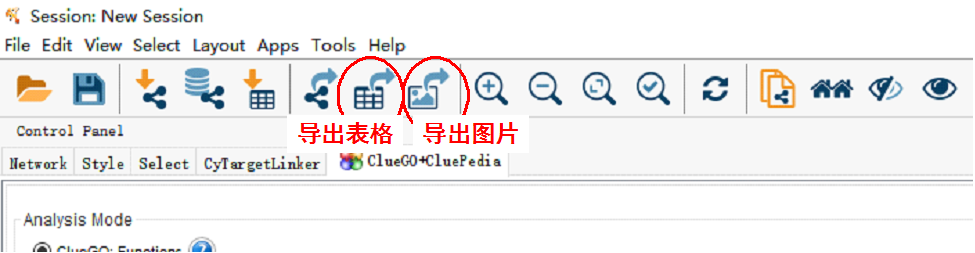
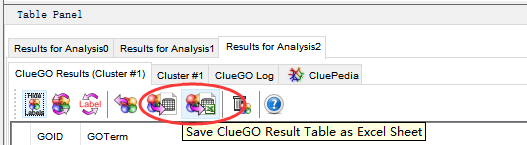
右下角的ClueGO面板中也可以将ClueGO的结果导出为表达。
 ClueGO+CluePedia的使用就介绍到这里,Cytoscape中还有许多非常实用的插件,今后会有更多相关介绍。
ClueGO+CluePedia的使用就介绍到这里,Cytoscape中还有许多非常实用的插件,今后会有更多相关介绍。
转自生信草堂