全基因组关联分析(GWAS)目前已经成为研究复杂性状和疾病遗传变异的有效手段,但是由于群体结构的存在,导致分析结果出现假阳性。经过数十年的发展,新的方法的不断出现,才使得群体结构对分析的影响进一步降低。
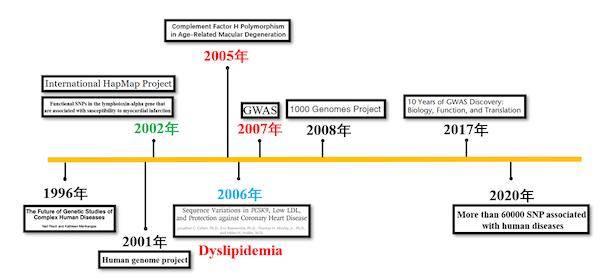
Timeline of GWAS
目前GWAS主要采取两种实验设计,一种是基于情缘关系的群体(Population-based cohorts)设计;另一种是基于无关个体的病例-对照(Case-control)设计。
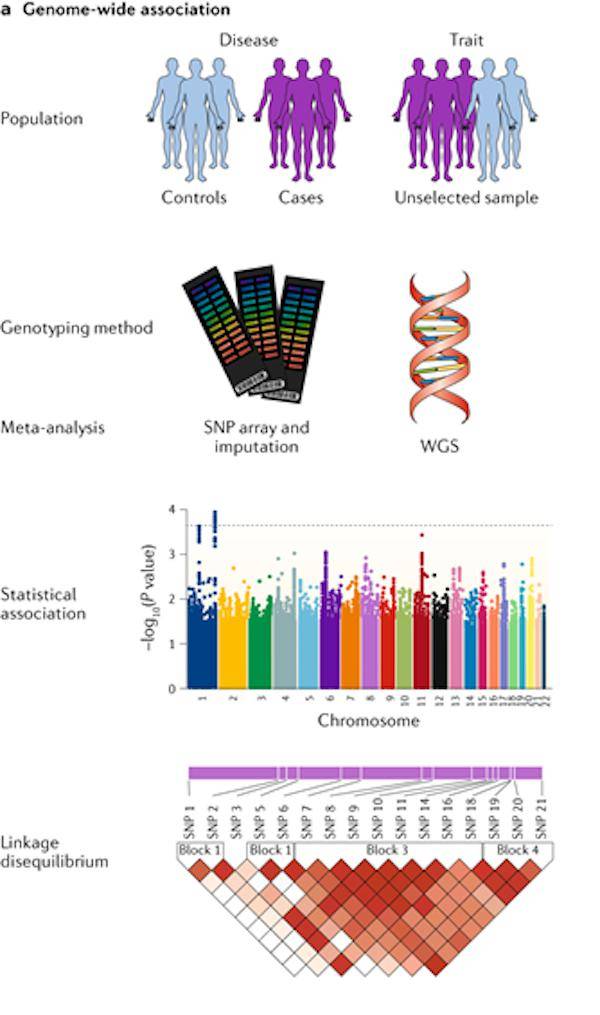
GWAS study design(原文:DOI 10.1038/s41576-019-0127-1)
然而,在进行GWAS分析时,无论采取那种方法,试验个体均会面临由于群体分层和亲缘关系导致的假阳性。
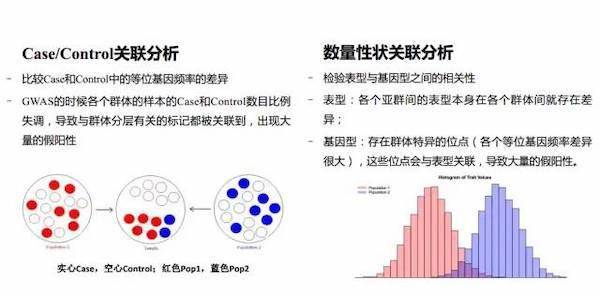
图片来源:简书

群体分层对GWAS的影响(原文 :DOI 10.1097/MOL.0b013e3282f5dd77)
Population
stratification是最常见的差异来源,指的是case/control组的样本来自于不同的祖先群体,其分型结果自然是有差异的。经过十多年的发展,出现了一系列处理群体结构的方法,如Genomic
control、Stratification analysis、Principal components
analysis(PCA)、Mixed-linear model association(MLMA)等,接下来介绍一下这些方法。
1.Genomic control
B Devlin最早提出利用Genomic control来衡量群体结构对关联分析的影响。
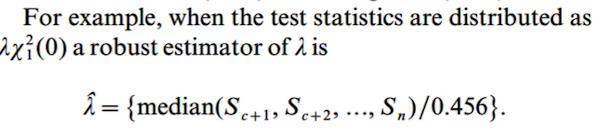
原文:Doi 10.1006/tpbi.2001.1542
虽然Genomic control可以处理群体结构造成的影响,但也具有一定的局限性。当群体结构影响较大时,Genomic control可能效果就不太好了。Genomic control对原始同剂量进行统一矫正,在一定程度上降低了检验的功效。
2.Stratification anlysis
Stratification anlysis是通过聚类将样本分成明确的k个亚群,更直观的解释了群体结构。
3.Principal components analysis
PCA没有具体的模型,可用于亚群分层不明显的样本群体,通过特征向量反映个体间的遗传差异是连续的。

原文:DOI 10.1038/ng1847
4.Mixed-linear model association
使用Mixed-linear model
association做GWAS可以有效校正群体结构和群体内复杂的亲缘关系。其他三种方法均存在不足。目前绝大多数的GWAS都是使用的混合线性模型,GWAS的软件基本也都是基于混合线性模型的,如GAPIT、GCTA、GEMMA,EMMAX等。

原文:Doi 10.1038/ng.2376