什么是SNP?
SNP全称Single Nucleotide
Polymorphism,即单核苷酸多态性,是指在基因组DNA序列中由于单个核苷酸(A,T,C和G)的突变引起的多态性。一个SNP表示基因组某个位点有一个核苷酸的变化,源于单个碱基的转换,颠换,插入和缺失。理论上讲,SNP既可能是二等位多态性,也可能是3个或4个等位多态性,但实际上,后两者非常少见,几乎可以忽略。通常所说的SNP都是二等位多态性的。这种变异可能是转换(C
T,在其互补链上则为GA),也可能是颠换(CA,GT,CG,AT)。转换的发生率总是明显高于其它几种变异,具有转换型变异的SNP约占2/3。
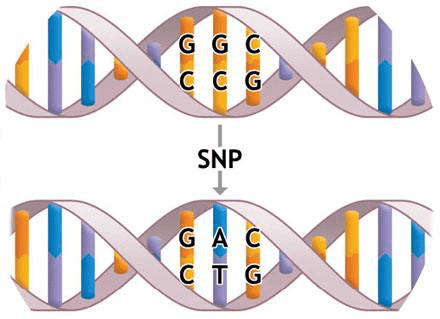
为啥要检测SNP分型呢?
常见的SNP既可以出现在基因编码区,也可以出现在非编码区,虽然发生在编码区的概率相对较小,但会影响基因的功能,导致生物性状改变,在遗传性疾病的研究中具有非常重要的意义。SNP作为第三代遗传标记,其分布密度高,遍布于整个动物和人类基因组中;与功能基因具有高度关联性;突变率低,遗传稳定性强;检测通量高便于自动化分析。某些SNP位点并不直接与疾病基因的表达相关联,但因为它与某些致病基因相邻,所以成为重要的遗传标记。可用于以下研究:

如何获得未知的SNP位点呢?
①利用高通量测序技术
通过与参考基因组(转录组亦是同理)进行比较,获得该物种或群体在全基因组范围内的遗传变异信息(SNP、InDel、SV、CNV)等。

②通过直接测序法查找SNP位点
许多老师在做完测序后会得到大量的SNP(尤其是在做QTL定位或者BSA定位时),这些SNP该如何进行验证呢?
下面小派君就给大家介绍几种SNP验证的方法(派森诺均可以提供这些服务,有意向的老师)。
1)直接测序法
直接测序法是目前最直观,准确性相对而言最高的SNP分型方法,是金标准。在SNP位点两侧设计扩增引物,扩出目标SNP所在的片段,通过一代测序的结果来验证SNP位点碱基情况。
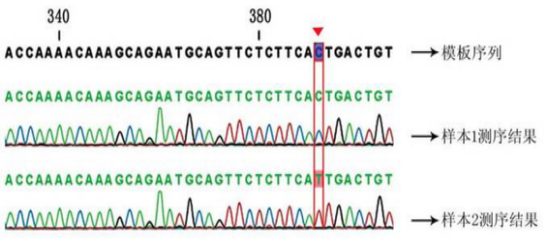
原始测序SNP位点图
2)SNaPshot技术
基于荧光标记单碱基延伸原理的分型技术,含有测序酶、四种荧光标记ddNTP、紧临多态位点5’端的不同长度延伸引物和PCR产物模板的反应体系中,引物延伸一个碱基即终止,经ABI测序仪检测后,根据峰的移动位置确定该延伸产物对应的SNP位点,根据峰的颜色可得知掺入的碱基种类,从而确定该样本的基因型。
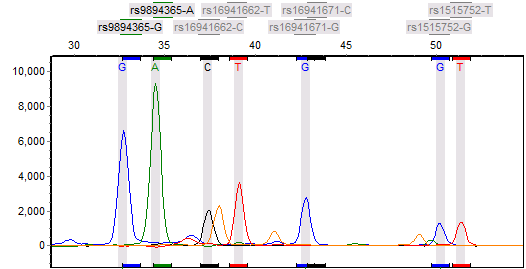
SNP位点分型图
3)PCR-LDR 分型技术
LDR是利用高温连接酶实现对基因多态性位点的识别。高温连接酶一旦检测到DNA与互补的两条寡聚核苷酸接头对应处存在着基因点突变类型的碱基错配,则连接反应就不能进行。
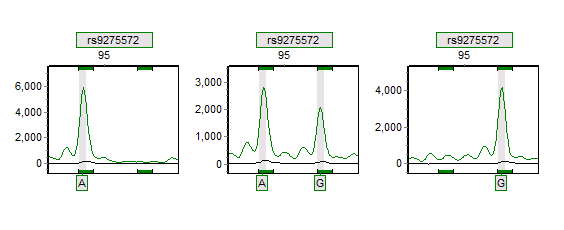
SNP位点分型图
4)TaqMan探针法
针对染色体上的不同的SNP位点别设计1对PCR引物和TaqMan探针,进行实时荧光PCR扩增,该探针的5’端和3’端分别标记一个报告荧光基团和一个淬灭荧光基团。当溶液中有PCR产物时,该探针与模板退火,即产生了适合于核酸外切酶活性的底物,从而将探针5’端连接的荧光分子从探针上切割下来,破坏两荧光分子间的PRET,发出荧光。通常用于少量SNP位点分析。
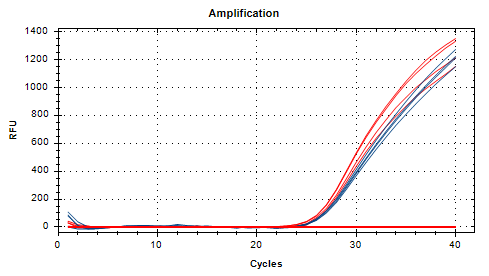
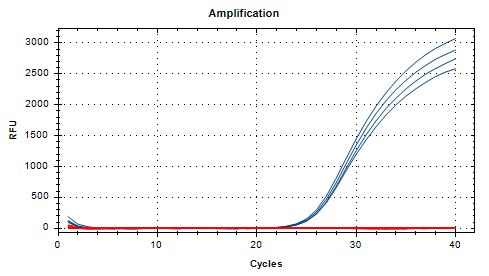
FAM:蓝色,基因型T;VIC:红色,基因型C
5)焦磷酸测序法
焦磷酸测序技术是一种基于四种酶(DNA聚合酶、 硫酸化酶、荧光素酶和双磷酸酶)的级联反应来进行定量序列分析的技术。在整个反应体系中, 以
PCR产物的一条单链为模板,与测序引物退火结合后,按照事先计算好的顺序将四种 dNTP 依次加到样品中。如果加入的这种 dNTP
与测序引物3’下游+1位所对应的模板链上的碱基互补,就会发生一系列酶的级联反应。通过测序结果可以直观的看出各位点基因型的占比,其可重复性和精确性能与Sanger
DNA测序法相媲美,而速度却大大的提高。
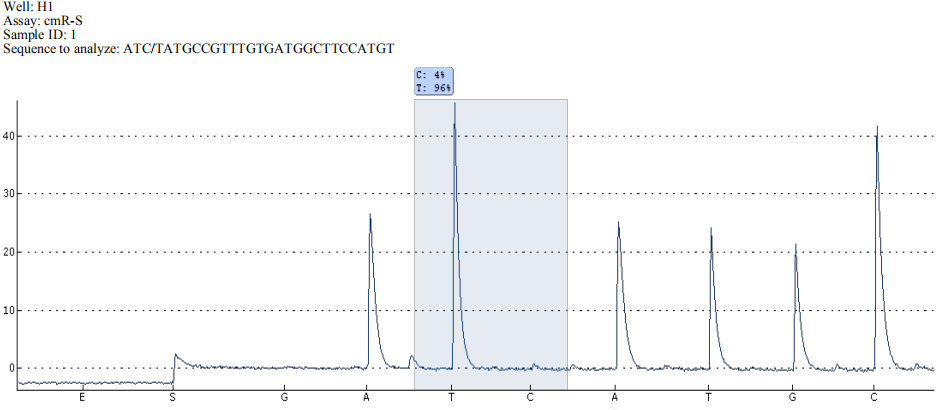
焦磷酸测序结果展示
SNP分子标记多态性高、密度大、遗传稳定性高、易于实现自动化高通量基因分型,但现阶段仍存在一些不足,不过随着SNP分型技术的不断改进,各类数据库的逐步完善,SNP在各方面研究应用的广度和深度将不断推进。不同的类型需要使用不同的方法,不同的方法努努力总是可以达到大家的预期,欢迎大家咨询!