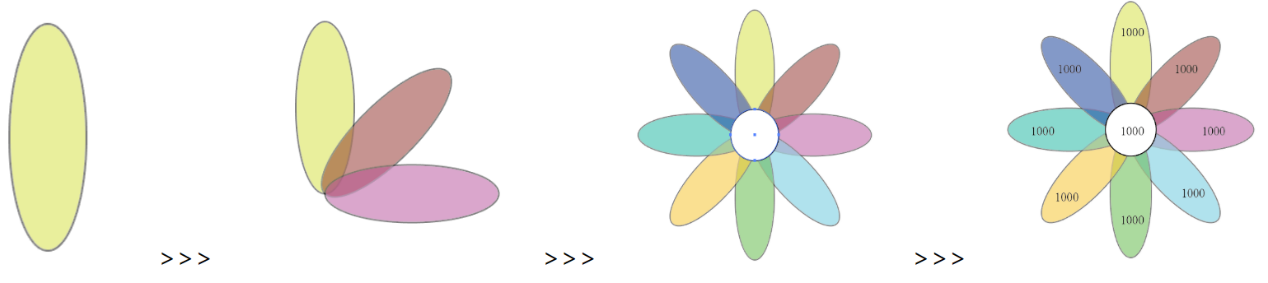
此处结合微生物群落研究中的16S扩增子分析数据,给大家分享怎样在R中进行韦恩图(Venn plot)和花瓣图(Flower plot)绘制。
在R中,可用于绘制韦恩图的R包有很多可供选择,本文中选择了VennDiagram、venn、UpSetR共3个R包进行介绍。同时在本篇博文的最后,给出了3个可在线绘制韦恩图的网站,以提供更多的可选方法。
作图示例文件、R脚本等,已上传至百度盘(若失效请在下方留言)。
示例文件简要
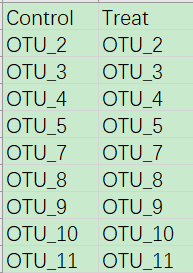
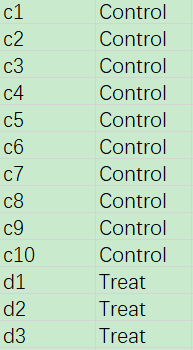
文件“venn.txt”为韦恩图作图文件,其内容展示如下。
该文件中共两列信息,Control和Treat为两个样本,其下的内容为各样本中所含OTU种类名称(这里即丰度大于0的OTU的名称)。
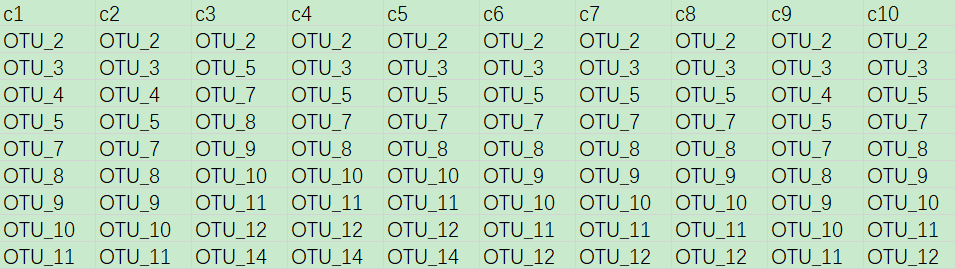
文件“flower.txt”为韦恩图/花瓣图作图文件,其内容展示如下。
该文件中共20列信息,即20个样本,其下的内容为各样本中所含OTU种类名称(这里即丰度大于0的OTU的名称)。
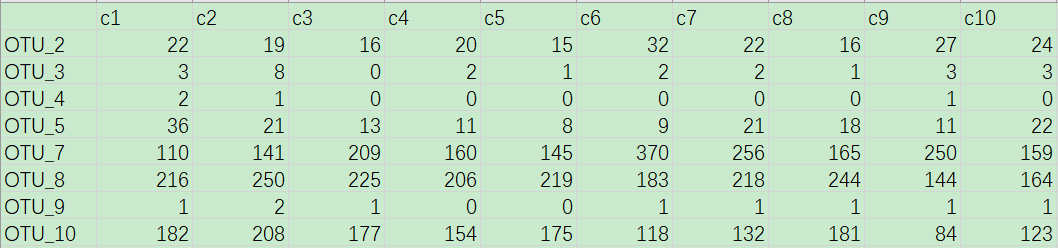
文件“otu_table.txt”为OTU丰度表格,其内容展示如下。
每一列为一个样本,此处共计20个样本;每一行为一种OTU;交叉区域为每种OTU在各样本中的丰度。
文件“group1.txt”和“group2.txt”为样本分组信息,其内容展示如下。
第一列为各样本名称,共计20个样本;第二列为各样本的分组信息。这里提供了两种不同的分组信息文件,用于信息提取构建作图数据,详见下文。
基于OTU丰度表获得韦恩图(Venn plot)/花瓣图(Flower plot)作图输入文件
对于作图输入文件样式,上述已经做了简要说明。
在正文开始之前,首先提供一个自编的R脚本,方便基于OTU丰度表转化为韦恩图或花瓣图作图输入格式。
这里的R脚本也就是附件中的“make_venn_flower.r”,在linux命令行界面下的运行示例如下。该脚本可依据样本分组信息,将属于同一分组的样本合并,并且依据合并后的结果,将丰度数值大于0的元素名称(本示例中为OTU)提取出,得到输出文件(即后续用来作图的输入文件)。
#shell 命令行样式
Rscript make_venn_flower.r <otu_file> <group_file> <output_file>
#shell 命令行示例
Rscript make_venn_flower.r otu_table.txt group1.txt venn.txt
Rscript make_venn_flower.r otu_table.txt group2.txt flower.txt
使用示例命令,可将“otu_table.txt”分别转化为“venn.txt”和“flower.txt”,即上文中提及的用于绘制韦恩图和花瓣图的文件。
若不使用命令行,直接在R中运行的话,可编辑此脚本,将“args[1]”、“args[2]”、“args[3]”分别修改为OTU丰度表名称、样本分组文件名称、输出文件名称。之后直接在R中运行即可。

使用R绘制韦恩图(Venn plot)
(VennDiagram包,常规韦恩图,2<=样本数<=5)
首先展示一个使用R的VennDiagram包绘制常规韦恩图的方法,该包适用于2<=样本数<=5的情形。
首先读入韦恩图作图数据,并从中提取出每列(样本)所含所有OTU的名称。
#读入作图文件,预处理
venn_dat <- read.delim('venn.txt', header = T, sep = '\t', stringsAsFactors = F, check.names = F)
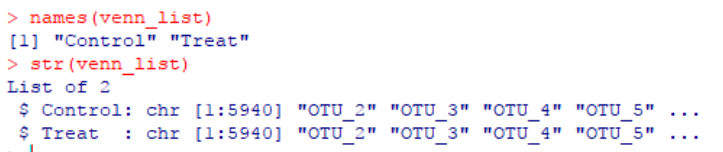
venn_list <- list(venn_dat[,1], venn_dat[,2])
names(venn_list) <- colnames(venn_dat)
本示例使用的数据共2列内容,将2列内容提取出赋值给新列表venn_list,同时将样本名称赋值到列表中。
导入VennDiagram包绘制韦恩图,以下示例命令中的参数设置较为简略,对于venn.diagram()的详细参数,可使用?venn.diagram()查看帮助。
library(VennDiagram)
#作图
venn.diagram(venn_list, filename = 'venn.png', imagetype = 'png', margin = 0.2,
fill = c('red', 'blue'), alpha = 0.50, col = 'black', cex = 1, fontfamily = 'serif', cat.col = c('black', 'black'), cat.cex = 1, cat.fontfamily = 'serif')
上述所得新列表venn_list即为可被venn.diagram()所识别的作图数据;fill设置圆圈填充颜色,alpha设置透明度,col设置边界颜色;cex为圈内数值字体大小,col为圈内数值字体颜色,fontfamily设置字体样式;cat.cex为样本标签字体大小,cat.col为样本标签字体颜色,cat.fontfamily设置字体样式。更详细的参数说明可查看帮助。
最终作图结果“venn.png”如下所示。Control样本特有OTU共计2458个,Treat样本特有OTU共计806个,两样本共有OTU共计3482个。

使用R绘制韦恩图(Venn plot)(venn包,常规韦恩图,2<=样本数<=7)
通常韦恩图所展示的样本数量不会超过5,因为当样本数量大于5时,绘图结果将难以观测。因此对于VennDiagram包来讲,它所支持的样本数量也是最多为5个。其实不止R的VennDiagram包,其它可绘制韦恩图的R包或除R外的其它绘图工具等,一般也不支持样本数大于5个的情形。
因此,在这里继续给大家分享一个叫“venn”的R包,它最多可支持7个样本的韦恩图。
首先读入韦恩图作图数据,并从中提取出每列(样本)所含所有OTU的名称。
#读入作图文件,预处理
venn_dat <- read.delim('flower.txt', header = T, sep = '\t', stringsAsFactors = F, check.names = F)[1:7]
venn_list <- list(venn_dat[,1], venn_dat[,2], venn_dat[,3], venn_dat[,4], venn_dat[,5], venn_dat[,6], venn_dat[,7])
names(venn_list) <- colnames(venn_dat)
与上述使用VennDiagram包绘制韦恩图的流程相似,首先我们需要将样本列的内容全部提取出,赋值给新列表venn_list,同时将样本名称赋值到列表中。本示例共使用了7个样本数据。

导入venn包绘制韦恩图。作为简要演示,以下示例命令中的参数均使用默认参数,对于venn()的详细参数,可使用?venn()查看帮助。
library(venn)
#作图
png('venn_7.png', width = 1500, height = 1500, res = 200, units = 'px')
venn(venn_list, zcolor = 'style')
dev.off()
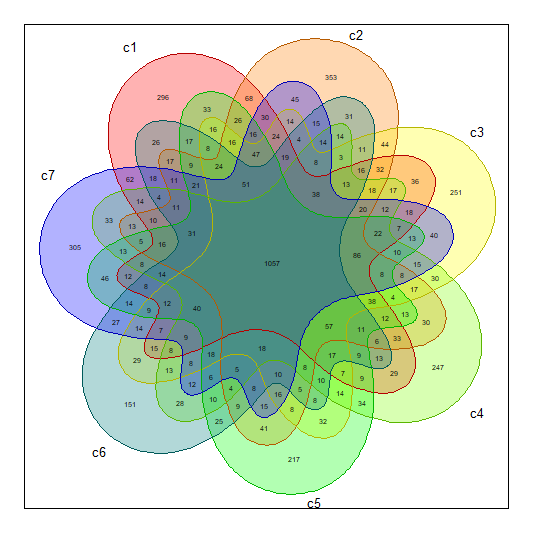
最终作图结果“venn_7.png”如下所示,包含了7个样本。
使用R绘制韦恩图(Venn plot)(UpSetR包,特殊韦恩图,不受样本数量限制)
上述介绍了如何在R中绘制常规样式的韦恩图。我们不难发现,当样本数量增多时,韦恩图将难以表现多样本间的共有/特有元素。因此,这种样式的韦恩图适用于样本数量较少的情况(样本数通常小于5)。
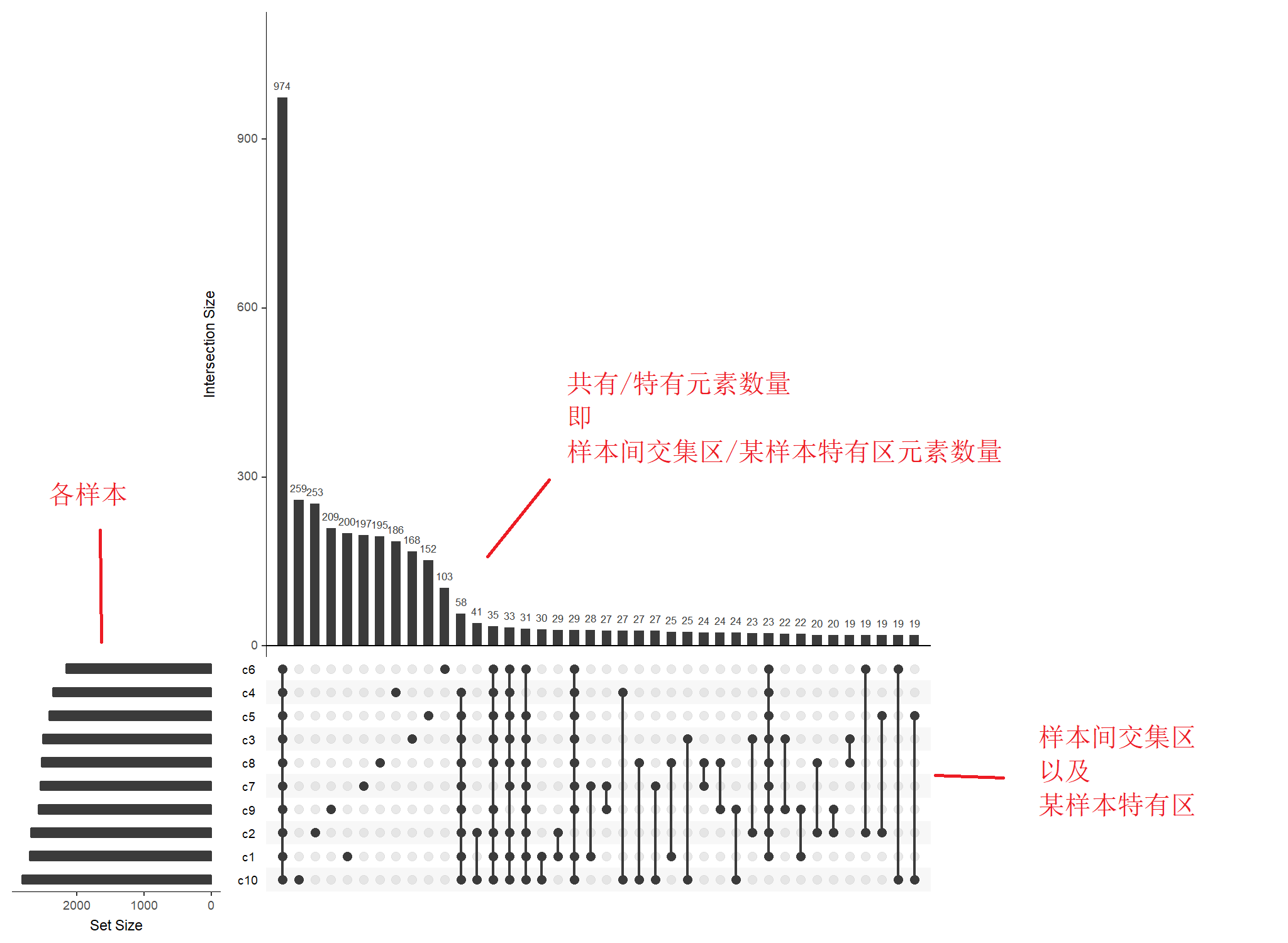
那么,当样本量较多时,应该如何使用韦恩图表示呢?此处给大家介绍一种特殊的韦恩图,它使用UpSetR包实现,不受样本数量的限制,只要样本数大于2均可适用。下述示例使用UpSetR包绘制10个样本数的韦恩图。尽管样本量很多时可能仍难以表示样本间共有/特有元素,但比常规韦恩图要好很多。
此处我们使用OTU丰度表格作为输入文件,并将OTU丰度表转化为0-1数值。
#读入作图文件,预处理
otu <- read.delim('otu_table.txt', header = T, row.names = 1, sep = '\t', stringsAsFactors = F, check.names = F)[1:10]
otu[otu > 0] <- 1
导入UpSetR包绘制韦恩图。此处绘制10个样本的韦恩图,对于upset()的详细参数,可使用?upset()查看帮助。
#作图
png('venn_10.png', width = 1500, height = 1500, res = 200, units = 'px')
upset(otu, nsets = 10, order.by = "freq")
dev.off()
upset()参数中,nsets用于指定所统计样本的个数;此处作为简要演示,其余参数使用默认参数。更详细的参数说明可查看帮助。
最终作图结果“venn_10.png”如下所示,包含了10个样本。这类的韦恩图可能第一眼看去有些抽象,其实所含内容很简单。
左侧为各样本名称。右侧点阵区域,为样本间交叉数据集。例如,若两个样本间有相同的元素,则使用连线将两个样本相连;同样地,若多个样本间存在连线,那这部分即为这几个样本内的共有元素。右侧柱状图区,为样本间交叉数据集中元素的数量。
例如,图中展示了10个样本,10个样本间共有元素数量为974;c10样本特有元素259;c2和c10共有元素41;以此类推。
UpSetR包功能十分强大,且已经得到了广泛的应用。当我们对该包的语法用熟练了之后,可以尝试进行一些高级统计绘图,如下所示,不再多加说明了(本人也没深入研究过……)。
使用R绘制花瓣图(Flower plot)
在早期UpSetR等工具包未出现时,更多时候在大样本量的情况下,考虑使用花瓣图展示共有/特有元素数量分布。其优点是内容简洁易懂,缺点是只能给出所有样本的共有元素数量,无法给出某几个样本间的共有元素数量。
其实对于怎样在R中绘制花瓣图我也是纠结了好久的,因为找不到相关的R包。最后偶然看到了某大神的博客(https://www.cnblogs.com/xudongliang/p/7884667.html),提到可以考虑依次绘制椭圆并按一定角度旋转得到花瓣图,并分享了绘图的R代码。这里就搬运此大神的代码,同时添加了一些改动便于文件读入和输出以及颜色赋值等。
首先读入花瓣图作图数据,并计算得到各样本所包含OTU种类总数以及所有样本共有OTU的数量等。
#读入做图文件,预处理
flower_dat <- read.delim('flower.txt', header = T, sep = '\t', stringsAsFactors = F, check.names = F)
sample_id <- colnames(flower_dat)
otu_id <- unique(flower_dat[,1])
otu_id <- otu_id[otu_id != '']
core_otu_id <- otu_id
otu_num <- length(otu_id)
for (i in 2:ncol(flower_dat)) {
otu_id <- unique(flower_dat[,i])
otu_id <- otu_id[otu_id != '']
core_otu_id <- intersect(core_otu_id, otu_id)
otu_num <- c(otu_num, length(otu_id))
}
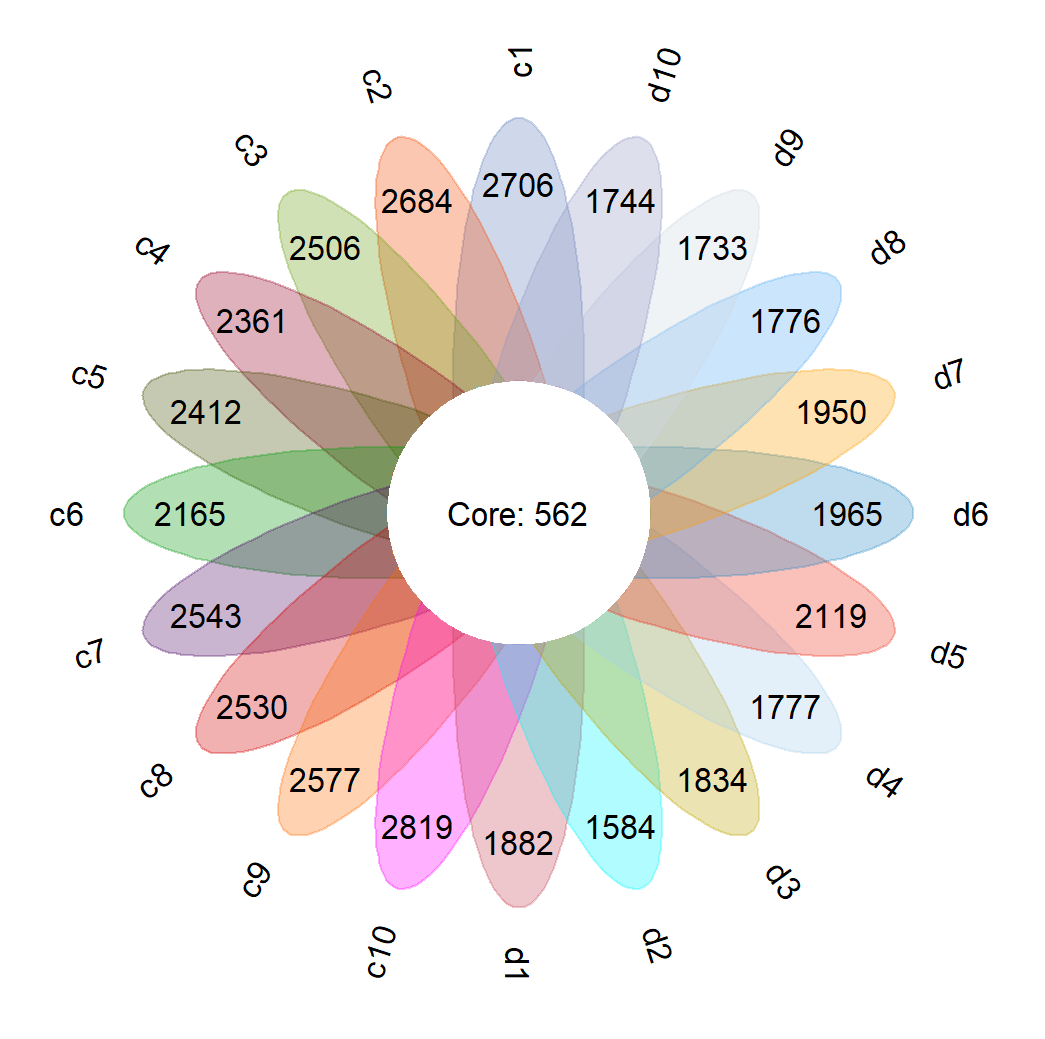
core_num <- length(core_otu_id)
向量sample_id为各样本名称;otu_num为各样本中所含OTU种类总数,各数值与sample_id中的各样本名称相对应;core_num为所有样本的共有OTU种类数。

然后构建一个绘制花瓣图的函数。该函数中的部分命令(绘制椭圆的功能,下文中的draw.ellipse()、draw.circle()等命令)需要plotrix包实现。
library(plotrix)
#定义备选颜色
ellipse_col <- c('#6181BD4E','#F348004E','#64A10E4E','#9300264E','#464E044E','#049a0b4E','#4E0C664E','#D000004E','#FF6C004E','#FF00FF4E','#c7475b4E','#00F5FF4E','#BDA5004E','#A5CFED4E','#f0301c4E','#2B8BC34E','#FDA1004E','#54adf54E','#CDD7E24E','#9295C14E')
#构建作图函数(参考自 https://www.cnblogs.com/xudongliang/p/7884667.html)
flower_plot <- function(sample, otu_num, core_otu, start, a, b, r, ellipse_col, circle_col) {
par( bty = 'n', ann = F, xaxt = 'n', yaxt = 'n', mar = c(1,1,1,1))
plot(c(0,10),c(0,10),type='n')
n <- length(sample)
deg <- 360 / n
res <- lapply(1:n, function(t){
draw.ellipse(x = 5 + cos((start + deg * (t - 1)) * pi / 180),
y = 5 + sin((start + deg * (t - 1)) * pi / 180),
col = ellipse_col[t],
border = ellipse_col[t],
a = a, b = b, angle = deg * (t - 1))
text(x = 5 + 2.5 * cos((start + deg * (t - 1)) * pi / 180),
y = 5 + 2.5 * sin((start + deg * (t - 1)) * pi / 180),
otu_num[t])
if (deg * (t - 1) < 180 && deg * (t - 1) > 0 ) {
text(x = 5 + 3.3 * cos((start + deg * (t - 1)) * pi / 180),
y = 5 + 3.3 * sin((start + deg * (t - 1)) * pi / 180),
sample[t],
srt = deg * (t - 1) - start,
adj = 1,
cex = 1
)
} else {
text(x = 5 + 3.3 * cos((start + deg * (t - 1)) * pi / 180),
y = 5 + 3.3 * sin((start + deg * (t - 1)) * pi / 180),
sample[t],
srt = deg * (t - 1) + start,
adj = 0,
cex = 1
)
}
})
draw.circle(x = 5, y = 5, r = r, col = circle_col, border = NA)
text(x = 5, y = 5, paste('Core:', core_otu))
}
最后使用提取出的做图数据以及构建好的作图函数作图。
#作图
png('flower.png', width = 1500, height = 1500, res = 200, units = 'px')
flower_plot(sample = sample_id, otu_num = otu_num, core_otu = core_num,
start = 90, a = 0.5, b = 2, r = 1, ellipse_col = ellipse_col, circle_col = 'white')
dev.off()
#注:参数a和b用于设置花瓣椭圆的尺寸,ellipse_col用于设置花瓣椭圆的颜色;参数r用于设置中心圆圈尺寸,circle_col用于设置中心圆圈的颜色
本示例的最终结果“flower.png”如下。共包含20个样本,外圈数值为各样本内所含OTU种类总数,中心圆圈内为共有OTU数量。
附加内容:可在线绘制韦恩图的网站
在这里,给大家介绍3个常用的可在线绘制韦恩图的网站,使用起来也挺方便的。
当然还有更多的可在线绘制韦恩图的网站。由于现有的方法足以满足我们使用了,因此这里就不再一一列出了。
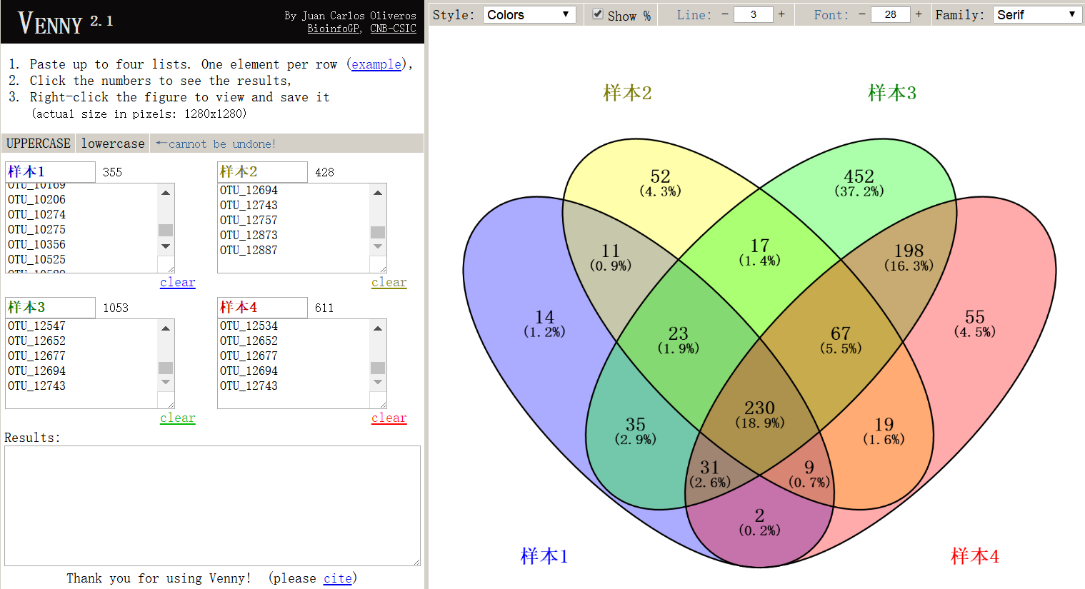
第一个,http://bioinfogp.cnb.csic.es/tools/venny/index.html
这个在线网站操作起来最为简单,将每个样本中出现的OTU粘贴至左侧,最多可以展示4个样本。右侧自动出图,在图的上方可以设置颜色显示、文字显示等选项。缺点是颜色是固定的。
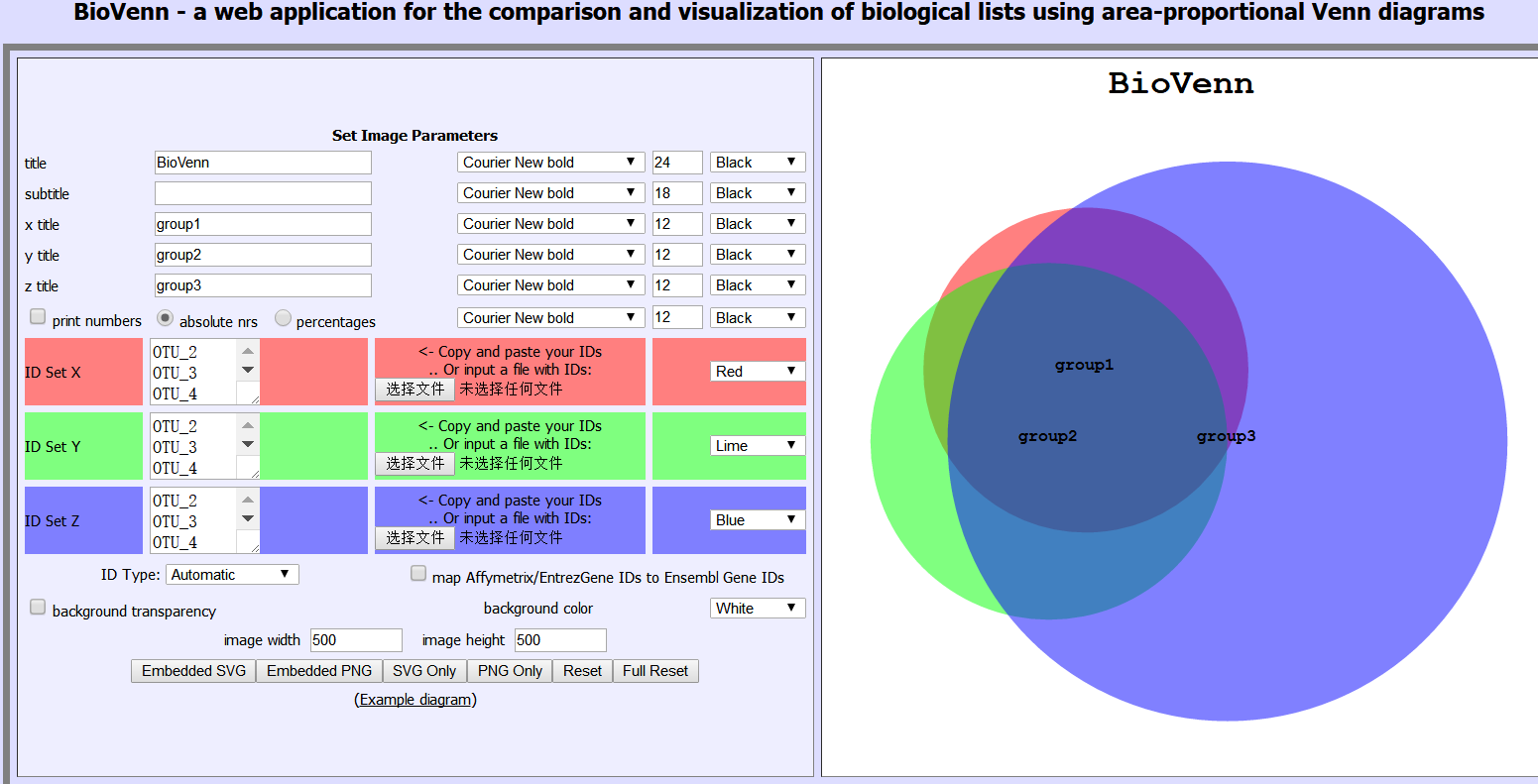
第二个,http://www.biovenn.nl/index.php
相较于上一个在线网站,优势在于有多种颜色可以选择,并且按样本所含元素多少将每个样本以不同大小的圆圈展示出来。缺点是最多只能展示3个样本,无法给出不同样本中所共有(或特有)OTU的数目,且样本名称排列的位置无法调整(样本名称默认展示在每个样本圆圈的中心位置,但会造成与其它样本圈的重叠影响查看)。数据导入既可以直接粘贴,也可以以文件(一个文件中只能包含一个样本的全部OTU)的形式导入避免数据量大时粘贴麻烦。
第三个,http://bioinformatics.psb.ugent.be/webtools/Venn/
第三个网站功能强大。其一,数据导入既可以直接粘贴,也可以以文件(一个文件中只能包含一个样本的全部OTU)的形式导入避免数据量大时粘贴麻烦;其二,可最多展示5个样本;其三,结果中不仅给出了不同样本中所共有(或特有)OTU的数目,还精确到出现在相同(或不同)样本中的OTU具体是哪些OTU。但是它无法做到上述第二个网站那样,根据不同样本内所含元素个数将各样本以不同大小圆圈表示;相较于第一个网站,无法同时给出样本共有或特有元素的数量和比例信息。
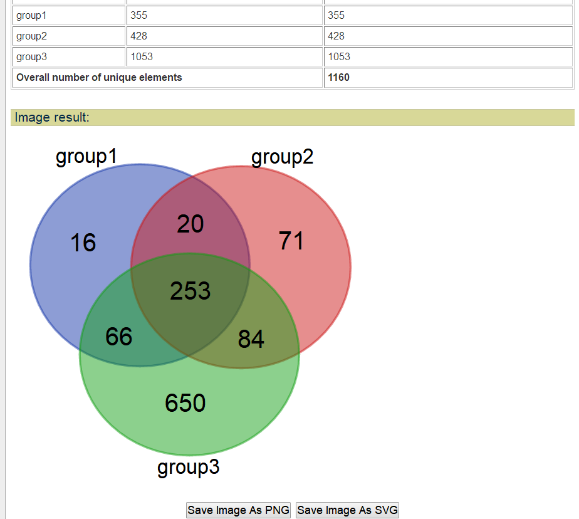

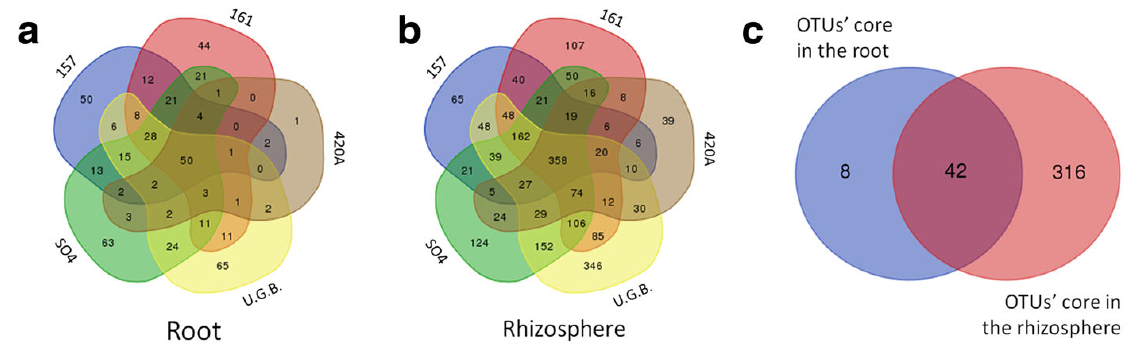
这里有必要说明一点,第三个在线网站的效果非常好不比R差,很多文章中的韦恩图就使用这个网站绘制。例如以下这张截图出自某文献,这种风格的韦恩图即来自该在线网站。
附加内容:后期调整及手动绘图
后期使用AI、PS等工具,可以对所得韦恩图或花瓣图进行调整,例如字体大小、字体位置、椭圆颜色、或多图组合等。此外,我们可以灵活运用多种方式绘制韦恩图,然后再用AI、PS等工具编辑组合,将各绘图方法的优点结合起来,得出集多个优点于一身的韦恩图。
比如以下示例韦恩图的做法:首先,使用上述第二个在线网站生成3个圈的轮廓,导出圆圈背景的svg矢量图(不要导出文字);然后使用上述第一个在线网站计算每个区域共有(或特有)OTU数目及比例;使用AI打开导出的矢量图,在每个区域添加计算好的数值,然后组合多个图形,排列位置即可成型。
此外,鉴于AI等工具功能强大,加上韦恩图或花瓣图构造简单,其实我们也可考虑手动绘制韦恩图或花瓣图。当然这种方法可能不建议,因为会比较繁琐,特别是样本较多时。但因为此前我并没能找到绘制花瓣图的R命令,最后出于无奈均是使用AI从头绘制花瓣图,前后也画了许多……所以在这里顺便分享一下绘图经验了。
这里就简单展示一个使用AI手绘花瓣图的流程,思路如下:第一步,计算多样本中共有otu以及特有otu;第二步,在AI中绘制一个椭圆,调整好椭圆参数;第三步,根据样本数量的多少,按一定角度(360度/样本数)复制旋转椭圆,最后在得到的“花瓣”正中心添加正圆;第四步,添加文字注释信息等。