ChIP测序中关键的结果图,这些结果即体现了ChIP实验和测序的成功,可以帮助我们了解目的蛋白的结合甚至调控特征,都是撰写文章可以利用的极佳素材。说了这么多好像还是不太会用,没关系,我们结合发表的文章,让它们一较高下。
1. Peak在TSS上下游的分布图
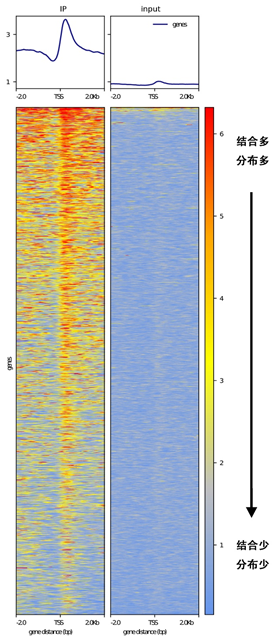
这张图实际包括两部分,上部整体展示了基因转录起始位点(TSS)及上下游的input和IP的reads的覆盖深度分布,可以看出结合峰主要位于TSS附近,下部就详细展示了每一个基因TSS上下游的reads覆盖深度,红色表示覆盖深度高、目的蛋白结合多。我们在不少文献中都可以看到这张图,既可以完整展示,也可以部分展示。
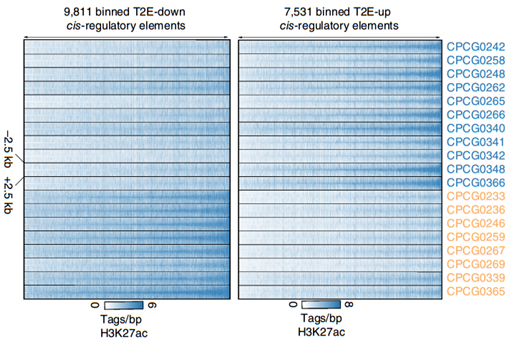
两组样本(蓝色和橙色)组蛋白修饰H3K27ac分布,同一基因(位点)在一组中修饰程度较高,而在另一组中修饰程度较低。(图片来源:TMPRSS2–ERG
fusion co-opts master transcription factors and activates NOTCH
signaling in primary prostate cancer)
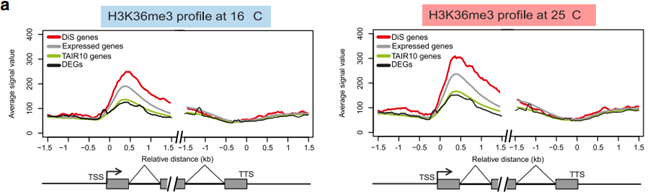
不同温度条件对组蛋白H3K36me3修饰的整体丰度产生影响。(Histone H3 lysine 36 methylation
affects temperature-induced alternative splicing and flowering in
plants)
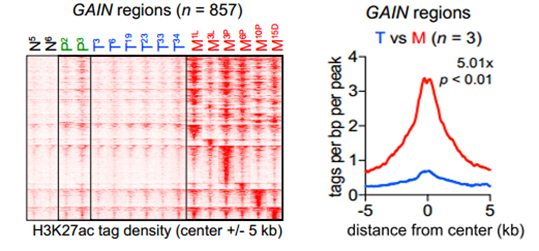
不同类型(T、M、N、P)样本的H3K27ac修饰分布差异,M组在整体及多个基因的H3K27ac修饰均高于T组。(图片来源:Enhancer Reprogramming Promotes Pancreatic Cancer Metastasis)
Peak在TSS上下游的分布图侧重于整体展示目的蛋白的结合、分布情况,较多用于组蛋白修饰在全基因组范围的分布,因为组蛋白修饰在全基因组范围分布较广。
2.reads在Peak关联基因上的分布图
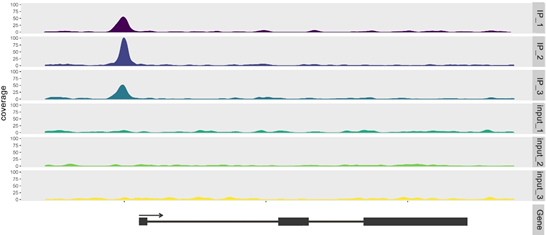
选取筛选到Peak的某些关键基因作图,展示该基因上目的蛋白的结合分布,横坐标显示基因位置,纵坐标表示reads覆盖深度,比较IP和input的reads覆盖深度可知目的蛋白结合位置。相比Peak在TSS上下游的分布图,它更有针对性,也更加直观。
它侧重展示目的蛋白在某个关键基因的特殊位点的结合、分布情况,在阐述转录因子的结合位点及调控机制时,能起到较好的作用。
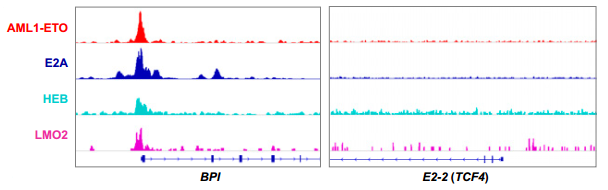
转录因子复合体AETFC中的AML1-ETO、E2A、HEB、LMO2均结合基因BPI而非E2-2(TCF4)。(图片来源:Different
roles of E proteins in t(8;21) leukemia: E2-2 compromises the function
of AETFC and negatively regulates leukemogenesis)

基因unc-104、linc-73附近的组蛋白修饰分布(图片来源:Systematic evaluation of C. elegans lincRNAs with CRISPR knockout mutants)
而且它还可用于不同组样本(不同处理条件)的差异结合峰的展示:
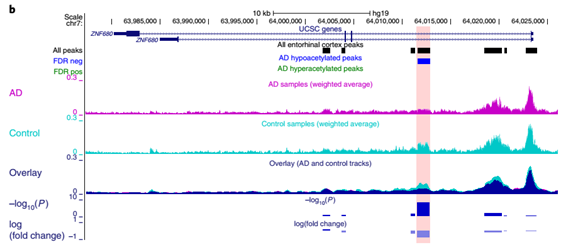
比较AD组和Control组,寻找差异结合峰,overlay的标红区域为差异结合位置。(图片来源:A histone
acetylome-wide association study of Alzheimer’s disease identifies
disease-associated H3K27ac differences in the entorhinal cortex)
两种展示方式各有侧重,根据自己的需要灵活运用吧。