今天和大家分享的是2020年3月发表在Frontiers in Genetics(IF:3.517)上的一篇文章,“Modulator-Dependent
RBPs Changes Alternative Splicing Outcomes in Kidney
Cancer”,文章中作者使用TCGA数据库中的肾透明细胞癌数据集,以MISO软件计算可变剪接事件的包含率,进一步通过构建调节剂-RBP-AS三元组,分析调节剂对RBPs的AS调控功能的影响。
Modulator-Dependent RBPs Changes Alternative Splicing Outcomes in Kidney Cancer
肾癌中调节剂依赖的RBPs改变可变剪接的结果
一、研究背景
肾细胞癌(RCC)是一种是一种常见的恶性肿瘤,占所有新发癌症病例的4.2%,具有放疗和化疗耐药性,目前手术仍是一线治疗方案。并且经过早期手术治疗仍有30%的患者最终发生转移,而转移性的肾透明细胞癌(KIRC)的患者2年生存率不到20%。RNA可变剪接(AS)失调的产物被认为是潜在的生物标志物,在疾病中具有关键作用。AS事件很大程度上受到RNA结合蛋白(RBPs)的调控,而RBP的调控作用受到特定细胞刺激下调节剂(如信号蛋白,miRNA,lncRNA等分子)的影响。作者希望揭示调节剂、RBPs与AS之间的关系。
二、分析流程
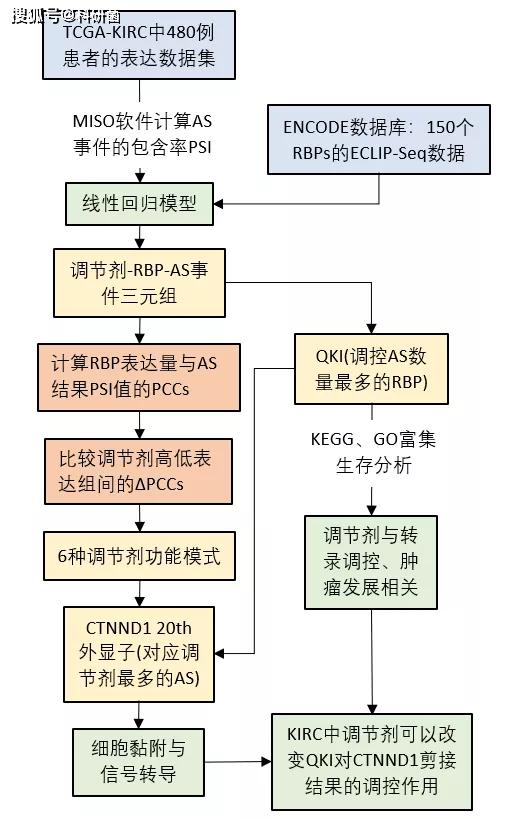
三、结果解读
1.调节剂-RBP-AS三元组的构建方法与调节剂作用的分类方法
数据来源:
RBPs与调节剂表达数据:TCGA-KIRC数据库中下载的480名KIRC患者的RNA-seq数据。
AS的量化数据:使用MISO软件计算样本中AS事件的包含率(PSI,percent spliced
isoform),保留在全部480个样本中至少100个样本PSI值大于0.1的事件,对原始AS事件进行处理以生成高置信度事件(总样本的约21%)。本研究仅关注外显子跳跃skipped
exon(SE)事件。
RBPs的结合信息:从ENCODE数据库下载的150个RBPs的ECLIP-Seq数据作为结合信息。
从Ensemble下载hg19参考基因组。
数据过滤:仅保留 log2 (CPM) >= 1的表达数据以及在全部480个样本中至少100个样本PSI值大于0.1,且PSI的变异系数(CV)大于0.1的事件。
使用线性回归模型预测可能的triplets(三元组)
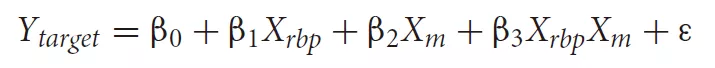
Ytarget为目标基因的AS结果,Xrbp和Xm分别为RBP和调节剂对AS结果的单独作用,XrbpXm为二者的共同作用,如果RBP与调节剂的互作可以影响AS结果,那么模型中β3不为0。作者筛选出其中β3不为0的三元组进行下一步分析。
接着作者在每一个三元组中根据调节剂的表达水平将样本分为高和低(前33%和后33%)两组,分别计算RBP表达量与AS结果的PSI值的皮尔逊相关系数PCCs。
最后比较两组间的ΔPCCs来确定调节剂的功能模式:“attenuates splicing” ,“enhances
splicing”,“inverts
splicing”(减弱,增强,反转)。如表一所示,作者根据|PCClow|、|PCChigh|以及p值将调节功能定义为减弱/增强外显子排除/包含,外显子包含/排除反转为排除/包含这6种模式。
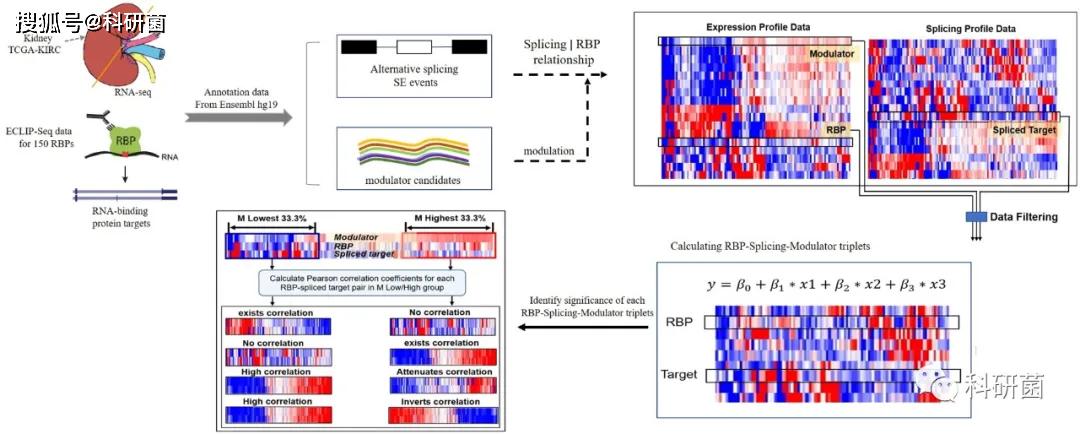
图1:工作流程图,调节剂-RBP-AS triplets的构建与调节剂作用分类
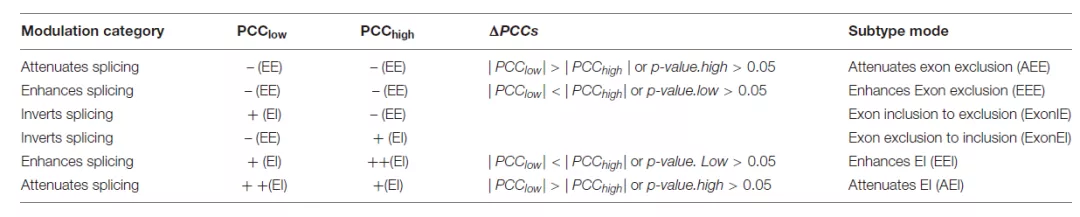
表1:调节剂介导的RBP剪接调控类别
2.确定肾癌中QKI的调节剂
作者使用上述方法在TCGA-KIRC数据中鉴定了1,040,254个FDR<=0.01的潜在三元组,经过数据过滤后最终确定了调控AS结果数量最多的RNA结合蛋白Quaking
(QKI),对应2,014个Modulator-QKI-Splicing三元组(包含1,101个调节剂,187个剪接事件和130个相应基因)。作者对这些调节剂的功能进行了分析。
图A展示了2,014个三元组的调节作用在6个模式中的分布。作者发现作用于多个剪接事件的调节剂大多数具有多种作用模式,并且同一调节剂作用于QKI的不同AS事件,结果可能是相反的。
比如在ARMH4-QKI-CLTC中,ARMH4低表达时QKI的表达量增加与CLTC外显子的包含有关,ARMH4高表达时QKI的表达量增加与CLTC外显子的排除有关。而在ARMH4-QKI-STIM1中,ARMH4从低表达到高表达时,QKI表达量与STIM1外显子包含率的相关性是增加的。
作者认为这一发现表明不同的调节模式依赖于调节剂-RBP-AS三元组,而不是特定的RBP或目标基因,即调节剂的表达水平可以影响RBPs对AS结果的调控功能。
作者以调节剂的作用模式对三元组进行聚类,同一组中的调节剂倾向于以同种模式对QKI的剪接作用进行调节(图B),表明这些调节剂可以作为QKI的拮抗剂或激活剂来影响AS结果。
作者进一步发现许多调节剂作用于相同的QKI剪接目标,提示这些调节剂可能在相关途径中共表达或者具有相似的功能。
图C展示了QKI调节剂的相关性热图,可以看到结果聚为明显的四类。
KEGG通路富集结果显示这些调节剂在细胞因子-细胞因子受体相互作用,Th1和Th2细胞分化等通路中显著富集(图D)。
图E展示了根据基因的生物类型和特征对QKI调节剂的分类结果(包括免疫相关基因,转录因子和lncRNA)。
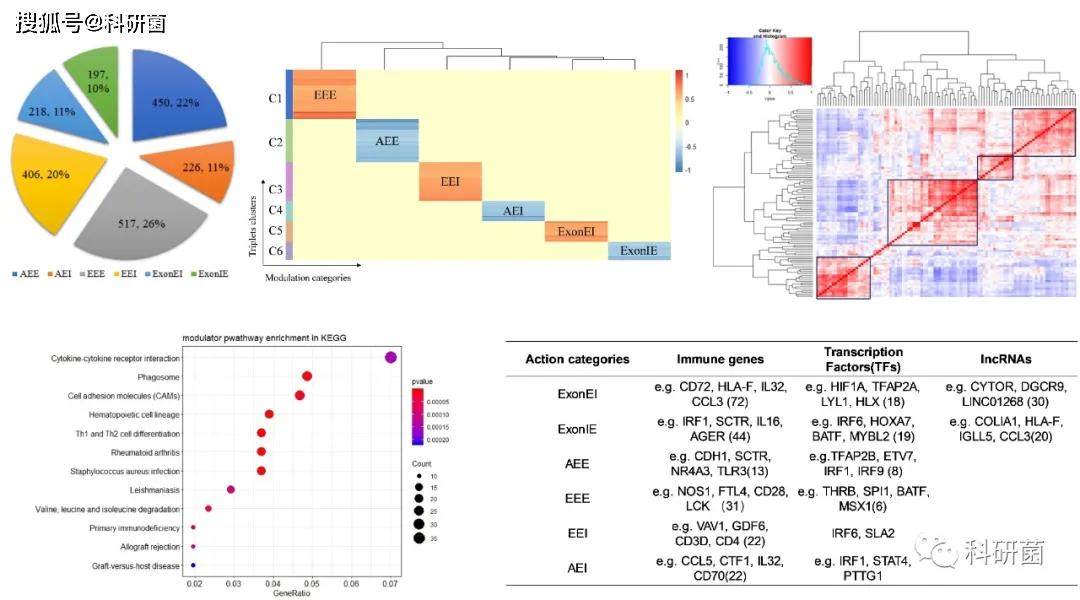
图2:RNA结合蛋白QKI相关调节剂的鉴定
3.QKI调节剂的功能分析
作者从三类调节剂中挑选了六个,包括免疫基因(CCL3,HLA-F,AGER),转录因子(ARMH4,STAT4)和lncRNA(LINC01268),展示QKI-splicing-调节剂之间的调节关系。三维散点图中蓝色和红色分别表示调节剂表达低和高两组的样品,并拟合了QKI-splicing-调节剂的回归平面。(图3)
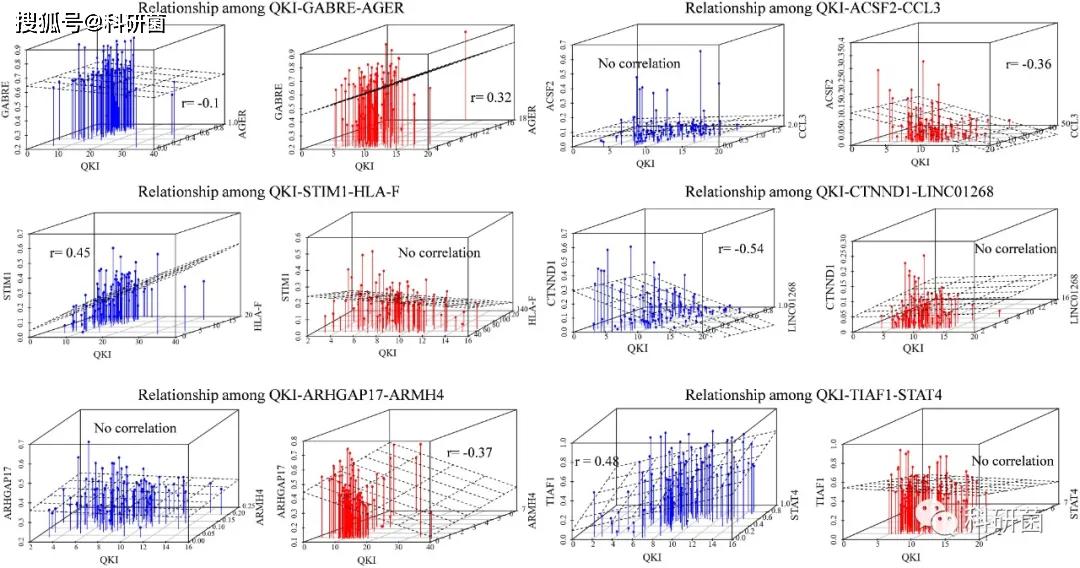
图3:QKI-splicing-调节剂的相关性
作者进一步研究AS失调与KIRC发展之间的关系,在TCGA-KIRC数据集中分析了肾癌样本与正常样本间的调节剂表达差异以及生存差异,结果显示大部分调节剂具有表达差异并与生存相关。
图A,B展示其中对目标基因AS影响最为显著的10个调节剂。可以看到箱线图中肿瘤组织(红色框)与正常组织(灰色框)调节剂表达水平具有显著差异。生存分析中以调节剂表达水平中值将样本分为高表达组和低表达组,可以看到组间具有显著生存差异。
KEGG通路富集显示,QKI调节剂在肿瘤进展相关通路中存在富集(图C)。GO富集分析显示调节剂在转录调控相关的过程中富集,包括RNA剪接、细胞生长和蛋白结合等(图D-F)。作者认为富集结果表明QKI主要在剪接水平对基因进行调控,当调节剂对QKI表达水平进行干扰时,QKI的RNA结合、剪接和调控等功能失调。
图G中作者使用Network of Cancer Genes (NCG) 和 Tumor suppressor gene
database (TSGene)
数据库鉴定调节剂中的肿瘤相关基因。结果显示149(13%)个调节剂基因为NCG中的肿瘤驱动基因,77(7%)个为TSGene中的蛋白质编码抑癌基因。
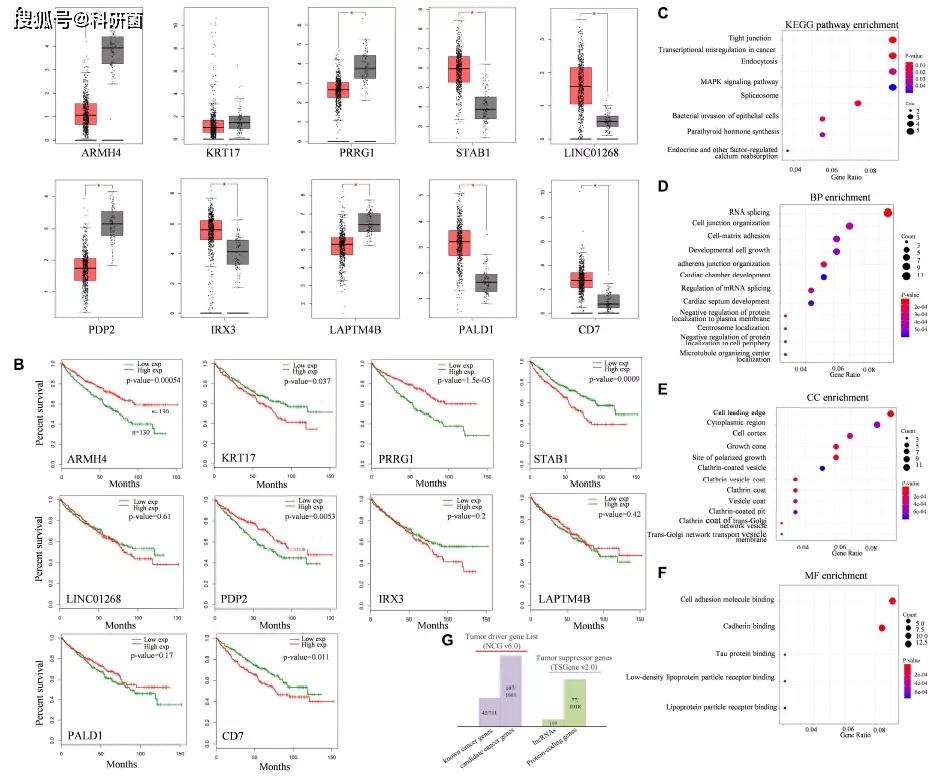
图4:QKI调节剂的功能分析
4.分析肾癌中调节剂影响的CTNND1剪接结果
CTNND1的第20号外显子剪接是相关调节剂最多的AS事件:“chr11:57582866: 57582972: + @ chr11:
57583387:57583473: + @ chr11:57583769:
57586652:+”,已有研究显示CTNND1编码Armadillo蛋白家族中的一个成员,该成员在细胞黏附与信号转导中发挥作用。多种CTNND1剪接亚型在细胞中表达,仅表达full-length
CTNND1的细胞具有侵袭性。
在CTNND1的相关调节剂中鉴定出了107个减弱外显子排除和7个增强外显子排除的调节剂。图A为剪接调控示意图。
图B中作者展示了其中四种调节剂的作用,可以看到调节剂高低表达组中QKI的剪接调控作用明显不同。TNFRSF14低表达组中QKI表达量与CTNND1
PSI之间的相关系数为-0.38 (p = 1.7e-05),而在TNFRSF14高表达组中两者不具有相关性(correlation =
0.001, p = 0.98),表明TNFRSF14的高表达可能抑制QKI的剪接活性。
以上结果表明差异表达的调节剂可以改变QKI在调控CTNND1剪接结果中的作用,作者认为这种调节模式为研究疾病相关的AS失调提供了新的思路。
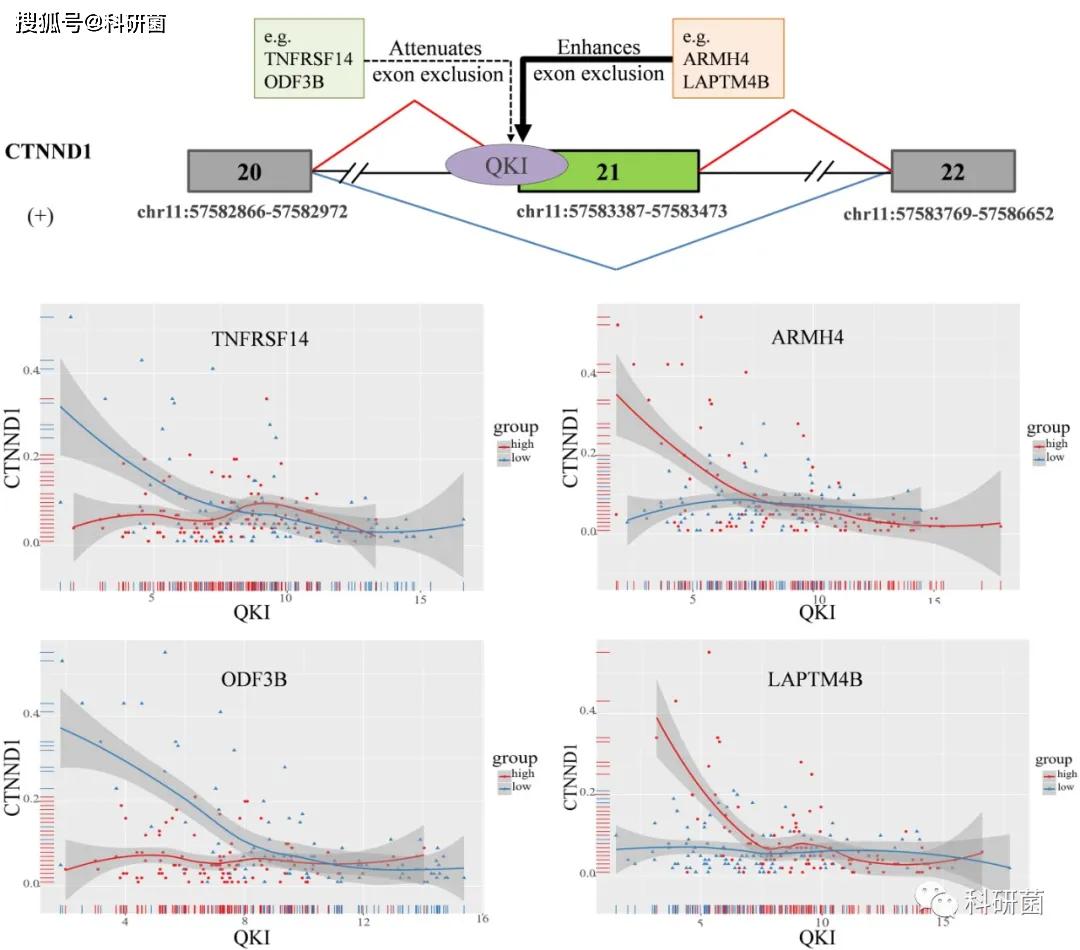
图5:KIRC中CTNND1的剪接结果受相关调节剂表达水平的影响
小结
这篇文章中作者主要是使用一种新颖的工作框架研究调节剂-RBPs-AS结果之间的调节作用,在可变剪接的调控中提供了一条新的研究思路。首先作者使用MISO软件计算TCGA-KIRC中480例样本的AS事件的PSI,接着计算RBPs表达水平与PSI的相关性系数PCC,并以线性回归模型建立调节剂-RBPs-AS结果三元组,然后以调节剂表达水平将三元组中的样本分为高低两组,计算组间的PCC差值,从而确定调节剂的调控模式。作者进一步选出其中调控AS最多的RNA结合蛋白QKI和对应最多调节剂的AS事件,对三元组的调控关系进行了展示,并对调节剂基因进行了KEGG和GO富集,分析其在KIRC中的可能作用,最后得出KIRC中调节剂影响RBPs的AS调控作用,且AS的失调与肿瘤进展相关