前语
ATAC-seq/ChIP-Seq中重复样本的处理
ATAC-Seq要求有必要有2次或更多次生物学重复(非常珍贵或者稀有样本除外,但有必要做至少2次技能重复)。理论上重复样本的peaks应该有高度的一致性,实际情况并不完全与预期一致。怎么点评重复样本的重复性的好坏?怎么得到一致性的peaks?
这一节将介绍两种办法:
1. 用Bedtools进行简略的overlap合并重复样本
2. 用IDR(Irreproducibility Discovery Rate)的办法获得高重复性的peaks
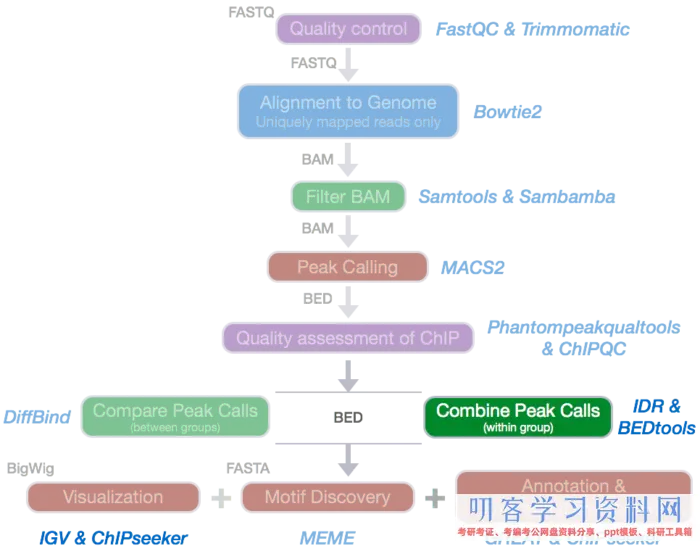
1. Overlapping peaks using bedtools
怎么得到两个重复样本间一致性的peaks? 一种简略粗暴的办法就是用bedtools核算peaks的overlaps。
用法:bedtools intersect [OPTIONS] -a <bed/gff/vcf/bam> -b <bed/gff/vcf/bam>
-a: 参数后加重复样本1(A)
-b:参数后加重复样本2(B),也能够加多个样本
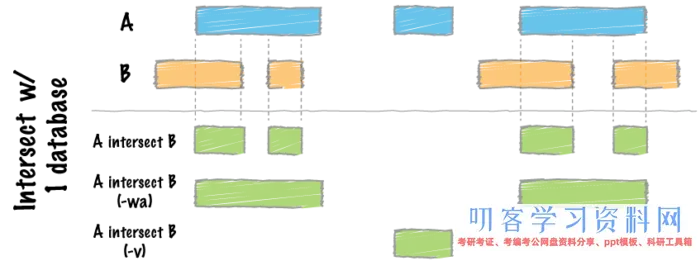
其他常用参数解释和图解如下:
-wo:Write the original A and B entries plus the number of base pairs of overlap between the two features.
-wa:Write the original entry in A for each overlap.
-v:Only report those entries in A that have no overlaps with B
如果只要-a和-b参数,回来的是相对于A的overlaps。加上参数-wo回来A和B原始的记载加上overlap的记载。参数-wa回来每个overlap中A的原始记载。
运用代码示例:
bedtools intersect -a macs2/Nanog-rep1_peaks.narrowPeak -b macs2/Nanog-rep2_peaks.narrowPeak -wo > bedtools/Nanog-overlaps.bed
2. Irreproducibility Discovery Rate (IDR)
评价重复样本间peaks一致性的另一种办法是IDR。IDR是经过比较一对经过排序的regions/peaks 的列表,然后核算反映其重复性的值。
IDR在ENCODE和modENCODE项目中被广泛运用,也是ChIP-seq指南和规范中的一部分。
IDR的 长处:
避免了初始阈值的选择,处理了不同callers的不行比较性
IDR不依靠于阈值的选择,一切regions/peaks都被考虑在内。
它是依靠regions/peaks的排序,不要求对输入信号进行校准或规范化
IDR的具体阐明参阅:
https://github.com/nboley/idr
https://github.com/hbctraining/In-depth-NGS-Data-Analysis-Course/blob/master/sessionV/lessons/06_handling-replicates.md#irreproducibility-discovery-rate-idr
运用IDR的注意事项:
主张运用IDR时,MACS2 call peaks的步骤参数设置不要过于严格,以便鉴定出更多的peaks。
运用IDR需要先对MACS2的成果文件narrowPeak依据-log10(p-value)进行排序。
# Call peaks
macs2 callpeak -t sample.final.bam -n sample --shift -100 --extsize 200 --nomodel -B --SPMR -g hs --outdir Macs2_out 2> sample.macs2.log
#Sort peak by -log10(p-value)
sort -k8,8nr NAME_OF_INPUT_peaks.narrowPeak > macs/NAME_FOR_OUPUT_peaks.narrowPeak
运用IDR示例
idr --samples sample_Rep1_sorted_peaks.narrowPeak sample_Rep2_sorted_peaks.narrowPeak --input-file-type narrowPeak --rank p.value --output-file sample-idr --plot --log-output-file sample.idr.log
--samples:narrowPeak的输入文件(重复样本)
--input-file-type:输入文件格局包括narrowPeak,broadPeak,bed
--rank p.value:以p-value排序
--output-file: 输出文件路径
--plot:输出IDR度量值的成果
输出文件解读:
具体内容可参阅:https://github.com/nboley/idr#output-file-format
输出文件包括:
sample-idr
sample-idr.log
sample-idr.png
(1)sample-idr
sample-idr是common peaks的成果输出文件,格局与输入文件格局类似,仅仅多了几列信息。前10列是规范的narrowPeak格局文件,包括重复样本整合后的peaks信息。
第5列:包括缩放的 IDR 值
score int
如min(int(log2(-125IDR), 1000),那么IDR=0,缩放的IDR就是1000;IDR=0.05, int(-125log2(0.05)) = 540;IDR=1.0, int(-125log2(1.0) = 0。
第11列和第12列:分别是local和global IDR值
col11: localIDR float -log10(Local IDR value)
col12: globalIDR float -log10(Global IDR value)
global IDR值是第5列中用于核算缩放的IDR值,类似于对p值进行多个假定校对以核算FDR;local
IDR类似于属于不行重复噪声部分的峰值的后验概率。具体内容可阅览Measuring reproducibility of
high-throughput experiments。
第13、14、15、16列:是重复样本1相关的信息。
col13: rep1_chromStart int
col14: rep1_chromEnd int
col15: rep1_signalValue float
col16: rep1_summit int
第17-20列:是重复样本2相关的信息,和13-16四列信息类似。
其他列信息如下:
col1: chrom string
col2: chromStart int
col3: chromEnd int
col4: name string
col6: strand [ -.] Use ‘.’ if no strand is assigned.
col7: signalValue float
col8: p-value float
col9: q-value float
col10: summit int
wc -l *-idr 核算下common peaks的个数,接着可再核算下与总peaks的比率。
如果想看IDR<0.05的,能够经过第5列信息过滤:
awk \'{if($5 >= 540) print $0}\' sample-idr | wc -l
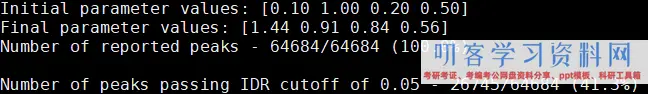
(2)sample-idr.log
log文件会给出peaks经过IDR < 0.05的比率,如下图所示
(3)sample-idr.png
png文件包括4个图
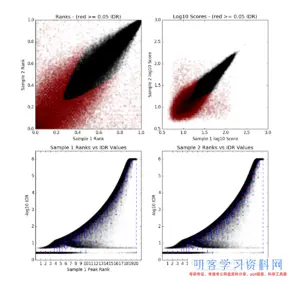
左上: Rep1 peak ranks vs Rep2 peak ranks, 没有经过特定IDR阈值的peaks显现为赤色。
右上:Rep1 log10 peak scores vs Rep2 log10 peak scores,没有经过特定IDR阈值的peaks显现为赤色。
下面两个图: Peak rank vs IDR scores,箱线图展现了IDR值的分布,默许情况下,IDR值的阈值为-1E-6。