热图是科研论文中一种常见的可视化手段,因其丰富的色彩变化和饱满的信息涵盖量,往往是一篇文章中最引人注目的所在之一。上至
CNS 顶刊,下至1到 2分小文,可以说热图无处不在,堪称文章C位。如何做出一张完美的热图,是居家旅行(科研写作),拜访亲朋好友(征服
editor和reviewer 的心)必备技能。本次教程,我们将为大家详细讲述如何使用R语言绘制高大上的热图。
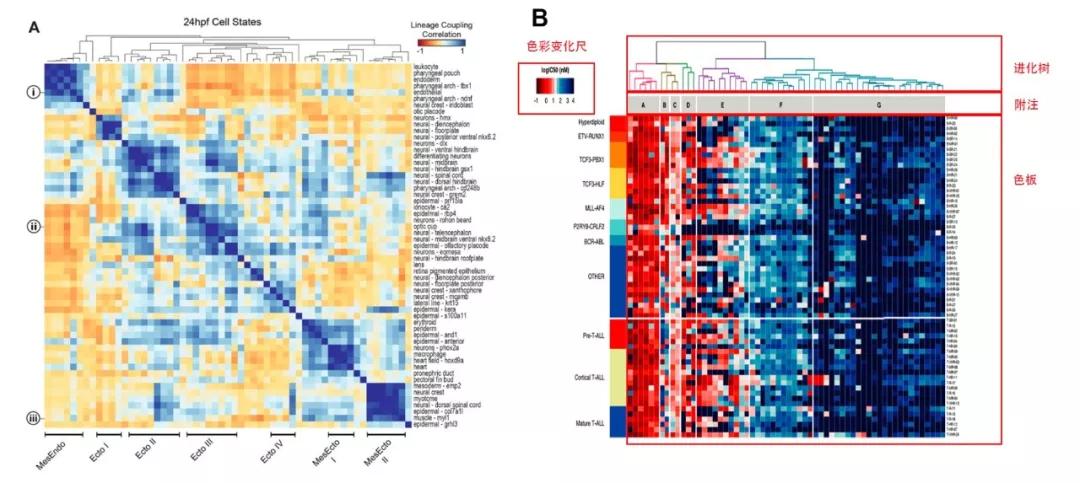
什么是热图,比如上图是来源于两篇 CNS 级别文章中截取下来的主图,一眼看去,一张合格的热图主要由四大部分组成,一个是像浴室瓷砖一样的小色块铺成的色板,也可以称作热图本身,是热图必不可少的部分,一个是色板上面的聚类树,一个是色板和聚类树中间附注,用来标注样本的信息,最后一个便是标注色板的颜色变化尺。
热图的本质是表现数值矩阵,色板中的每个方格都是一个数值,按照色彩变化尺的要求,根据数值大小显示出不同颜色。在生物医学领域的文章中,热图通常用来以样本为列,基因等实验得到的表型数值为行,用来展示不同组别/样本之间的差异。
好了,理论知识就介绍到这里,接下来就直接上干货啦~
本次教程介绍 pheatmap 这个 R 包,此包功能强大,制作热图方便给力。
1. pheatmap包安装及加载
我们先在R上安装pheatmap这个包,首先打开Rstudio。

2. 实战演练
pheatmap包的数据输入是一个矩阵(matrix),我们先读入基因表达谱数据 expression.txt。

使用 head函数查看文件,expression.txt 文件中一共包含10个样本,50个基因,其中每一列为一个样本,每一行为一个基因
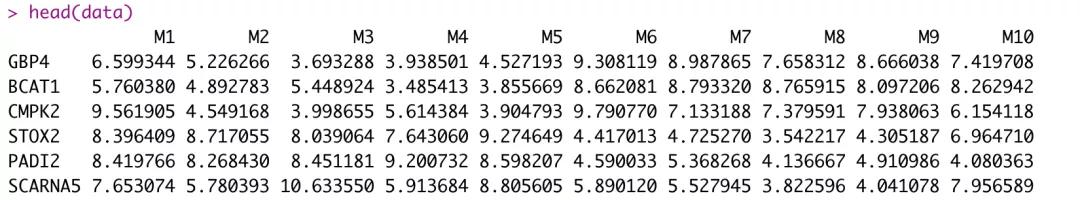
比如第一行第一列数据,代表M1样本中GBP4的表达量,直接使用pheatmap(data),便可以得到一张热图。


这样,一张热图就出来啦~

如果我们要修改热图的颜色,只需要使用color这个参数就可以啦。这里给大家推荐一个网址,https://www.bootcss.com/p/websafecolors/,里面有非常多的颜色可供选择。使用color颜色更改颜色变化尺之后的热图会更好看啦。我们可以参考文章中的绘图颜色,这样会让我们的热图更富有视觉效果。

color.key是我们构建的颜色变化尺,以后只需要修改color.key的内容,就可以轻松搞定各种色图颜色了。
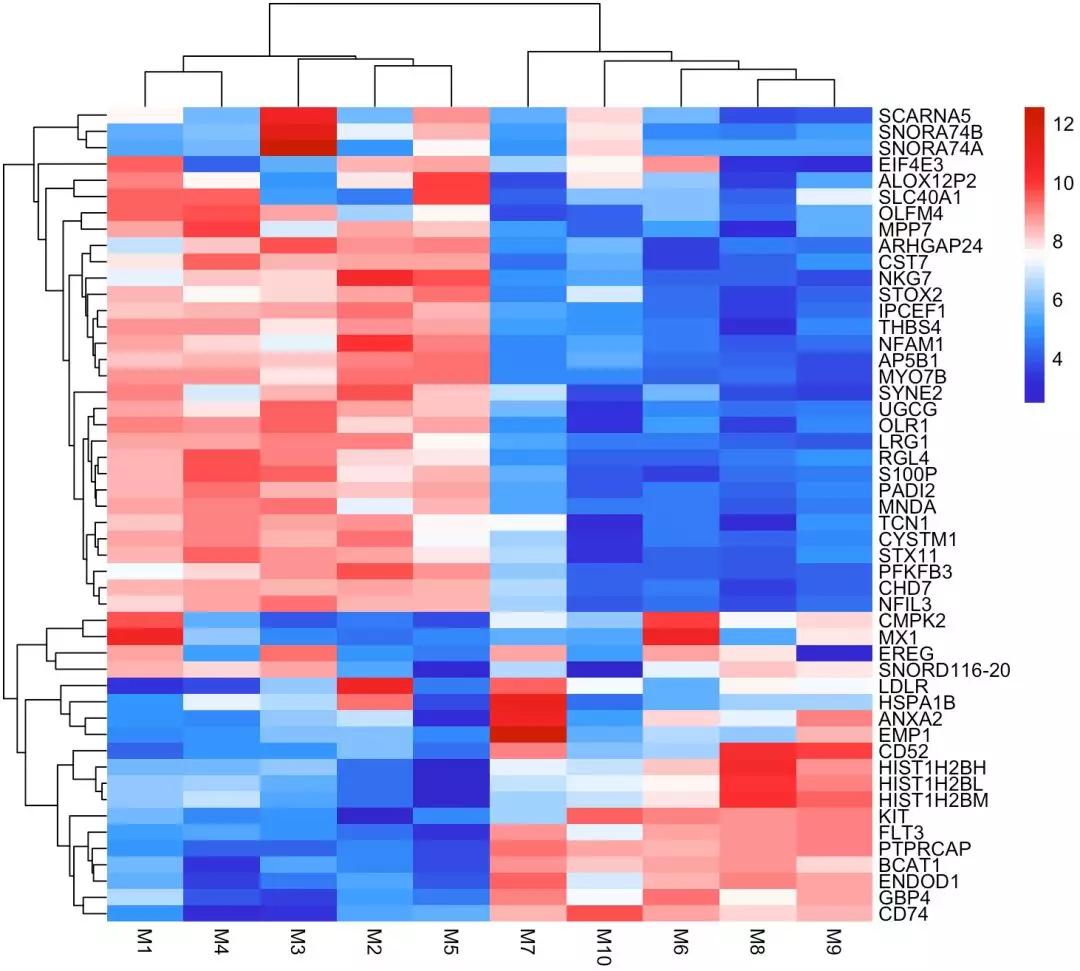
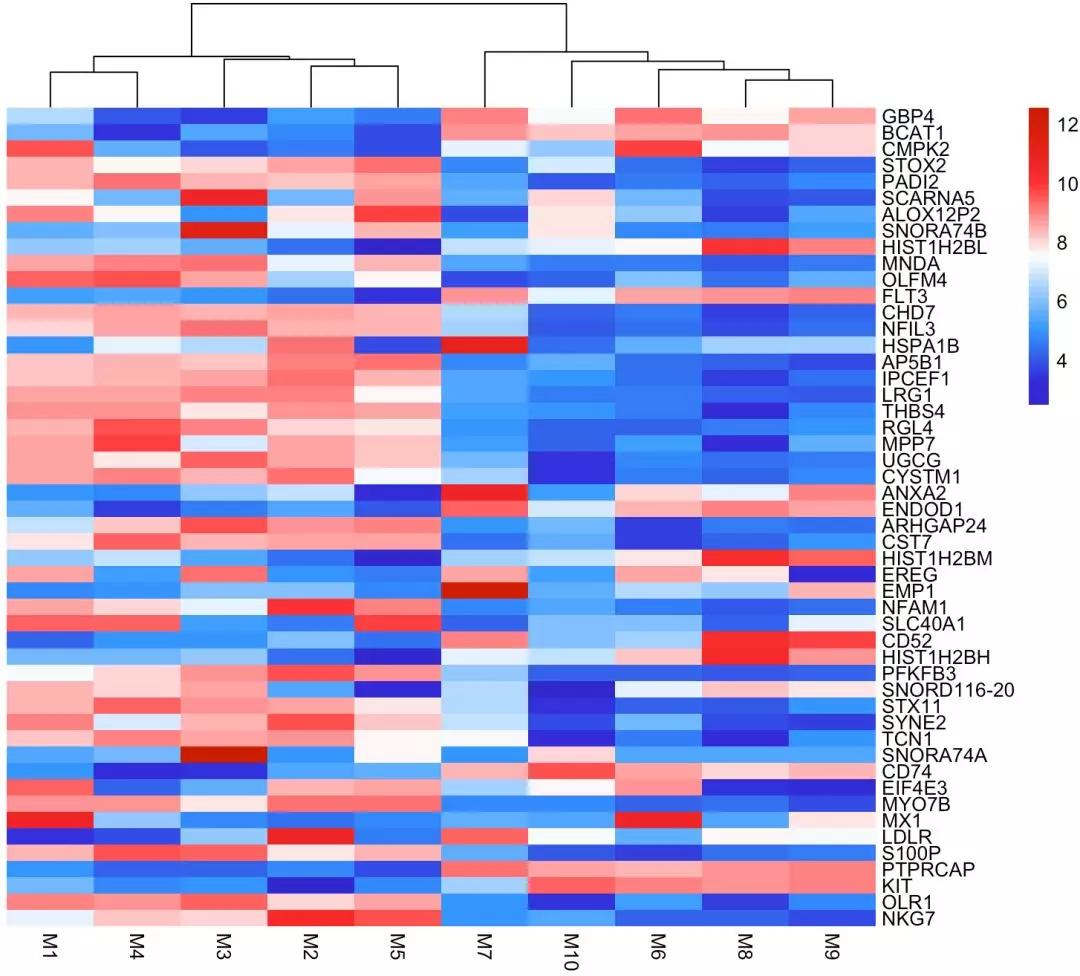
在科研做图过程中,有时候我们并不需要对基因或者样本进行聚类,那么如何调整热图中的聚类树呢,在pheatmap函数中,cluster_row参数可以控制基因的聚类,cluster_col可以控制样本的聚类。比如将cluster_row设置为FALSE,那么基因就不会进行聚类运算了。

参考科研期刊中的文章热图,还有一个重要的部分,就是样本的附注,用来标注哪些样本是实验组,哪些样本是对照组。那么如何实现在热图中添加标注呢?代码如下:

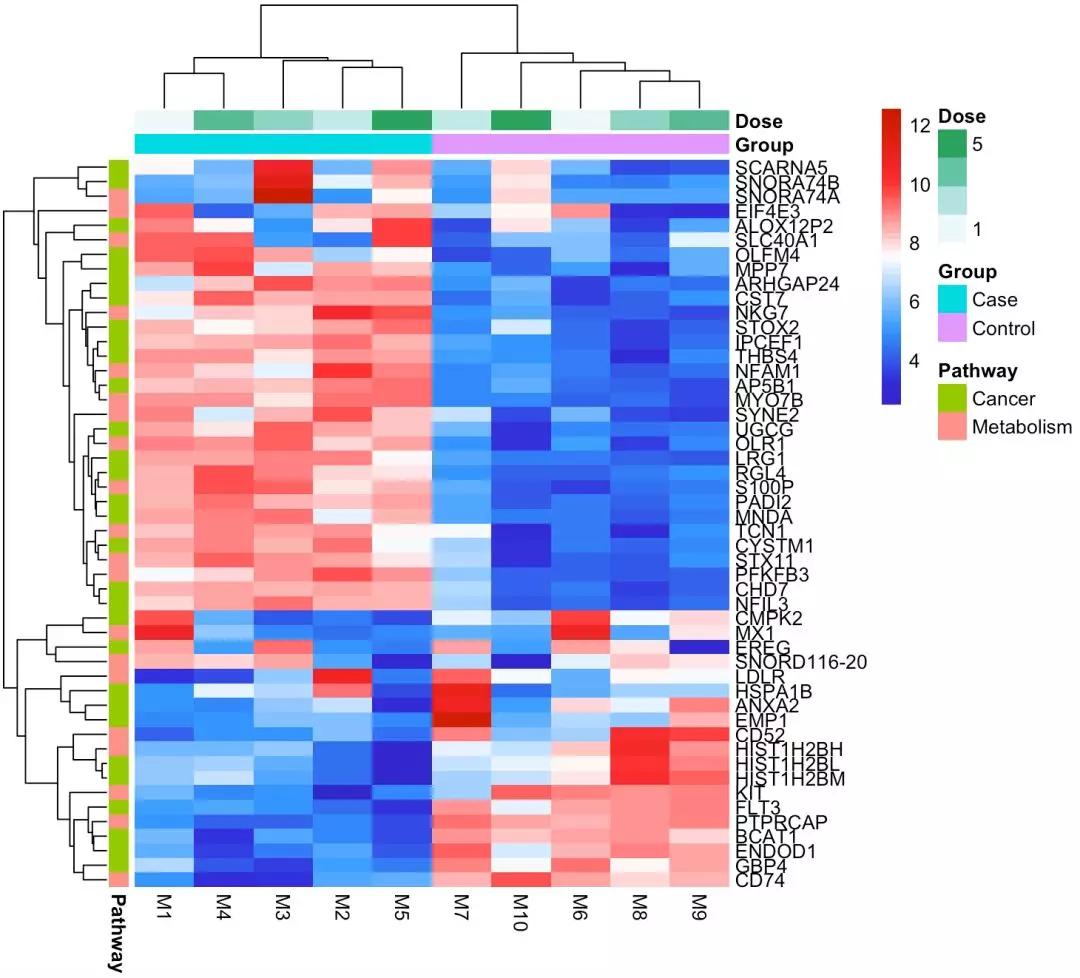
如此,一张热腾腾的热图就这么出炉啦~
转自:科研猫